15.8 Un esempio concreto
Applichiamo ora il risultato precedente ad un caso concreto. Consideriamo i dati utilizzati nella validazione italiana del Cognitive Style Questionnaire - Short Form (CSQ-SF, Meins et al. 2012). Il CSQ-SF viene utilizzato per misurare la vulnerabilità all’ansia e alla depressione. È costituito da cinque sottoscale: Internality, Globality, Stability, Negative consequences e Self-worth.
Leggiamo i dati in \(\textsf{R}\):
Il numero di partecipanti è
Le statistiche descrittive si ottengono con la seguente istruzione:
psych::describe(csq, type = 2)
#> vars n mean sd median trimmed mad min max range skew kurtosis se
#> I 1 540 47.76 5.78 48 47.87 4.45 21 64 43 -0.31 1.07 0.25
#> G 2 540 45.00 11.94 42 44.55 11.86 16 78 62 0.34 -0.70 0.51
#> S 3 540 44.60 12.18 42 44.24 13.34 16 77 61 0.27 -0.77 0.52
#> N 4 540 22.01 6.92 21 21.86 7.41 8 39 31 0.21 -0.74 0.30
#> W 5 540 44.05 13.10 43 43.66 13.34 16 79 63 0.31 -0.53 0.56Esaminiamo la matrice di correlazione:
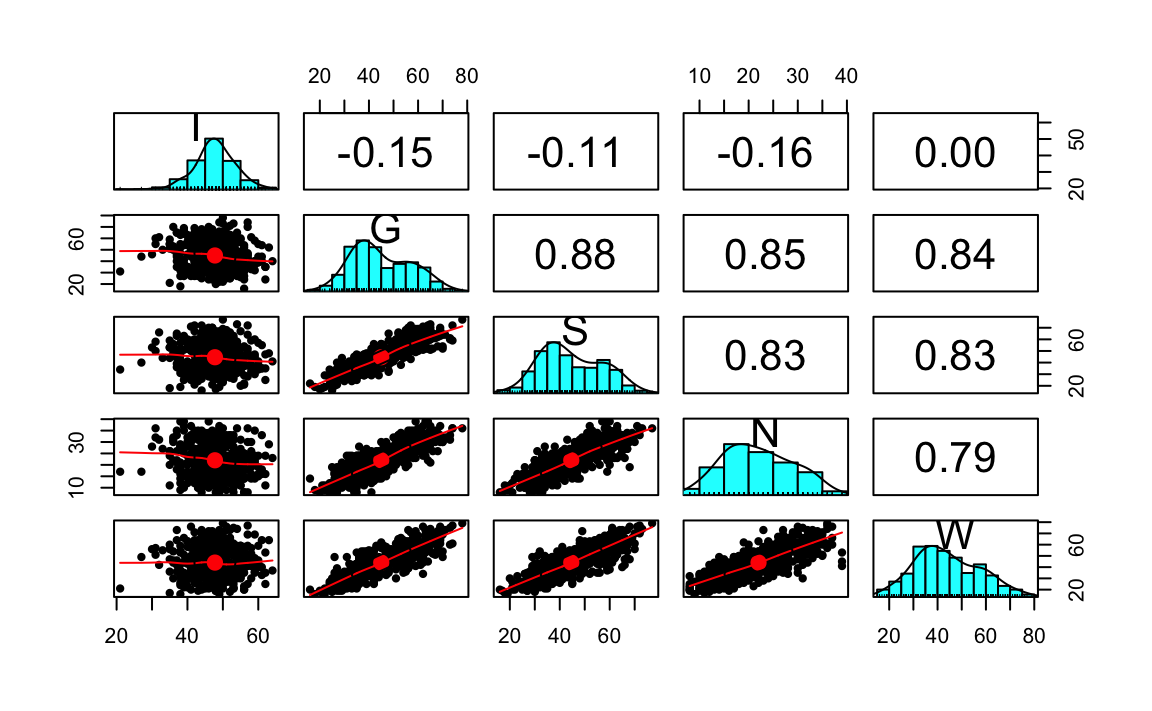
La sottoscala di Internality è problematica, come messo anche in evidenza dall’autore del test. La consideriamo comunque in questa analisi statistica.
Specifichiamo il modello unifattoriale nella sintassi di lavaan:
Adattiamo il modello ai dati:
Esaminiamo i risultati:
summary(
fit,
standardized = TRUE,
fit.measures = TRUE
)
#> lavaan 0.6.15 ended normally after 26 iterations
#>
#> Estimator ML
#> Optimization method NLMINB
#> Number of model parameters 10
#>
#> Number of observations 540
#>
#> Model Test User Model:
#>
#> Test statistic 46.716
#> Degrees of freedom 5
#> P-value (Chi-square) 0.000
#>
#> Model Test Baseline Model:
#>
#> Test statistic 2361.816
#> Degrees of freedom 10
#> P-value 0.000
#>
#> User Model versus Baseline Model:
#>
#> Comparative Fit Index (CFI) 0.982
#> Tucker-Lewis Index (TLI) 0.965
#>
#> Loglikelihood and Information Criteria:
#>
#> Loglikelihood user model (H0) -8741.781
#> Loglikelihood unrestricted model (H1) -8718.423
#>
#> Akaike (AIC) 17503.562
#> Bayesian (BIC) 17546.478
#> Sample-size adjusted Bayesian (SABIC) 17514.734
#>
#> Root Mean Square Error of Approximation:
#>
#> RMSEA 0.124
#> 90 Percent confidence interval - lower 0.093
#> 90 Percent confidence interval - upper 0.158
#> P-value H_0: RMSEA <= 0.050 0.000
#> P-value H_0: RMSEA >= 0.080 0.989
#>
#> Standardized Root Mean Square Residual:
#>
#> SRMR 0.033
#>
#> Parameter Estimates:
#>
#> Standard errors Standard
#> Information Expected
#> Information saturated (h1) model Structured
#>
#> Latent Variables:
#> Estimate Std.Err z-value P(>|z|) Std.lv Std.all
#> F =~
#> I 0.725 0.253 2.867 0.004 0.725 0.126
#> G -11.322 0.384 -29.481 0.000 -11.322 -0.949
#> S -11.342 0.398 -28.513 0.000 -11.342 -0.932
#> N -6.163 0.233 -26.398 0.000 -6.163 -0.891
#> W -11.598 0.444 -26.137 0.000 -11.598 -0.886
#>
#> Variances:
#> Estimate Std.Err z-value P(>|z|) Std.lv Std.all
#> F 1.000 1.000 1.000
#> .I 32.840 2.000 16.420 0.000 32.840 0.984
#> .G 14.038 1.473 9.532 0.000 14.038 0.099
#> .S 19.508 1.718 11.353 0.000 19.508 0.132
#> .N 9.847 0.725 13.573 0.000 9.847 0.206
#> .W 36.892 2.685 13.737 0.000 36.892 0.215Esaminiamo solo le stime dei parametri del modello:
parameterEstimates(fit)
#> lhs op rhs est se z pvalue ci.lower ci.upper
#> 1 F =~ I 0.725 0.253 2.867 0.004 0.229 1.220
#> 2 F =~ G -11.322 0.384 -29.481 0.000 -12.075 -10.569
#> 3 F =~ S -11.342 0.398 -28.513 0.000 -12.122 -10.563
#> 4 F =~ N -6.163 0.233 -26.398 0.000 -6.621 -5.705
#> 5 F =~ W -11.598 0.444 -26.137 0.000 -12.467 -10.728
#> 6 F ~~ F 1.000 0.000 NA NA 1.000 1.000
#> 7 I ~~ I 32.840 2.000 16.420 0.000 28.920 36.759
#> 8 G ~~ G 14.038 1.473 9.532 0.000 11.151 16.924
#> 9 S ~~ S 19.508 1.718 11.353 0.000 16.140 22.876
#> 10 N ~~ N 9.847 0.725 13.573 0.000 8.425 11.269
#> 11 W ~~ W 36.892 2.685 13.737 0.000 31.628 42.155Recuperiamo le specificità:
psi <- parameterEstimates(fit)$est[7:11]
psi
#> [1] 32.839665 14.037578 19.508119 9.846927 36.891617Stimiamo l’errore standard della misurazione con la (15.1):
Applichiamo ora la formula della TCT:
\[ \sigma_E = \sigma_X \sqrt{1 -\rho_{XX^\prime}}. \]
Per trovare \(\sigma\) calcoliamo prima il punteggio totale:
La deviazione standard di tot_score ci fornisce una stima di \(\sigma_X\):
Per applicare la formula della TCT abbiamo bisogno dell’attendibilità. La stimiamo usando la funzione reliability del pacchetto semTools dall’oggetto creato da lavaan:::cfa():
rel <- semTools::reliability(fit)
rel
#> F
#> alpha 0.8506572
#> omega 0.9330313
#> omega2 0.9330313
#> omega3 0.9273385
#> avevar 0.7916575Utilizzando \(\Omega\) otteniamo:
Si noti come il risultato sia molto simile a quello trovato con la formula della TCT.
15.8.1 Correlazioni osservate e riprodotte
Le correlazioni riprodotte dal modello si ottengono nel modo seguente dall’oggetto fit.
cor_mat <- lavInspect(fit, "cor.ov")
cor_mat
#> I G S N W
#> I 1.000
#> G -0.119 1.000
#> S -0.117 0.885 1.000
#> N -0.112 0.846 0.830 1.000
#> W -0.111 0.841 0.825 0.789 1.000Abbiamo visto come il modello unifattoriale predice che la correlazione tra due variabili manifeste sia il prodotto delle rispettive correlazioni fattoriali. Estraiamo le saturazioni fattoriali.
l <- inspect(fit, what = "std")$lambda
l
#> F
#> I 0.126
#> G -0.949
#> S -0.932
#> N -0.891
#> W -0.886Per esempio, se consideriamo I e G, la correlazione predetta dal modello fattoriale tra queste due sottoscale è data dal prodotto delle rispettive saturazioni fattoriali.
La matrice di correlazioni riprodotte riportata sopra mostra il risultato di questo prodotto per ciascuna coppia di variabili manifeste.
15.8.2 Scomposizione della varianza
Consideriamo la variabile manifesta W. Calcoliamo la varianza.
La varianza riprodotta di questa variabile, secondo il modello fattoriale, dovrebbe esere uguale alla somma di due componenti: la varianza predetta dall’effetto causale del fattore latente e la varianza residua. La varianza predetta dall’effetto causale del fattore latente è uguale alla saturazione elevata al quadrato:
Calcolo ora la proporzione di varianza residua normalizzando rispetto alla varianza osservata (non a quella riprodotta dal modello):
Il valore così ottenuto è molto simile al valore della varianza residua di W.
Ripeto i calcoli per la variabile G
e per la variabile I
In tutti i casi, i valori ottenuti sono molto simili alle varianze residue ipotizzate dal modello unifattoriale.
15.8.3 Correlazione tra variabili manifeste e fattore comune
Un modo per verificare il fatto che, nel modello unifattoriale, la saturazione fattoriale della \(i\)-esima variabile manifesta è uguale alla correlazione tra i punteggi osservati sulla i$-esima variabile manifesta e il fattore latente è quella di calcolare le correlazioni tra le variabili manifeste e i punteggi fattoriali. I punteggi fattoriali rappresentano una stima del punteggio “vero”, ovvero del punteggio che ciascun rispondente otterrebbe in assenza di errori di misurazione. Vedremo in seguito come si possono stimare i punteggi fattoriali. Per ora ci limitiamo a calcolarli usando lavaan.
head(lavPredict(fit))
#> F
#> [1,] 0.2693790
#> [2,] -0.9110820
#> [3,] 0.1871406
#> [4,] -0.3315541
#> [5,] 0.8306627
#> [6,] 1.1534515Abbiamo un punteggio diverso per ciascuno dei 540 individui che appartengono al campione di dati esaminato.
Calcoliamo ora le correlazioni tra i valori osservati su ciascuna delle cinque scale del CSQ e le stime dei punteggi veri.
c(
cor(csq$I, lavPredict(fit)),
cor(csq$G, lavPredict(fit)),
cor(csq$S, lavPredict(fit)),
cor(csq$N, lavPredict(fit)),
cor(csq$W, lavPredict(fit))
) |>
round(3)
#> [1] 0.128 -0.970 -0.952 -0.910 -0.905Si noti che i valori ottenui sono molto simili ai valori delle saturazioni fattoriali. La piccola differenza tra le correlazioni ottenute e i valori delle saturazioni fattoriali dipende dal fatto che abbiamo stimato i punteggi fattoriali.