5.5 Diagramma a dispersione
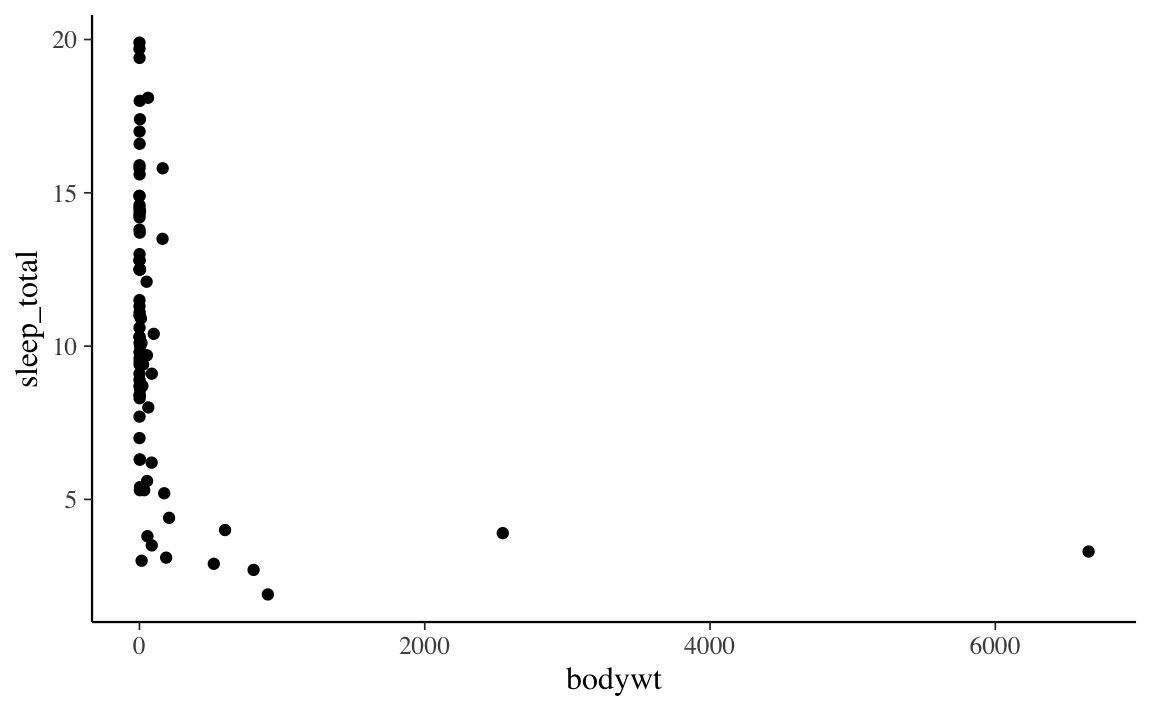
Consideriamo nuovamenti i dati contenuti nel tibble msleep e poniamoci il problema di rappresentare graficamente la relazione tra il numero medio di ore di sonno giornaliero (sleep_total) e il peso dell’animale (bodywt). Usando le impostazioni di default di ggplot2, con le istruzioni seguenti, otteniamo il grafico fornito dalla figura seguente.
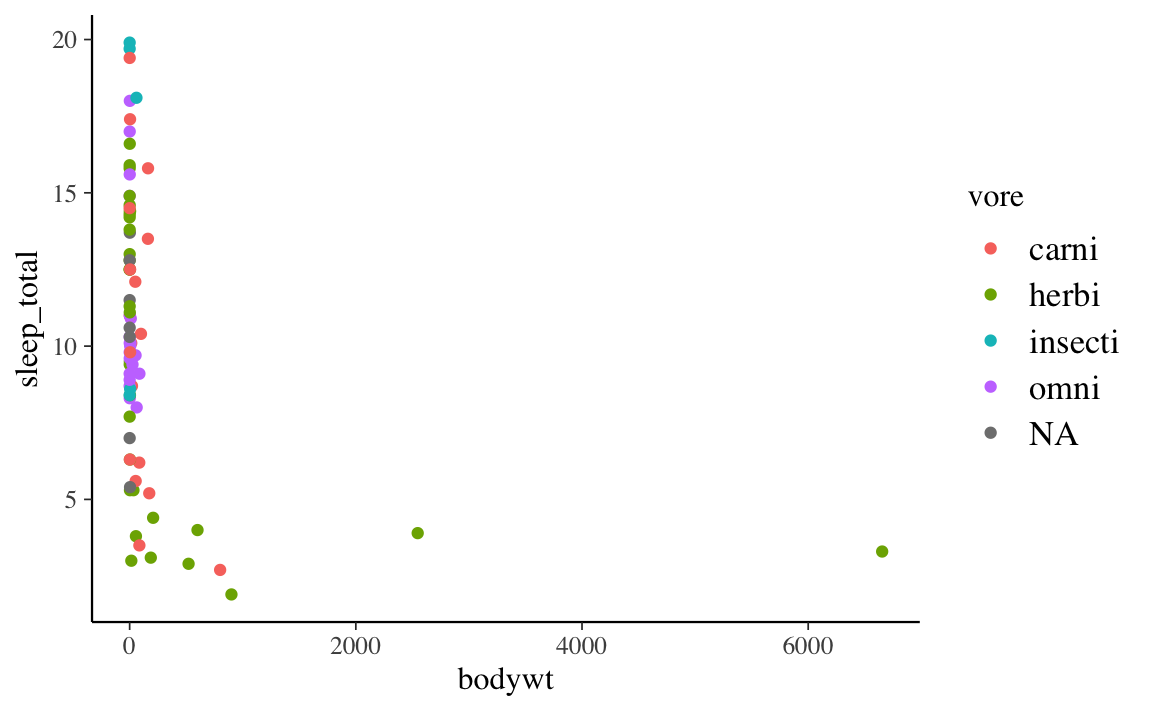
Coloriamo ora in maniera diversa i punti che rappresentano animali carnivori, erbivori, ecc.
È chiaro, senza fare alcuna analisi statistica, che la relazione tra le due variabili non è lineare. Trasformando in maniera logaritmica i valori dell’asse \(x\) la relazione si linearizza.
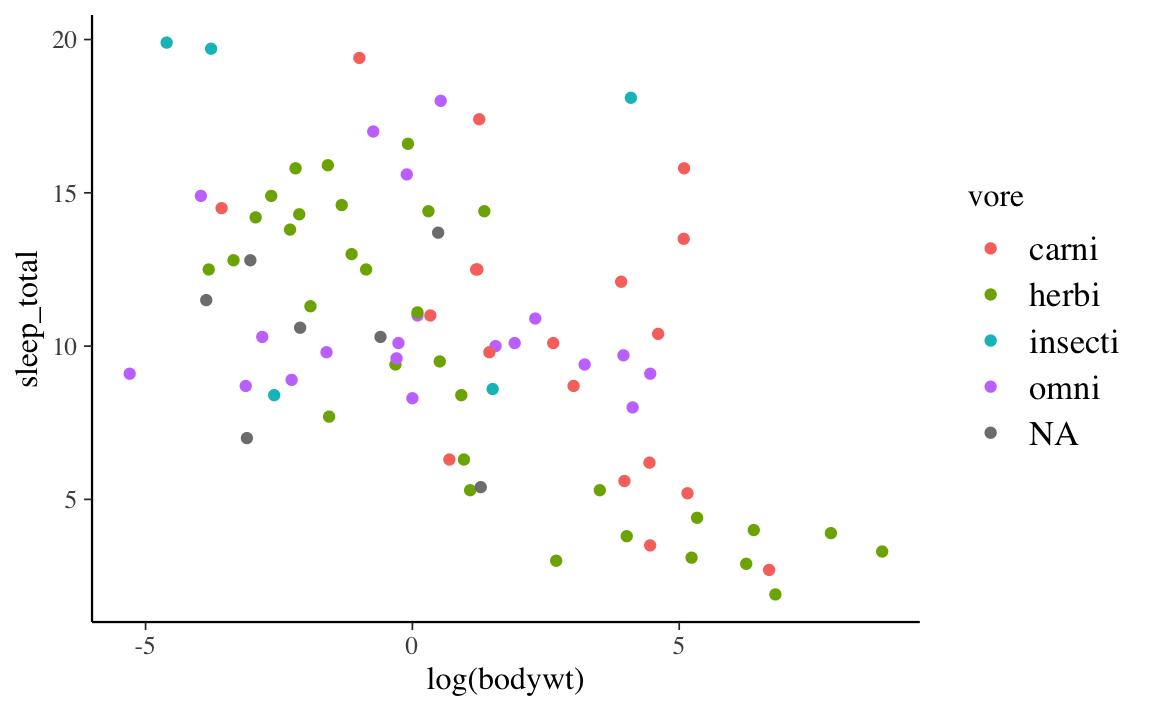
Infine, aggiustiamo il “tema” del grafico (si noti l’utilizzo di una tavolozza di colori adatta ai daltonici mediante il pacchetto viridis), aggiungiamo le etichette sugli assi e il titolo.
library("viridis")
msleep %>%
ggplot(
aes(x = log(bodywt), y = sleep_total, col = vore)
) +
geom_point(size = 2, alpha = .8) +
labs(
x = "Peso corporeo (log)",
y = "Ore di sonno",
title = "Il sonno in 83 specie di mammiferi",
subtitle = "Un esempio di visualizzazione con ggplot()",
caption = "Fonte: Savage e West (2007)"
) +
scale_fill_viridis(discrete = TRUE, option = "viridis") +
theme_minimal() +
theme(legend.title = element_blank())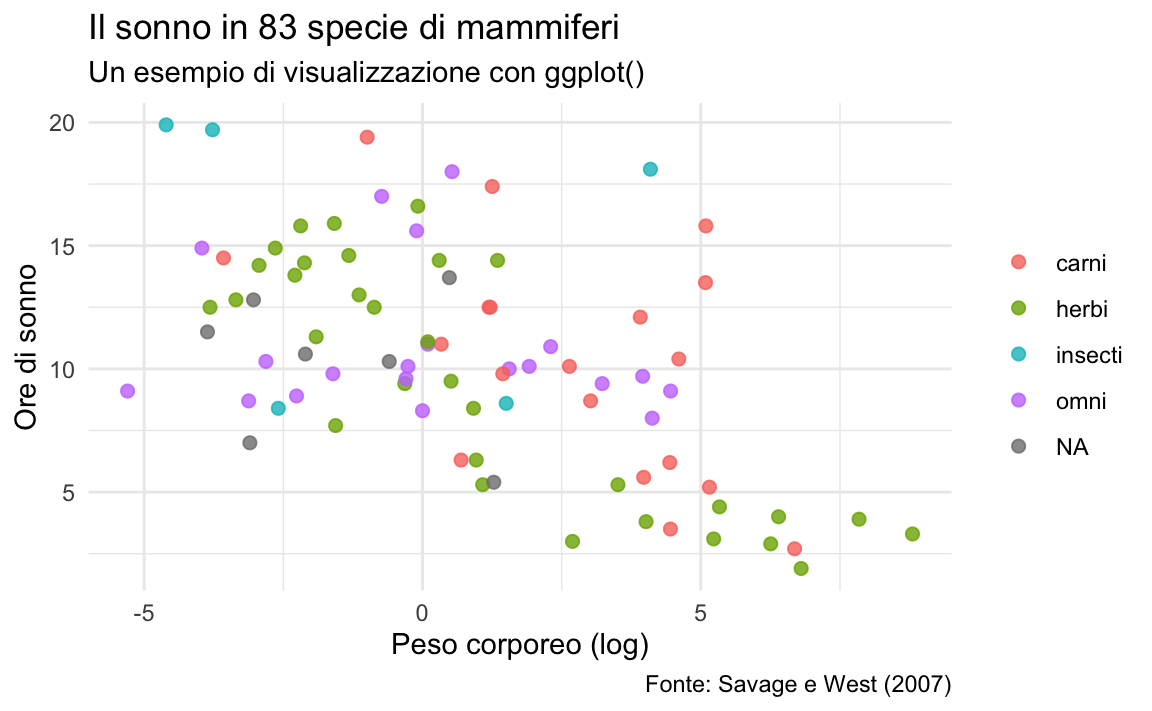
La visualizzazione può essere migliorata cambiando le etichette della legenda del grafico. Per fare questo è necessario intervenire sui dati prima di usare ggplot() – per esempio, come abbiamo fatto in precedenza con la funzione forcats::fct_recode().
5.5.1 Istogramma
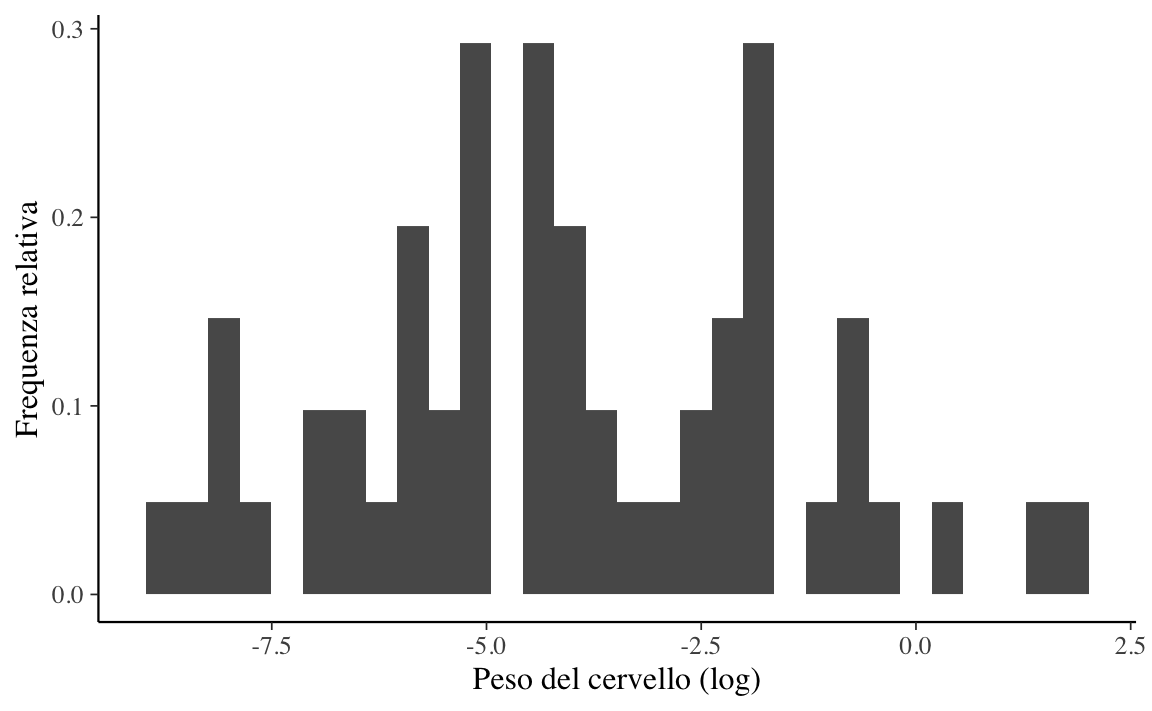
Creiamo ora un istogramma che rappresenta la distribuzione del (logaritmo del) peso medio del cervello delle 83 specie di mammiferi considerate da Savage and West (2007). L’argomento aes(y = ..density..) in geom_histogram() produce le frequenze relative. L’opzione di default (senza questo argomento) porta ggplot() a rappresentare le frequenze assolute.
msleep %>%
ggplot(
aes(log(brainwt))
) +
geom_histogram(aes(y = ..density..)) +
labs(
x = "Peso del cervello (log)",
y = "Frequenza relativa"
) +
theme(legend.title = element_blank())