here::here("code", "_common.R") |>
source()
if (!requireNamespace("pacman")) install.packages("pacman")
pacman::p_load(cmdstanr, posterior, bayesplot, ggplot2, dplyr, tibble, forcats)
conflicts_prefer(posterior::ess_bulk)
conflicts_prefer(posterior::ess_tail)10 Confrontare due medie
Introduzione
Nel manuale didattico il confronto tra due medie viene introdotto nel quadro della regressione lineare con un predittore dicotomico (0 = controllo, 1 = intervento): lì si mostra che la differenza tra le medie di due gruppi coincide con il coefficiente associato al predittore e che l’incertezza su tale differenza si ottiene dalla varianza stimata del coefficiente.
In questo capitolo riprendiamo lo stesso problema ma lo affrontiamo in chiave generativa-bayesiana con Stan, così da (i) rendere esplicite le assunzioni stocastiche sul processo che genera i dati, (ii) specificare prior debolmente informative coerenti con una variabile standardizzata, (iii) stimare congiuntamente la differenza tra le medie e — quando serve — la differenza tra le varianze, e (iv) scegliere tra modelli in base alla capacità predittiva (ELPD con PSIS-LOO).
Immaginiamo uno studio in cui confrontiamo il punteggio medio di affetto negativo settimanale tra un gruppo che segue un breve training di mindfulness e un gruppo di controllo. La domanda sostantiva non è “c’è qualche differenza?” ma “di quanto differiscono i gruppi e quanto siamo incerti su questa differenza?”. È questa la prospettiva che adottiamo qui: invece di ridurre il problema a un verdetto sì/no, stimiamo l’intera distribuzione a posteriori della differenza tra le medie e traduciamo tale distribuzione in affermazioni pratiche rilevanti per la psicologia.
Un aspetto spesso trascurato è la variabilità: i due gruppi possono avere dispersioni diverse. Se, per esempio, il training riduce l’affetto negativo medio ma produce risposte più eterogenee, allora la differenza tra deviazioni standard è informativa quanto la differenza tra medie. L’impostazione bayesiana consente di stimare entrambe le quantità e la loro incertezza in modo naturale.
Per stabilizzare le stime e semplificare le scelte di prior, standardizziamo l’esito sull’intero campione (media 0, deviazione standard 1). Lavoreremo con due modelli didatticamente complementari: uno a varianza comune e uno a varianze distinte. In entrambi useremo prior Normali centrate in 0 per le medie e prior Esponenziali per le deviazioni standard, coerenti con la scala standardizzata.
La domanda metodologica non è quale ipotesi sia “vera”, ma quale modello predice meglio dati simili ai nostri con la massima parsimonia. Useremo quindi l’ELPD stimata con PSIS-LOO per confrontare i due modelli. Il cuore dell’analisi rimane comunque la distribuzione a posteriori della differenza tra le medie: su di essa riportiamo intervalli di credibilità e probabilità a posteriori per scenari di interesse (ad es., la probabilità che la differenza sia inferiore a −0.2 SD, soglia ritenuta clinicamente rilevante). Inoltre, quando l’obiettivo applicativo è dimostrare l’irrilevanza pratica, useremo una ROPE attorno a zero per quantificare la plausibilità di effetti trascurabili.
Obiettivo dell’approfondimento in Stan. Rispetto alla trattazione tramite regressione lineare con predittore dicotomico, questo capitolo ha l’obiettivo di:
- mostrare l’equivalenza concettuale tra “differenza tra medie” e coefficiente del predittore (e come ciò emerga in un modello generativo);
- fornire un workflow completo in Stan (modello, prior, diagnostiche MCMC, PPC) per passare da stime puntuali a distribuzioni posteriori interpretabili;
- introdurre il confronto predittivo tra modelli (ELPD/PSIS-LOO) come criterio operativo di scelta tra varianza comune e varianze distinte;
- integrare la lettura sostantiva con ROPE ed eventuali estensioni robuste (es. likelihood \(t\)) quando la distribuzione empirica lo suggerisce.
In sintesi, useremo Stan per stimare due modelli semplici e confrontarli sul piano predittivo, mantenendo il focus sulle conclusioni sostantive: “di quanto” il training modifica l’affetto negativo medio, “quanto” è plausibile una differenza nulla o trascurabile, e “come” cambia l’eterogeneità delle risposte tra i partecipanti.
Panoramica del capitolo
- Comprendere il problema del confronto tra due medie in un’ottica bayesiana.
- Motivare la standardizzazione dei dati e la scelta di priors debolmente informative.
- Implementare in Stan un modello con varianza comune e uno con varianze distinte.
- Valutare la capacità predittiva dei modelli attraverso PSIS-LOO.
- Interpretare la distribuzione a posteriori della differenza tra le medie e le verifiche predittive.
10.1 Codice R
Simuliamo i dati di un piccolo studio psicologico: Gruppo A = controllo, Gruppo B = training mindfulness.
set.seed(123) # per rendere i risultati riproducibili
n_A <- 80 # numero partecipanti controllo
n_B <- 90 # numero partecipanti training
# Medie "vere" dei gruppi (in SD standardizzate)
true_mean_A <- 0.2 # controllo con affetto negativo leggermente più alto
true_mean_B <- -0.3 # training mindfulness riduce affetto negativo
# Deviazioni standard "vere"
true_sd_A <- 1.0
true_sd_B <- 1.0 # prova a cambiare in 1.4 per simulare varianze diverse
# Creiamo un data frame con le osservazioni
df <- tibble(
score = c(rnorm(n_A, true_mean_A, true_sd_A),
rnorm(n_B, true_mean_B, true_sd_B)),
group = factor(c(rep("Controllo", n_A), rep("Mindfulness", n_B)))
)
# Standardizziamo la variabile di esito
df <- df %>%
mutate(
score_std = (score - mean(score)) / sd(score),
g = as.integer(group) # Stan richiede indici numerici dei gruppi
)
# Prepariamo la lista di dati per Stan
stan_data <- list(
N = nrow(df), # numero totale osservazioni
J = 2L, # numero di gruppi
y = df$score_std, # variabile risposta standardizzata
g = df$g # indice di gruppo
)
glimpse(stan_data)
#> List of 4
#> $ N: int 170
#> $ J: int 2
#> $ y: num [1:170] -0.3016 0.0317 1.8369 0.3352 0.3945 ...
#> $ g: int [1:170] 1 1 1 1 1 1 1 1 1 1 ...In questo esempio, la variabile score rappresenta il livello di affetto negativo. Abbiamo simulato una piccola differenza: in media, il gruppo mindfulness ha punteggi più bassi (cioè minore affetto negativo) rispetto al gruppo controllo. Standardizzando la variabile, lavoriamo sempre in “unità di deviazione standard”, semplificando l’interpretazione delle priors e delle stime.
10.1.1 Differenza di medie e ROPE
Prima ancora di stimare i modelli in Stan, possiamo calcolare la differenza empirica tra le medie e confrontarla con una regione di irrilevanza pratica (ROPE). Per esempio, supponiamo che differenze inferiori a 0.1 SD non abbiano rilevanza psicologica. Questo ci permette di introdurre un confronto diretto con il test t: il t-test risponde solo alla domanda “la differenza è zero?”, mentre l’approccio bayesiano ci consente di valutare “la differenza è psicologicamente importante?”.
# Differenza empirica tra le medie
mean_diff <- mean(df$score_std[df$group == "Mindfulness"]) -
mean(df$score_std[df$group == "Controllo"])
mean_diff
#> [1] -0.535
# Definiamo una ROPE di ±0.1 SD
rope_lower <- -0.1
rope_upper <- 0.1
cat("Differenza osservata =", round(mean_diff, 3),
" | ROPE = [", rope_lower, ",", rope_upper, "]\n")
#> Differenza osservata = -0.535 | ROPE = [ -0.1 , 0.1 ]Naturalmente, la vera potenza del metodo bayesiano emergerà quando stimiamo la distribuzione a posteriori della differenza di medie. Allora potremo calcolare direttamente la probabilità a posteriori che la differenza ricada dentro o fuori dalla ROPE, ottenendo un’informazione più ricca e interpretabile rispetto al semplice p-value.
10.2 Modello a varianza comune
L’ipotesi è che i due gruppi condividano la stessa dispersione attorno alle loro medie. Questa è spesso una buona approssimazione iniziale, riduce il numero di parametri e permette stime più stabili con campioni piccoli o moderati. Il punto centrale resta la differenza tra le medie: se il training mindfulness riduce l’affetto negativo, la media del gruppo “Mindfulness” sarà più bassa e la differenza “Mindfulness − Controllo” sarà negativa in unità di deviazione standard.
# Salviamo il modello Stan con varianza comune
stan_equal_var <- "
data {
int<lower=1> N; // numero di osservazioni
int<lower=2> J; // numero di gruppi (qui: 2)
vector[N] y; // esito standardizzato
array[N] int<lower=1, upper=J> g; // indice di gruppo per ogni osservazione
}
parameters {
vector[J] mu; // medie dei due gruppi
real<lower=0> sigma; // deviazione standard comune
}
model {
// Priors debolmente informative, coerenti con y standardizzata
mu ~ normal(0, 1.5);
sigma ~ exponential(1);
// Likelihood: ogni y appartiene al suo gruppo g[i]
for (i in 1:N)
y[i] ~ normal(mu[g[i]], sigma);
}
generated quantities {
vector[N] log_lik; // log-verosimiglianze per PSIS-LOO
vector[N] y_rep; // repliche per posterior predictive checks
real diff_mu; // differenza tra le medie (Gruppo 2 - Gruppo 1)
// calcolo log_lik e repliche
for (i in 1:N) {
log_lik[i] = normal_lpdf(y[i] | mu[g[i]], sigma);
y_rep[i] = normal_rng(mu[g[i]], sigma);
}
// differenza di interesse sostantivo:
// attenzione: per coerenza didattica, assumiamo g=1 -> Controllo, g=2 -> Mindfulness
diff_mu = mu[2] - mu[1];
}
"mod_eq <- cmdstanr::cmdstan_model(write_stan_file(stan_equal_var))Eseguiamo il campionaento dalla distribuzione a posteriori:
fit_eq <- mod_eq$sample(
data = stan_data, # creato nella sezione precedente
seed = 2025,
chains = 4, parallel_chains = 4,
iter_warmup = 1000, iter_sampling = 1000,
refresh = 0
)Dopo l’esecuzione, verifichiamo a colpo d’occhio che i diagnostici principali siano buoni. Se R-hat è vicino a 1 e gli effective sample size sono adeguati, passiamo all’interpretazione. Usiamo direttamente le quantità generate nel blocco generated quantities, così lo studente vede continuità tra il modello e l’analisi a valle.
# Differenza tra medie: estraiamo il vettore 'diff_mu'
diff_draws <- fit_eq$draws(variables = "diff_mu", format = "draws_matrix")
# Riassunto compatto della posteriore: media, sd e intervallo di credibilità al 95%
q025 <- function(x) posterior::quantile2(x, probs = 0.025)
q975 <- function(x) posterior::quantile2(x, probs = 0.975)
posterior::summarize_draws(diff_draws, mean, sd, q025, q975)
#> # A tibble: 1 × 5
#> variable mean sd q2.5 q97.5
#> <chr> <dbl> <dbl> <dbl> <dbl>
#> 1 diff_mu -0.536 0.148 -0.817 -0.243L’oggetto diff_mu è la quantità sostantiva principale. Se nel nostro esempio è negativo, indica che il gruppo mindfulness ha, in media, affetto negativo più basso del controllo. Per rendere più esplicito il legame con l’interpretazione psicologica, calcoliamo la probabilità a posteriori che la differenza sia clinicamente rilevante secondo una ROPE scelta dal ricercatore. Per esemplificare, usiamo ±0.1 SD come regione di irrilevanza pratica; adattare questa soglia al contesto è parte del lavoro scientifico.
# Calcolo ROPE: probabilità a posteriori dentro/fuori la regione
rope_lower <- -0.1
rope_upper <- 0.1
diff_vec <- as.numeric(diff_draws[, "diff_mu"])
p_in_rope <- mean(diff_vec > rope_lower & diff_vec < rope_upper)
p_below <- mean(diff_vec < rope_lower)
p_above <- mean(diff_vec > rope_upper)
tibble(
`P(diff in ROPE)` = p_in_rope,
`P(diff < lower)` = p_below,
`P(diff > upper)` = p_above
)
#> # A tibble: 1 × 3
#> `P(diff in ROPE)` `P(diff < lower)` `P(diff > upper)`
#> <dbl> <dbl> <dbl>
#> 1 0.0025 0.998 0Queste probabilità mostrano qualcosa che il t-test non fornisce: non solo se la differenza è “diversa da zero”, ma se è trascurabile, peggiorativa o migliorativa oltre una soglia ritenuta rilevante. In un report didattico, affiancherei a queste quantità un confronto predittivo e una verifica grafica della coerenza del modello con i dati.
A questo punto abbiamo tutto ciò che serve per la lettura sostantiva: una distribuzione a posteriori della differenza tra le medie con intervallo di credibilità, una probabilità a posteriori di effetti rilevanti o trascurabili e un controllo predittivo che ci dice se il modello è in grado di generare dati verosimili. Nel passaggio successivo introdurremo il modello con varianze distinte e useremo PSIS-LOO per verificare se l’aumento di complessità offre un miglioramento predittivo stabile; se non lo offre, resteremo sul modello parsimonioso a varianza comune, senza perdere nulla sul piano interpretativo centrale.
10.3 Modello a varianze distinte
L’ipotesi qui è che i due gruppi possano avere eterogeneità diversa: ad esempio, il training di mindfulness potrebbe ridurre l’affetto negativo in media, ma con risposte più variabili tra i partecipanti rispetto al controllo. Stimiamo quindi \(\mu_1,\mu_2\) e \(\sigma_1,\sigma_2\). Come prima, lavoriamo su dati standardizzati: priors mu ~ Normal(0, 1.5) e sigma ~ Exponential(1).
# Modello Stan: varianze distinte
stan_unequal_var <- "
data {
int<lower=1> N; // numero di osservazioni
int<lower=2> J; // numero di gruppi (qui: 2)
vector[N] y; // esito standardizzato
array[N] int<lower=1, upper=J> g; // indice di gruppo
}
parameters {
vector[J] mu; // medie per gruppo
vector<lower=0>[J] sigma; // sd specifica di gruppo
}
model {
// Priors debolmente informative
mu ~ normal(0, 1.5);
sigma ~ exponential(1);
// Likelihood con sd per gruppo
for (i in 1:N)
y[i] ~ normal(mu[g[i]], sigma[g[i]]);
}
generated quantities {
vector[N] log_lik; // per PSIS-LOO
vector[N] y_rep; // posterior predictive checks
real diff_mu; // mu[2] - mu[1], Mindfulness - Controllo
real diff_sigma; // sigma[2] - sigma[1]
for (i in 1:N) {
log_lik[i] = normal_lpdf(y[i] | mu[g[i]], sigma[g[i]]);
y_rep[i] = normal_rng(mu[g[i]], sigma[g[i]]);
}
diff_mu = mu[2] - mu[1];
diff_sigma = sigma[2] - sigma[1];
}
"Compilazione:
mod_neq <- cmdstanr::cmdstan_model(write_stan_file(stan_unequal_var))Campionamento:
fit_neq <- mod_neq$sample(
data = stan_data,
seed = 2025,
chains = 4, parallel_chains = 4,
iter_warmup = 1000, iter_sampling = 1000,
refresh = 0
)
#> Running MCMC with 4 parallel chains...
#> Chain 1 finished in 0.1 seconds.
#> Chain 2 finished in 0.1 seconds.
#> Chain 3 finished in 0.1 seconds.
#> Chain 4 finished in 0.1 seconds.
#>
#> All 4 chains finished successfully.
#> Mean chain execution time: 0.1 seconds.
#> Total execution time: 0.3 seconds.10.3.1 Lettura sostantiva dei parametri
Come prima, l’oggetto chiave è diff_mu = mu[2] − mu[1] (Mindfulness − Controllo). Qui però abbiamo anche diff_sigma = sigma[2] − sigma[1], utile per capire se il training modifica l’eterogeneità delle risposte.
# Estraiamo le quantità principali
neq_draws_mu <- fit_neq$draws(variables = c("mu[1]", "mu[2]", "diff_mu"), format = "draws_matrix")
neq_draws_sigma <- fit_neq$draws(variables = c("sigma[1]", "sigma[2]", "diff_sigma"), format = "draws_matrix")
q025 <- function(x) posterior::quantile2(x, probs = 0.025)
q975 <- function(x) posterior::quantile2(x, probs = 0.975)
# Riassunti posteriori
posterior::summarize_draws(neq_draws_mu, mean, sd, q025, q975)
#> # A tibble: 3 × 5
#> variable mean sd q2.5 q97.5
#> <chr> <dbl> <dbl> <dbl> <dbl>
#> 1 mu[1] 0.282 0.102 0.0821 0.483
#> 2 mu[2] -0.254 0.108 -0.461 -0.0414
#> 3 diff_mu -0.536 0.148 -0.826 -0.249posterior::summarize_draws(neq_draws_sigma, mean, sd, q025, q975)
#> # A tibble: 3 × 5
#> variable mean sd q2.5 q97.5
#> <chr> <dbl> <dbl> <dbl> <dbl>
#> 1 sigma[1] 0.940 0.0762 0.804 1.10
#> 2 sigma[2] 1.00 0.0759 0.871 1.17
#> 3 diff_sigma 0.0629 0.107 -0.144 0.274Se diff_mu è negativo, il gruppo mindfulness ha in media affetto negativo più basso del controllo. Se diff_sigma è positivo, il gruppo mindfulness è più eterogeneo; se negativo, è più omogeneo. Questo secondo aspetto è spesso ignorato dal t-test, ma è scientificamente rilevante: un intervento può ridurre la media e cambiare la variabilità delle risposte.
Per continuità con la sezione precedente, calcoliamo anche la ROPE sulla differenza di medie. La ROPE serve a rispondere alla domanda: l’effetto è praticamente trascurabile?
# ROPE su diff_mu (stessa soglia di prima)
rope_lower <- -0.1
rope_upper <- 0.1
diff_mu_vec <- as.numeric(neq_draws_mu[, "diff_mu"])
p_in_rope_neq <- mean(diff_mu_vec > rope_lower & diff_mu_vec < rope_upper)
p_below_neq <- mean(diff_mu_vec < rope_lower)
p_above_neq <- mean(diff_mu_vec > rope_upper)
tibble(
Model = "Varianze distinte",
`P(diff in ROPE)` = p_in_rope_neq,
`P(diff < lower)` = p_below_neq,
`P(diff > upper)` = p_above_neq
)
#> # A tibble: 1 × 4
#> Model `P(diff in ROPE)` `P(diff < lower)` `P(diff > upper)`
#> <chr> <dbl> <dbl> <dbl>
#> 1 Varianze distinte 0.0005 1.000 010.3.2 Confronto predittivo con PSIS-LOO
Ora confrontiamo modello a varianza comune (fit_eq) e modello a varianze distinte (fit_neq) usando ELPD (più alto è meglio). Guardiamo anche i diagnostici Pareto-k: idealmente < 0.7. Se il vantaggio del modello più complesso è piccolo e incerto, restiamo sul modello parsimonioso; se è chiaro e stabile, accettiamo la maggiore complessità.
# LOO per entrambi i modelli
loo_eq <- fit_eq$loo()
loo_neq <- fit_neq$loo()
print(loo_eq)
#>
#> Computed from 4000 by 170 log-likelihood matrix.
#>
#> Estimate SE
#> elpd_loo -237.5 9.8
#> p_loo 3.1 0.5
#> looic 474.9 19.5
#> ------
#> MCSE of elpd_loo is 0.0.
#> MCSE and ESS estimates assume MCMC draws (r_eff in [0.8, 1.1]).
#>
#> All Pareto k estimates are good (k < 0.7).
#> See help('pareto-k-diagnostic') for details.print(loo_neq)
#>
#> Computed from 4000 by 170 log-likelihood matrix.
#>
#> Estimate SE
#> elpd_loo -238.3 9.8
#> p_loo 4.1 0.8
#> looic 476.6 19.6
#> ------
#> MCSE of elpd_loo is 0.0.
#> MCSE and ESS estimates assume MCMC draws (r_eff in [1.0, 1.3]).
#>
#> All Pareto k estimates are good (k < 0.7).
#> See help('pareto-k-diagnostic') for details.# Confronto
comp <- loo::loo_compare(list(equal_var = loo_eq, unequal_var = loo_neq))
comp_df <- as.data.frame(comp)
comp_df
#> elpd_diff se_diff elpd_loo se_elpd_loo p_loo se_p_loo looic
#> equal_var 0.000 0.00 -237 9.76 3.05 0.539 475
#> unequal_var -0.856 0.61 -238 9.81 4.06 0.780 477
#> se_looic
#> equal_var 19.5
#> unequal_var 19.6Per una decisione didatticamente “manuale”, possiamo usare una regola semplice: se |ΔELPD| < 2 × SE(Δ), consideriamo i modelli sostanzialmente equivalenti; in tal caso scegliamo il modello più semplice.
# Piccolo helper decisionale
delta_elpd <- comp_df$elpd_diff[1] # per la riga in cima
se_delta <- comp_df$se_diff[1]
decision <- if (abs(delta_elpd) < 2 * se_delta) {
"Modelli predittivamente equivalenti: preferire la varianza comune per parsimonia."
} else if (delta_elpd > 0) {
"Il modello a varianze distinte offre un vantaggio predittivo credibile: preferirlo."
} else {
"Il modello a varianza comune è migliore: preferirlo."
}
tibble(Delta_ELPD = delta_elpd, SE_Delta = se_delta, Decisione = decision)
#> # A tibble: 1 × 3
#> Delta_ELPD SE_Delta Decisione
#> <dbl> <dbl> <chr>
#> 1 0 0 Il modello a varianza comune è migliore: preferirlo.10.3.3 Posterior predictive checks
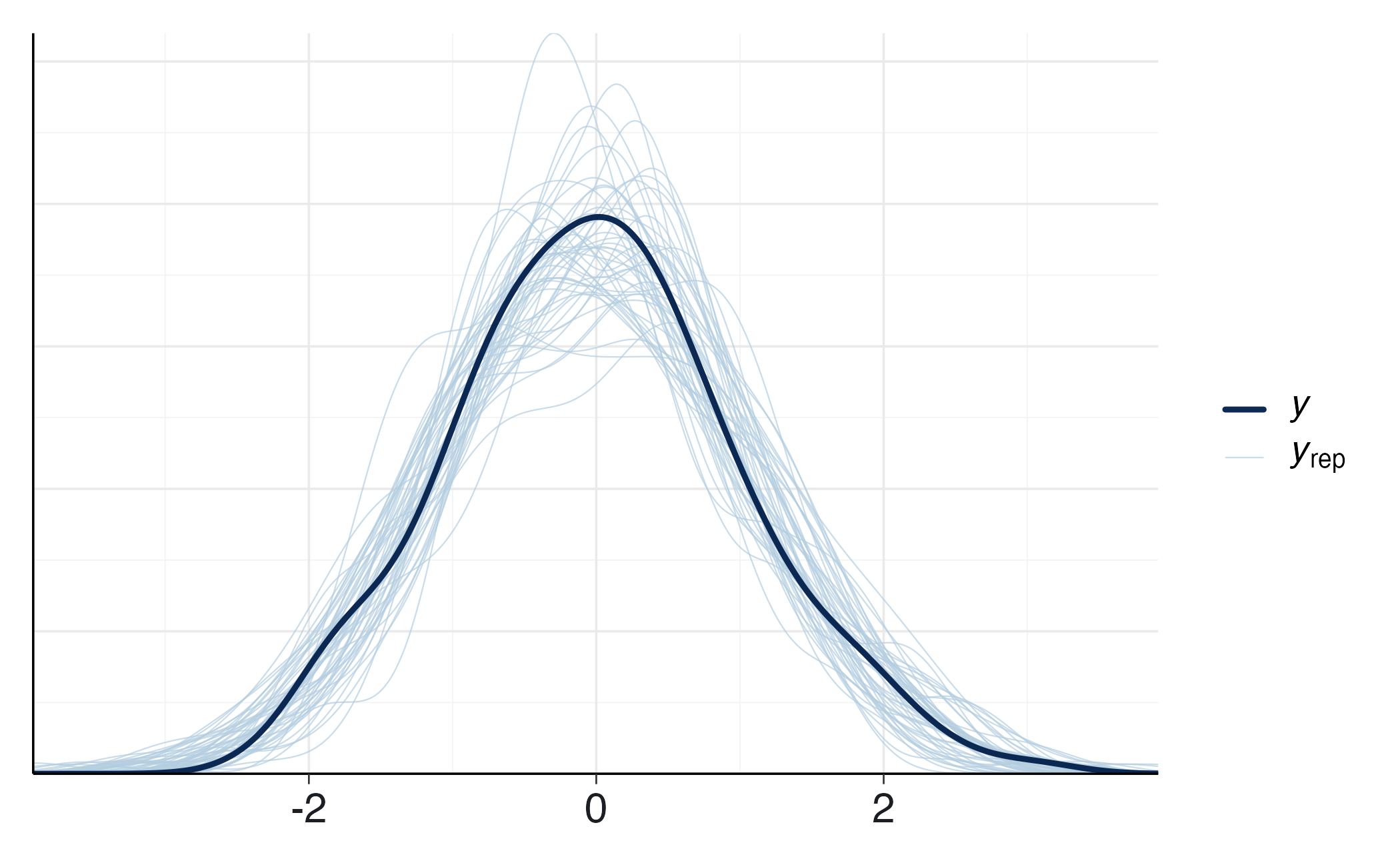
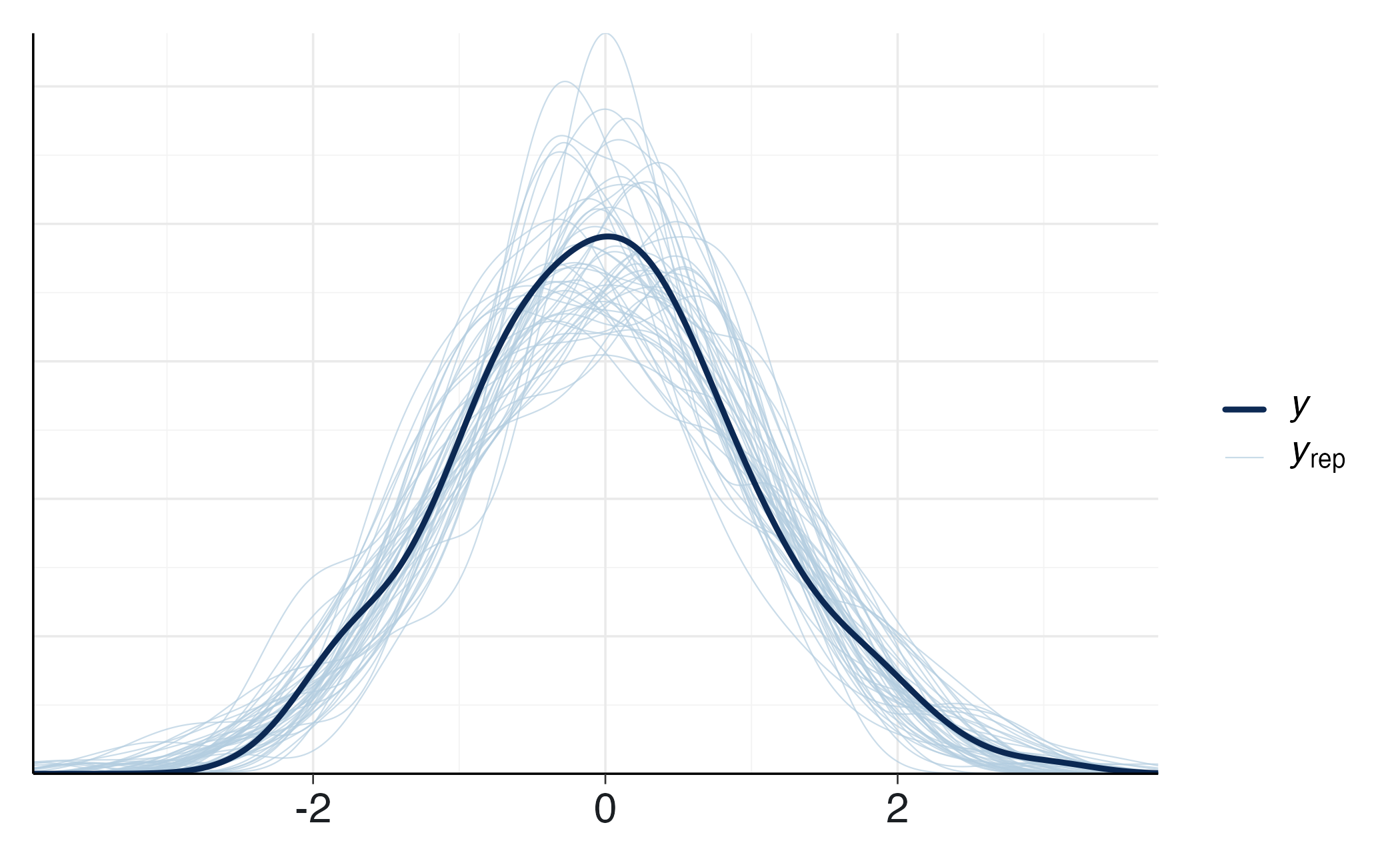
Verifichiamo che i due modelli riproducano la forma della distribuzione osservata. Se entrambi vanno bene, la scelta si gioca sull’ELPD; se uno mostra discrepanze sistematiche, quello è il segnale più forte.
# Equal variance
yrep_eq <- fit_eq$draws("y_rep", format = "matrix")
bayesplot::ppc_dens_overlay(y = stan_data$y, yrep = yrep_eq[1:50, ])# Unequal variances
yrep_neq <- fit_neq$draws("y_rep", format = "matrix")
bayesplot::ppc_dens_overlay(y = stan_data$y, yrep = yrep_neq[1:50, ])10.4 Cosa impariamo
Differenza tra le medie. La posteriore di diff_mu dice di quanto il training modifica l’affetto negativo medio, con intervallo di credibilità; la ROPE ci dice se l’effetto è praticamente trascurabile o rilevante. Questo è un vantaggio diretto rispetto al t-test, che non fornisce probabilità di “effetto rilevante” né gestisce l’equivalenza pratica in modo naturale.
Differenza tra varianze. La posteriore di diff_sigma chiarisce se l’intervento modifica l’eterogeneità delle risposte: informazione spesso ignorata, ma importante in psicologia applicata.
Capacità predittiva. Il confronto PSIS-LOO ci guida nella scelta del livello di complessità. Se ΔELPD è piccolo e incerto, preferiamo la parsimonia (varianza comune); se il vantaggio è stabile e sostanziale, ha senso adottare il modello con varianze distinte.
10.5 Dalle medie di gruppo all’analisi idiografica
L’obiettivo di questo approfondimento è di sposatare l’attenzione dagli effetti medi tra gruppi alla valutazione del cambiamento nel singolo partecipante.
10.5.1 Il modello di misurazione del cambiamento individuale
L’obiettivo consiste nel stimare il cambiamento vero di un individuo a partire dalla differenza osservata tra due misurazioni dello stesso test, tipicamente in contesti pre- e post-intervento. Indichiamo con \(D = X_{\text{post}} - X_{\text{pre}}\) la differenza osservata per una persona. Questo dato apparentemente semplice nasconde in realtà una distinzione fondamentale: mentre \(D\) rappresenta il dato “di superficie”, il cambiamento psicologico vero che vogliamo inferire è \(\Delta\), che risulta contaminato dall’inevitabile errore di misura.
10.5.2 La quantificazione dell’errore di misura
La base concettuale per trattare l’incertezza di misura proviene dalla teoria classica dei test (Lord & Novick, 2008), secondo cui ogni punteggio osservato \(X\) può essere scomposto in componente vera \(T\) ed errore casuale \(E\): \(X = T + E\). L’affidabilità (\(r\)) del test è definita come il rapporto tra varianza vera e varianza osservata: \(r = \mathrm{Var}(T) / \mathrm{Var}(X)\). Da questa relazione discende che la varianza d’errore è \(\mathrm{Var}(E) = \mathrm{Var}(X)(1-r)\), e quindi lo standard error of measurement (SEM) risulta \(\mathrm{SEM} = SD_X \sqrt{1-r}\), dove \(SD_X\) è la deviazione standard campionaria del test.
Per la varianza della differenza tra due misurazioni, il caso più semplice assume che gli errori di pre e post siano indipendenti e con la stessa deviazione standard SEM. Allora la varianza della differenza \(D = X_{\text{post}} - X_{\text{pre}}\) è la somma delle varianze d’errore: \(\mathrm{Var}(D) = \mathrm{Var}(E_{\text{post}}) + \mathrm{Var}(E_{\text{pre}}) = \mathrm{SEM}^2 + \mathrm{SEM}^2 = 2\mathrm{SEM}^2\). La sua deviazione standard è perciò \(\mathrm{SEdiff} = \sqrt{2}\,\mathrm{SEM}\).
10.5.3 Il modello bayesiano per il cambiamento individuale
Il modello probabilistico che collega il dato osservato \(D\) al cambiamento vero \(\Delta\) assume una verosimiglianza normale:
\[ D \mid \Delta \sim \mathcal{N}(\Delta,\mathrm{SEdiff}^2). \]
Questa specificazione implica che, condizionatamente al cambiamento vero \(\Delta\), le differenze osservate si distribuiscono normalmente attorno a \(\Delta\) con varianza determinata dall’errore di misura.
Completiamo il modello con una distribuzione a priori per \(\Delta\), scegliendo una normale centrata sull’assenza di effetto:
\[ \Delta \sim \mathcal{N}(0, \sigma_0^2), \]
dove \(\sigma_0^2\) rappresenta la varianza a priori. La scelta di centrare il prior sullo zero riflette l’assunzione di equiprobabilità tra piccoli miglioramenti e peggioramenti in assenza di dati, mentre \(\sigma_0\) definisce la scala dei cambiamenti considerati plausibili a priori.
10.5.4 L’inferenza e la sua interpretazione clinica
La combinazione di prior normale e verosimiglianza normale produce una distribuzione a posteriori normale per \(\Delta\) con parametri:
\[ \sigma_{\text{post}}^2 = \left( \frac{1}{\sigma_0^2} + \frac{1}{\mathrm{SEdiff}^2} \right)^{-1}, \quad \mu_{\text{post}} = \left( \frac{\sigma_{\text{post}}^2}{\mathrm{SEdiff}^2} \right) D. \]
Queste formule rivelano il meccanismo di regolarizzazione (shrinkage) tipico dell’inferenza bayesiana: la stima finale rappresenta un compromesso ponderato tra il dato osservato e il centro del prior, con pesi determinati dalle rispettive incertezze.
La distribuzione a posteriori permette di rispondere a domande clinicamente rilevanti attraverso il calcolo di probabilità direttamente interpretabili:
- Probabilità di cambiamento trascurabile: area della posteriori nell’intervallo \([-\rho, +\rho]\), dove \(\rho\) definisce la regione di irrilevanza pratica.
- Probabilità di miglioramento rilevante: area della coda destra oltre la soglia clinica \(\tau > 0\).
- Probabilità di peggioramento rilevante: area della coda sinistra sotto \(-\tau\).
10.5.5 Considerazioni metodologiche
Tutta questa costruzione resta volutamente minimale per scopi didattici. In contesti reali possiamo rilassare alcune ipotesi. Se l’affidabilità non è nota con precisione, possiamo trattarla come parametro incerto e propagare questa incertezza nel modello. Se i punteggi hanno outlier o code pesanti, possiamo sostituire la normale con una t di Student per descrivere meglio i dati. Se abbiamo molti soggetti, possiamo passare a un modello gerarchico in Stan in cui i cambiamenti individuali condividono una distribuzione comune: in questo modo le stime idiografiche fanno pooling e risultano più stabili, e al contempo restano coerenti con la stima della differenza media tra gruppi presentata nella prima parte del capitolo. Il punto cruciale, a ogni livello, è che le quantità che interessano davvero — cambiamento individuale, irrilevanza pratica, rilevanza clinica — vengono espresse come probabilità su intervalli nella metrica del problema, con ipotesi esplicite e controllabili.
10.5.5.1 Esempio numerico
Applichiamo il modello a un soggetto del gruppo sperimentale in uno studio sull’intervento mindfulness. I punteggi, espressi in deviazioni standard rispetto alla media normativa, sono: pre = 0.40, post = 0.05. Assumiamo un’affidabilità del test pari a 0.85 e utilizziamo la deviazione standard campionaria dell’intero dataset come metrica di riferimento.
# ------------------------------------------------------------
# RCI bayesiano (versione semplice, chiusa-forma)
# ------------------------------------------------------------
# Likelihood: D = post - pre ~ Normal(Delta, SEdiff^2)
# Prior: Delta ~ Normal(0, prior_sd_delta^2)
# Posterior: Delta | D ~ Normal(m_post, s_post^2)
# ------------------------------------------------------------
rci_bayes <- function(pre, post,
test_sd, reliability,
rope_width = 0.10, # ROPE ±0.10 SD (coerente con capitolo)
tau_clin = 0.30, # soglia clinica 0.30 SD (esempio)
prior_sd_delta = 0.60) {
# 1) Differenza osservata
D <- post - pre
# 2) SEM e SE della differenza
# SEM = SD * sqrt(1 - reliability)
SEM <- test_sd * sqrt(max(0, 1 - reliability))
SEdiff <- sqrt(SEM^2 + SEM^2) # ipotesi: stessa metrica/affidabilità a pre e post
# 3) Prior e posteriori (Normale–Normale)
s0 <- prior_sd_delta
s <- SEdiff
s2_post <- 1 / (1/s0^2 + 1/s^2)
s_post <- sqrt(s2_post)
m_post <- (s2_post / s^2) * D
# 4) Probabilità di interesse
p_in_rope <- pnorm( rope_width, mean = m_post, sd = s_post) -
pnorm(-rope_width, mean = m_post, sd = s_post)
p_gt_tau <- 1 - pnorm(tau_clin, mean = m_post, sd = s_post)
p_lt_neg_tau<- pnorm(-tau_clin, mean = m_post, sd = s_post)
list(
observed_diff = D,
SEM = SEM,
SEdiff = SEdiff,
prior_sd_delta = s0,
post_mean = m_post,
post_sd = s_post,
rope = c(-rope_width, rope_width),
tau_clin = tau_clin,
probs = c(
P_in_ROPE = p_in_rope,
P_diff_gt_tau = p_gt_tau,
P_diff_lt_neg_tau = p_lt_neg_tau
)
)
}# 'df' proviene dalle sezioni precedenti del capitolo (score in SD naturali)
test_sd <- sd(df$score)
res_demo <- rci_bayes(
pre = 0.40,
post = 0.05,
test_sd = test_sd,
reliability = 0.85,
rope_width = 0.10, # ROPE ±0.10 SD (trascurabile)
tau_clin = 0.30, # soglia clinica 0.30 SD (rilevante)
prior_sd_delta = 0.60
)
cat(sprintf(
"\nRCI bayesiano (soggetto esempio)\n---------------------------------\nDiff. osservata (post-pre): %.3f\nSEM: %.3f | SE diff: %.3f\nPosterior Delta ~ Normal(%.3f, %.3f^2)\nROPE: [%.2f, %.2f] | tau_clin: ±%.2f\nP(Delta in ROPE) = %.3f\nP(Delta > tau_clin) = %.3f\nP(Delta < -tau_clin) = %.3f\n",
res_demo$observed_diff, res_demo$SEM, res_demo$SEdiff,
res_demo$post_mean, res_demo$post_sd,
res_demo$rope[1], res_demo$rope[2], res_demo$tau_clin,
res_demo$probs["P_in_ROPE"], res_demo$probs["P_diff_gt_tau"], res_demo$probs["P_diff_lt_neg_tau"]
))
#>
#> RCI bayesiano (soggetto esempio)
#> ---------------------------------
#> Diff. osservata (post-pre): -0.350
#> SEM: 0.384 | SE diff: 0.543
#> Posterior Delta ~ Normal(-0.192, 0.403^2)
#> ROPE: [-0.10, 0.10] | tau_clin: ±0.30
#> P(Delta in ROPE) = 0.175
#> P(Delta > tau_clin) = 0.111
#> P(Delta < -tau_clin) = 0.395Probabilità inferite:
- cambiamento trascurabile: 17.5%;
- peggioramento clinicamente rilevante: 11.1%;
- miglioramento clinicamente rilevante: 39.5%.
Interpretazione clinica.
L’analisi bayesiana delinea un profilo di cambiamento probabilistico che supera le semplici classificazioni dicotomiche. I risultati indicano che il soggetto ha mostrato un miglioramento nell’affetto negativo corrispondente a -0.350 SD, che l’inferenza bayesiana regolarizza a -0.192 SD, integrando l’incertezza di misura con le aspettative a priori.
Il quadro probabilistico risultante evidenzia che il miglioramento clinicamente rilevante è l’ipotesi più plausibile, con una probabilità del 39.5% che il vero cambiamento superi la soglia di rilevanza clinica. Contemporaneamente, la possibilità di un peggioramento sostanziale appare remota (11.1%), mentre l’ipotesi di stabilità risulta poco convincente (17.5%).
Questa caratterizzazione sfumata consente di apprezzare appieno il valore aggiunto dell’approccio bayesiano: non un verdetto assoluto sul cambiamento, ma una mappa probabilistica che quantifica direttamente le ipotesi clinicamente rilevanti. La trasparenza metodologica consente inoltre di valutare criticamente l’influenza dei parametri scelti, quali l’affidabilità dello strumento, l’ampiezza della regione di equivalenza pratica e la soglia di rilevanza clinica, i quali devono essere motivati alla luce della letteratura scientifica e della pratica clinica consolidata.
L’analisi bayesiana del cambiamento individuale non è quindi solo uno strumento statistico, ma una metodologia decisionale che preserva e quantifica sistematicamente l’incertezza, supportando il clinico in valutazioni più articolate e basate sulle evidenze effettive.
Prospettive metodologiche.
La versione dell’analisi idiografica qui presentata, sebbene efficace dal punto di vista didattico, può essere estesa in contesti di ricerca avanzata attraverso implementazioni nel linguaggio Stan. Questa evoluzione metodologica consentirebbe di modellare esplicitamente gli errori di misura - attualmente trattati come quantità fisse - e di sviluppare architetture gerarchiche in grado di integrare le stime individuali con le differenze di gruppo.
L’approccio bayesiano manifesta qui la sua piena coerenza concettuale: sia la stima delle differenze tra gruppi che la valutazione del cambiamento individuale seguono lo stesso schema logico inferenziale. Questa uniformità metodologica garantisce un quadro analitico unificato e robusto per la ricerca psicologica, permettendo di transitare tra analisi di gruppo e valutazioni individuali senza modificare i principi epistemologici di base.
Riflessioni conclusive
L’analisi sviluppata in questo capitolo mette in evidenza i punti di forza dell’approccio bayesiano applicato al confronto tra gruppi e, più in generale, alla valutazione del cambiamento psicologico. La quantità centrale non è più rappresentata da un singolo numero o da un valore-\(p\), ma dall’intera distribuzione a posteriori delle differenze che ci interessano. È questa distribuzione che racconta la storia sostanziale: nel nostro esempio, di quanto il gruppo di mindfulness differisca, in media, da quello di controllo. Non si ottiene soltanto una stima puntuale, ma anche l’incertezza che accompagna ogni possibile valore della differenza. Ciò ci permette di andare oltre il verdetto dicotomico “c’è/non c’è differenza” e di ragionare invece in termini di “gradazioni di plausibilità”.
Un ulteriore vantaggio consiste nella possibilità di valutare la rilevanza pratica degli effetti. L’introduzione di una region of practical equivalence (ROPE) permette di distinguere tra differenze statisticamente rilevanti ma psicologicamente irrilevanti e differenze che invece presentano un impatto sostanziale. Questo approccio consente di rispondere a domande sostanziali per la pratica clinica, quali: “Qual è la probabilità che l’effetto non sia solo diverso da zero, ma anche di entità sufficiente per essere considerato rilevante?”
Il confronto tra modelli con varianza comune e modelli con varianze distinte ci ha mostrato, inoltre, come ogni assunzione statistica vada valutata in termini predittivi. Grazie al criterio ELPD, stimato con PSIS-LOO, abbiamo potuto verificare se la maggiore complessità introdotta dal modello con varianze distinte migliorasse davvero la capacità di predire dati simili ai nostri. Quando il guadagno è minimo, la parsimonia suggerisce di mantenere il modello più semplice, mentre quando il vantaggio predittivo è evidente, si può giustificare la maggiore complessità. Questo modo di ragionare è più trasparente e vicino alla logica scientifica rispetto all’alternativa frequentista, in cui le scelte modellistiche sono spesso delegate a test poco interpretabili.
Il passaggio all’analisi ideografica, con la versione bayesiana del Reliable Change Index, amplia ulteriormente la prospettiva. Se l’analisi di gruppo ci dice cosa accade in media, l’analisi del cambiamento individuale ci mostra quanto un intervento sia stato efficace per ciascun partecipante. In questo caso, il vantaggio del ragionamento bayesiano è evidente: invece di un verdetto rigido (“cambiamento affidabile sì/no”), è possibile stimare la probabilità che un miglioramento sia trascurabile o clinicamente rilevante per il singolo individuo. In questo modo, i due livelli di analisi, collettivo e individuale, non sono più separati, ma si integrano in un quadro unitario.
In sintesi, l’approccio bayesiano ci insegna che l’inferenza non riguarda tanto l’accettazione o il rifiuto di un’ipotesi nulla, quanto piuttosto la descrizione della plausibilità di diversi scenari e il loro collegamento al significato psicologico dei dati. Ciò vale sia per le differenze tra gruppi che per i cambiamenti nei singoli individui. Questo approccio allinea la statistica alle autentiche domande della psicologia. La ricerca non si limita a stabilire se un effetto esista, ma ne indaga l’entità, ne quantifica l’incertezza, ne valuta la rilevanza clinica e arriva a stimare la probabilità che un singolo paziente abbia tratto beneficio dall’intervento. È qui che l’approccio bayesiano mostra il suo valore aggiunto, trasformando l’analisi dei dati in uno strumento flessibile, trasparente e in grado di fornire informazioni sostanziali.
sessionInfo()
#> R version 4.5.1 (2025-06-13)
#> Platform: aarch64-apple-darwin20
#> Running under: macOS Tahoe 26.0.1
#>
#> Matrix products: default
#> BLAS: /Library/Frameworks/R.framework/Versions/4.5-arm64/Resources/lib/libRblas.0.dylib
#> LAPACK: /Library/Frameworks/R.framework/Versions/4.5-arm64/Resources/lib/libRlapack.dylib; LAPACK version 3.12.1
#>
#> locale:
#> [1] C.UTF-8/UTF-8/C.UTF-8/C/C.UTF-8/C.UTF-8
#>
#> time zone: Europe/Zagreb
#> tzcode source: internal
#>
#> attached base packages:
#> [1] stats graphics grDevices utils datasets methods base
#>
#> other attached packages:
#> [1] forcats_1.0.1 cmdstanr_0.9.0 ragg_1.5.0
#> [4] tinytable_0.13.0 withr_3.0.2 systemfonts_1.3.1
#> [7] patchwork_1.3.2 ggdist_3.3.3 tidybayes_3.0.7
#> [10] bayesplot_1.14.0 ggplot2_4.0.0 reliabilitydiag_0.2.1
#> [13] priorsense_1.1.1 posterior_1.6.1 loo_2.8.0
#> [16] rstan_2.32.7 StanHeaders_2.32.10 brms_2.23.0
#> [19] Rcpp_1.1.0 sessioninfo_1.2.3 conflicted_1.2.0
#> [22] janitor_2.2.1 matrixStats_1.5.0 modelr_0.1.11
#> [25] tibble_3.3.0 dplyr_1.1.4 tidyr_1.3.1
#> [28] rio_1.2.4 here_1.0.2
#>
#> loaded via a namespace (and not attached):
#> [1] gridExtra_2.3 inline_0.3.21 sandwich_3.1-1
#> [4] rlang_1.1.6 magrittr_2.0.4 multcomp_1.4-28
#> [7] snakecase_0.11.1 compiler_4.5.1 reshape2_1.4.4
#> [10] vctrs_0.6.5 stringr_1.5.2 pkgconfig_2.0.3
#> [13] arrayhelpers_1.1-0 fastmap_1.2.0 backports_1.5.0
#> [16] labeling_0.4.3 utf8_1.2.6 rmarkdown_2.30
#> [19] ps_1.9.1 purrr_1.1.0 xfun_0.53
#> [22] cachem_1.1.0 jsonlite_2.0.0 broom_1.0.10
#> [25] parallel_4.5.1 R6_2.6.1 stringi_1.8.7
#> [28] RColorBrewer_1.1-3 lubridate_1.9.4 estimability_1.5.1
#> [31] knitr_1.50 zoo_1.8-14 pacman_0.5.1
#> [34] Matrix_1.7-4 splines_4.5.1 timechange_0.3.0
#> [37] tidyselect_1.2.1 abind_1.4-8 yaml_2.3.10
#> [40] codetools_0.2-20 processx_3.8.6 curl_7.0.0
#> [43] pkgbuild_1.4.8 plyr_1.8.9 lattice_0.22-7
#> [46] bridgesampling_1.1-2 S7_0.2.0 coda_0.19-4.1
#> [49] evaluate_1.0.5 survival_3.8-3 RcppParallel_5.1.11-1
#> [52] pillar_1.11.1 tensorA_0.36.2.1 checkmate_2.3.3
#> [55] stats4_4.5.1 distributional_0.5.0 generics_0.1.4
#> [58] rprojroot_2.1.1 rstantools_2.5.0 scales_1.4.0
#> [61] xtable_1.8-4 glue_1.8.0 emmeans_1.11.2-8
#> [64] tools_4.5.1 data.table_1.17.8 mvtnorm_1.3-3
#> [67] grid_4.5.1 QuickJSR_1.8.1 colorspace_2.1-2
#> [70] nlme_3.1-168 cli_3.6.5 textshaping_1.0.4
#> [73] svUnit_1.0.8 Brobdingnag_1.2-9 V8_8.0.1
#> [76] gtable_0.3.6 digest_0.6.37 TH.data_1.1-4
#> [79] htmlwidgets_1.6.4 farver_2.1.2 memoise_2.0.1
#> [82] htmltools_0.5.8.1 lifecycle_1.0.4 MASS_7.3-65