here::here("code", "_common.R") |>
source()
# Load packages
if (!requireNamespace("pacman")) install.packages("pacman")
pacman::p_load(brms, posterior, cmdstanr, tidybayes)62 Zucchero sintattico
“Syntactic sugar: a notation designed to make a language sweeter to use, by providing an alternative, more concise, or more familiar style.”
– Peter J. Landin, The Next 700 Programming Languages (1966)
Introduzione
I modelli lineari sono così ampiamente utilizzati che sono stati sviluppati appositamente una sintassi, dei metodi e delle librerie per la regressione. Una di queste librerie è brms (Bayesian Regression Models using Stan), già introdotta nel Capitolo 61. brms è un pacchetto R progettato per adattare modelli gerarchici generalizzati lineari (di cui il modello lineare bivariato è un caso particolare), utilizzando una sintassi simile a quella presente nei pacchetti R, come lm, lme4, rstanarm, brms si basa su Stan, ma offre un’API di livello superiore.
In questo capitolo esploreremo in maniera dettagliata come condurre un’analisi di regressione utilizzando brms invece di Stan.
Panoramica del capitolo
- Costruire e adattare modelli lineari con la funzione
brm(). - Interpretare i risultati e confrontarli con quelli ottenuti da un approccio frequentista.
- Visualizzare le relazioni stimate e distinguere tra intervalli di credibilità e di predizione.
- Specificare priors personalizzati e valutare l’impatto sui risultati.
- Eseguire verifiche predittive a posteriori (posterior predictive checks).
- Gestire la presenza di outlier tramite la regressione robusta.
- Calcolare e interpretare l’indice di determinazione bayesiano (Bayes R²).
- Accedere e manipolare la distribuzione a posteriori dei parametri.
62.1 Interfaccia brms
Per fare un esempio, applicheremo il modello di regressione bivariato alla relazione tra altezza e peso. I dati contenuti nel file Howell_18.csv sono parte di un censimento parziale della popolazione !Kung San dell’area di Dobe, raccolti tramite interviste condotte da Nancy Howell alla fine degli anni ’60 (McElreath, 2020). I !Kung San sono una delle popolazioni di raccoglitori-cacciatori più conosciute del ventesimo secolo e sono stati oggetto di numerosi studi antropologici. In questa analisi, consideriamo un sottocampione di dati relativi alla popolazione adulta (di età superiore ai 18 anni).
Importiamo i dati contenuti nel file Howell_18.csv.
df |>
head()
#> height weight age male
#> 1 151.8 47.83 63 1
#> 2 139.7 36.49 63 0
#> 3 136.5 31.86 65 0
#> 4 156.8 53.04 41 1
#> 5 145.4 41.28 51 0
#> 6 163.8 62.99 35 1Generiamo un diagramma a dispersione tra le variabili height (altezza) e weight (peso):
ggplot(df, aes(x = weight, y = height)) +
geom_point() +
labs(x = "Weight", y = "Height") 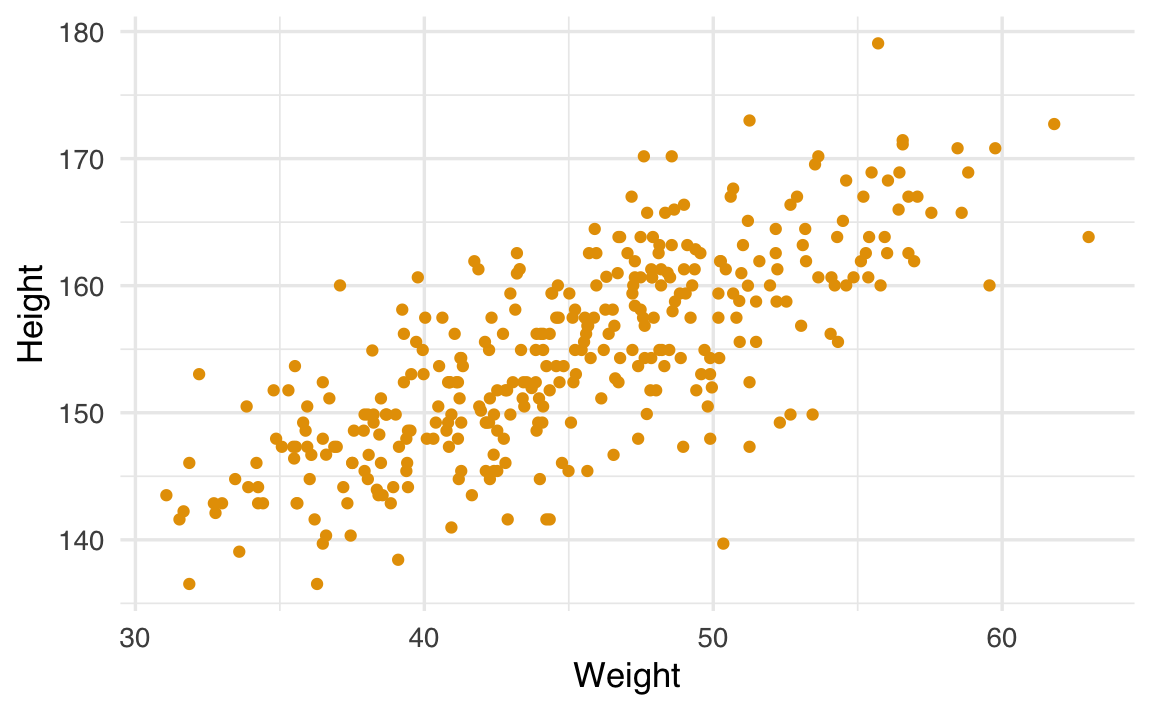
Il pacchetto brms si concentra sui modelli di regressione, e questa specializzazione permette di adottare una sintassi più semplice, conosciuta come sintassi di Wilkinson (Wilkinson & Rogers, 1973). Ad esempio, il modello \(y = \alpha + \beta x + \varepsilon\) si implementa come segue:
a_model = brm(y ∼ 1 + x, data = df)Nella sintassi di Wilkinson, il simbolo tilde (∼) separa la variabile dipendente (a sinistra) dalle variabili indipendenti (a destra). In questo caso, stiamo specificando solo la media (\(\mu\)) della \(y\). brms assume di default che la distribuzione di verosimiglianza sia gaussiana, ma è possibile modificarla tramite l’argomento family.
La notazione 1 si riferisce all’intercetta. L’intercetta viene inclusa di default. Per cui il modello precedente si può anche scrivere, in maniera equivalente, come
a_model = brm(y ∼ x, data = df)Se desideriaamo escludere l’intercetta dal modello, possiamo farlo in questo modo
no_intercept_model = brm(y ∼ 0 + x, data = df)oppure in questo modo
no_intercept_model = brm(y ∼ -1 + x, data = df)Per includere ulteriori variabili nel modello, possiamo procedere così:
model_2 = brm("y ∼ x + z", data)brms consente anche di includere effetti a livello di gruppo (gerarchici). Ad esempio, se desideriamo un modello ad effetti misti nel quale abbiamo un effetto diverso di \(x\) in ciascun gruppo g, possiamo usare la seguente sintassi:
model_h = brm(y ∼ x + z + (x | g), data = df)La sintassi di Wilkinson non specifica le distribuzioni a priori, ma solo come le variabili dipendenti e indipendenti sono collegate. brms definirà automaticamente delle distribuzioni a priori debolmente informative per noi, rendendo superflua la loro definizione esplicita. Tuttavia, se preferiamo avere un maggiore controllo, possiamo specificarle manualmente, come vedremo in seguito.
62.1.1 Centrare le variabili
Per interpretare più facilmente l’intercetta, centriamo la variabile weight rispetto alla media del campione:
df$weight_c <- df$weight - mean(df$weight)Ora, l’intercetta (\(\alpha\)) rappresenterà l’altezza media quando il peso corrisponde alla media del campione.
Adattiamo un modello lineare con la variabile weight centrata e esaminiamo i risultati:
fit_1 = brm(
bf(height ~ 1 + weight_c, center = FALSE),
data = df,
backend = "cmdstanr",
silent = 0
)Utilizzando center = FALSE nel modello Bayesiano garantiamo che il centraggio venga mantenuto e non applicato nuovamente da brms. L’argomento backend = "cmdstanr" indica a brms di usare cmdstan per il campionamento, al posto di Stan che è l’impostazione predefinita. Poiché in questo insegnamento utilizzeremo cmdstan, è essenziale specificare questo backend.
Le tracce dei parametri si ottengono nel modo seguente:
summary(fit_1)
#> Family: gaussian
#> Links: mu = identity; sigma = identity
#> Formula: height ~ 1 + weight_c
#> Data: df (Number of observations: 352)
#> Draws: 4 chains, each with iter = 2000; warmup = 1000; thin = 1;
#> total post-warmup draws = 4000
#>
#> Regression Coefficients:
#> Estimate Est.Error l-95% CI u-95% CI Rhat Bulk_ESS Tail_ESS
#> Intercept 154.59 0.27 154.06 155.11 1.00 3947 2871
#> weight_c 0.91 0.04 0.82 0.99 1.00 3945 2996
#>
#> Further Distributional Parameters:
#> Estimate Est.Error l-95% CI u-95% CI Rhat Bulk_ESS Tail_ESS
#> sigma 5.10 0.20 4.74 5.51 1.00 4902 3275
#>
#> Draws were sampled using sample(hmc). For each parameter, Bulk_ESS
#> and Tail_ESS are effective sample size measures, and Rhat is the potential
#> scale reduction factor on split chains (at convergence, Rhat = 1).La stima dell’intercetta \(\alpha\) = 154.60 suggerisce che, per le persone con un peso corrispondente al valore medio del campione analizzato, l’altezza prevista è di 154.60 cm.
Possiamo confrontare i risultati ottenuti da brm con quelli prodotti dall’approccio frequentista:
fit_2 <- lm(height ~ 1 + weight_c, data = df)summary(fit_2)
#>
#> Call:
#> lm(formula = height ~ 1 + weight_c, data = df)
#>
#> Residuals:
#> Min 1Q Median 3Q Max
#> -19.746 -2.884 0.022 3.142 14.774
#>
#> Coefficients:
#> Estimate Std. Error t value Pr(>|t|)
#> (Intercept) 154.597 0.271 570.2 <2e-16
#> weight_c 0.905 0.042 21.5 <2e-16
#>
#> Residual standard error: 5.09 on 350 degrees of freedom
#> Multiple R-squared: 0.57, Adjusted R-squared: 0.568
#> F-statistic: 463 on 1 and 350 DF, p-value: <2e-16L’uso di prior debolmente informativi fa in modo che i risultati dei due approcci siano praticamente equivalenti.
62.2 Visualizzazione dei risultati
Per comprendere visivamente la relazione stimata tra peso e altezza nel nostro modello bayesiano, utilizziamo la funzione conditional_effects:
conditional_effects(fit_1, effects = "weight_c")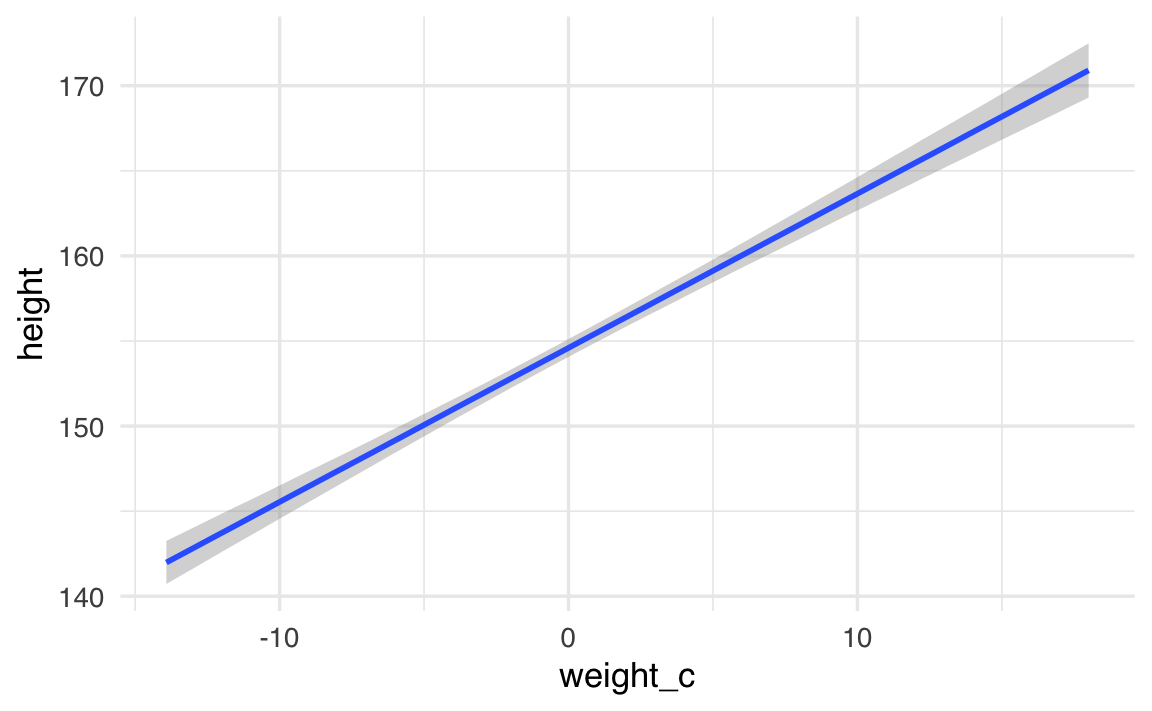
Il grafico generato fornisce una rappresentazione completa della relazione stimata:
- Linea centrale (media posteriore): rappresenta la stima più probabile dell’altezza per ciascun valore del peso centrato.
- Area colorata (intervallo di credibilità): mostra l’intervallo di densità più alta (HDI) al 95%, indicando l’incertezza attorno alla stima centrale.
È possibile adattare il livello di incertezza mostrato modificando l’argomento prob:
# Visualizzazione con intervallo di credibilità all'89%
conditional_effects(fit_1, effects = "weight_c", prob = 0.89)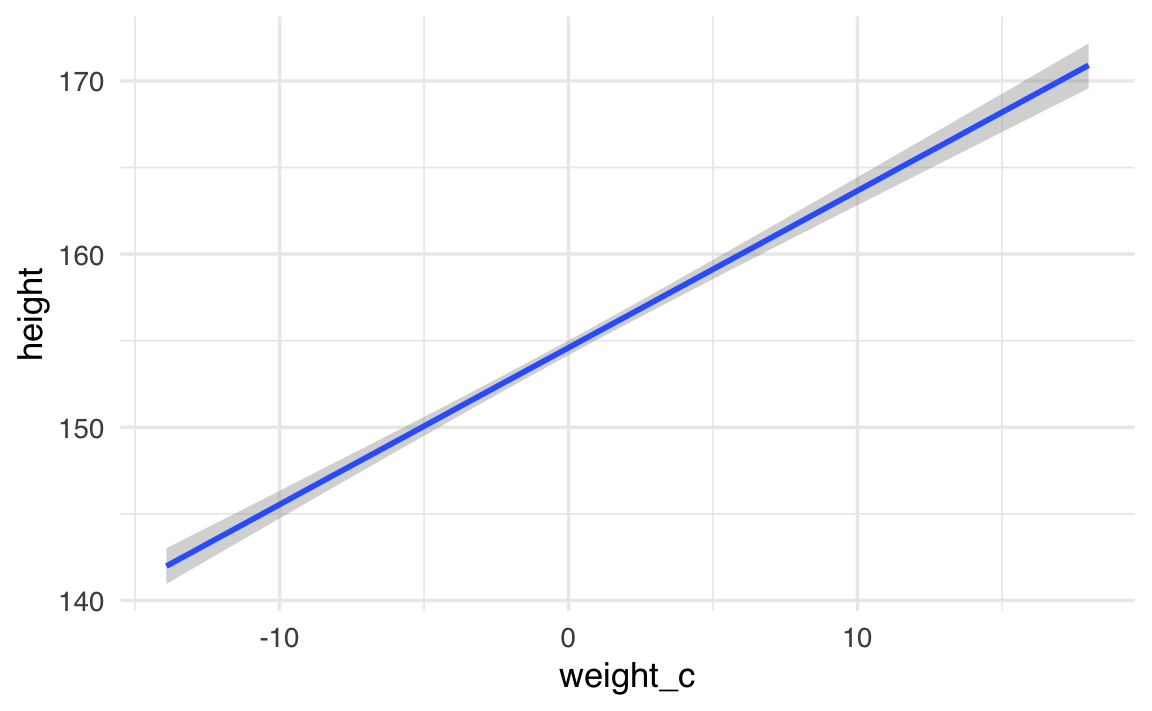
Ridurre il valore di prob (es. a 0.80 o 0.50) produce un intervallo più stretto, mentre aumentarlo (es. a 0.99) amplia l’area di incertezza visualizzata.
62.2.1 Interpretazione pratica del grafico
Nel grafico:
- il punto dove la linea attraversa
weight_c = 0corrisponde all’altezza prevista per un individuo con peso medio (poiché abbiamo centrato la variabile); - la pendenza della linea indica quanto ci aspettiamo che l’altezza aumenti per ogni kg di peso aggiuntivo;
- la larghezza dell’intervallo di credibilità indica la nostra certezza sulle stime: più stretto l’intervallo, maggiore la certezza.
62.3 Due tipi di incertezza nei modelli bayesiani
Immaginiamo di voler capire come il peso (variabile X) sia collegato all’altezza (variabile Y) in un gruppo di persone. Con un modello bayesiano ottieniamo due tipi distinti di informazioni incerte:
| Che cosa stiamo stimando? | Come si chiama l’incertezza? | Che intervallo disegniamo? |
|---|---|---|
| La media “vera” dell’altezza per ogni valore di peso | Incertezza del parametro (o dell’effetto medio) | Intervallo di credibilità (credibility/credible interval) |
| La singola osservazione futura (quanto sarà alta la prossima persona di quel peso) | Incertezza predittiva | Intervallo di predizione (prediction interval) |
62.3.1 Incertezza del parametro – «Quanto stiamo sbagliando la linea media?»
-
Che cosa fa il codice
conditional_effects(fit_1, effects = "weight_c")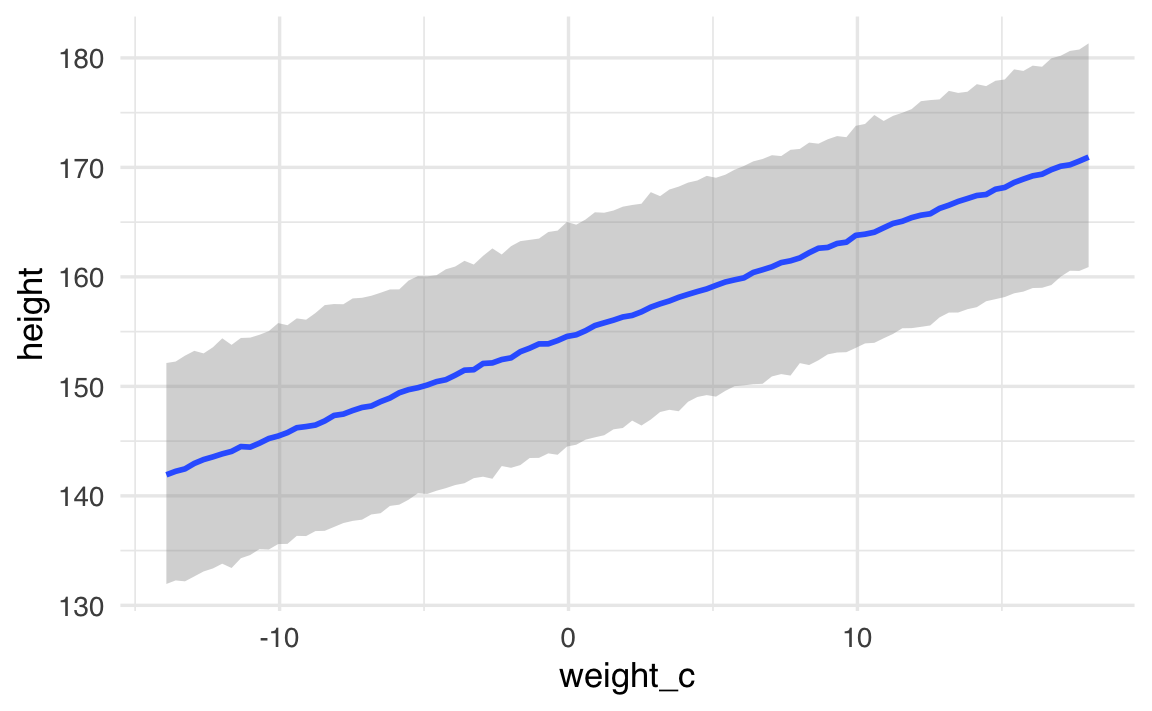
- Disegna la linea di regressione (in pratica: la media stimata dell’altezza per ogni peso).
- Aggiunge intorno una fascia stretta: è l’intervallo di credibilità al 95 %.
- Disegna la linea di regressione (in pratica: la media stimata dell’altezza per ogni peso).
-
Come leggerla
- Se la fascia copre, ad esempio, da 170 cm a 172 cm per un peso di 70 kg, significa che “con il 95 % di probabilità la vera media dell’altezza per quel peso sta lì dentro”.
- Non dice nulla sul fatto che le persone individuali possano essere molto più basse o molto più alte.
- Se la fascia copre, ad esempio, da 170 cm a 172 cm per un peso di 70 kg, significa che “con il 95 % di probabilità la vera media dell’altezza per quel peso sta lì dentro”.
Metafora veloce
Pensa a tirare freccette: la freccia media cade vicino al centro, ma ogni singola freccia può andare ovunque sul bersaglio. L’intervallo di credibilità descrive la posizione del centro.
62.3.2 Incertezza predittiva – «Quanto potrebbe variare la prossima persona?»
-
Che cosa fa il codice
conditional_effects(fit_1, effects = "weight_c", method = "predict")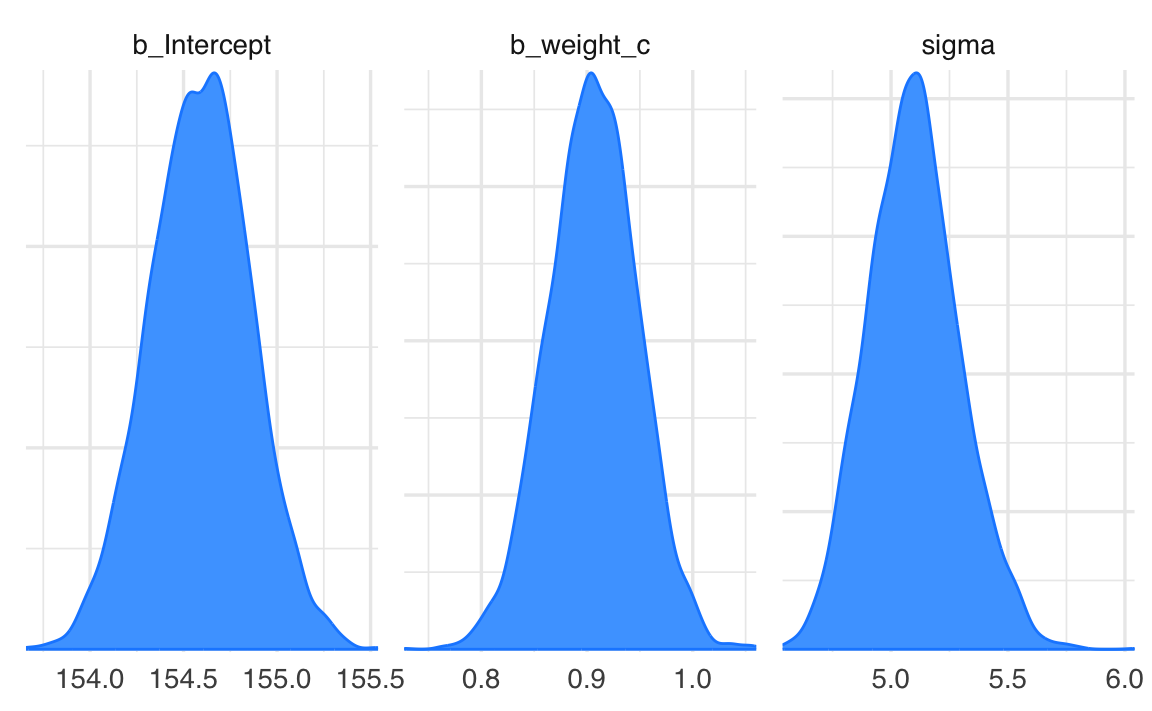
- Ripropone la stessa linea media.
- Disegna però una fascia molto più larga: l’intervallo di predizione.
- Ripropone la stessa linea media.
-
Perché è più largo
- Contiene le due fonti di variabilità:
-
Incertezza sulla linea media (come sopra).
- Variabilità residua: le differenze naturali tra persone di uguale peso (chi è più muscoloso, chi ha ossa più leggere ecc.).
-
Incertezza sulla linea media (come sopra).
- Contiene le due fonti di variabilità:
Metafora veloce
Ora guardi non il centro del bersaglio, ma l’intero disco dove ogni freccia potrebbe atterrare. Quell’area è molto più grande.
62.3.3 Quando usare l’una o l’altra fascia?
| Obiettivo della tua domanda | Funzione da usare | Che fascia guardare |
|---|---|---|
| Capire l’effetto medio (es. “quanto cresce in media l’altezza al crescere di 1 kg?”) | conditional_effects(...) |
Intervallo di credibilità |
| Fare previsioni su nuovi casi (es. “quanto sarà alta Maria che pesa 70 kg?”) | conditional_effects(..., method = "predict") |
Intervallo di predizione |
62.3.4 Riepilogo
-
Credibilità = incertezza sul parametro medio → fascia stretta perché stiamo stimando solo la retta di regressione.
-
Predizione = incertezza su osservazioni future → fascia larga perché somma l’incertezza della retta + la variabilità individuale.
- Scegliamo la visualizzazione che risponde alla nostra domanda: “qual è la media?” (credibilità) o “dove cadrà il prossimo dato?” (predizione).
In sintesi, la differenza principale sta nell’inclusione o meno della variabilità residua. La visualizzazione predittiva è più onesta riguardo alla nostra capacità di fare previsioni per singole osservazioni, mentre quella standard è più adatta per comprendere la relazione generale tra le variabili.
62.4 Distribuzione a posteriori dei parametri
Per esaminare la distribuzione a posteriori dei parametri usiamo la funzione mcmc_plot():
mcmc_plot(fit_1, type = "dens")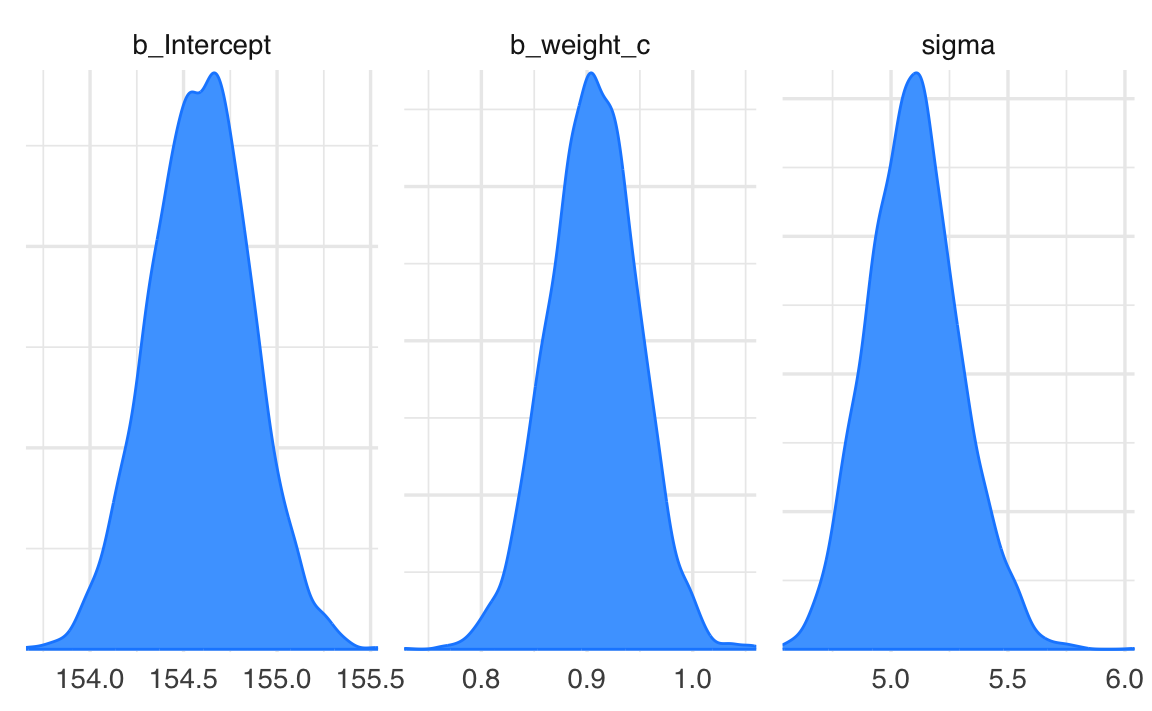
Esaminiamo in maggiori dettagli il sommario numerico dei parametri stimati:
draws <- posterior::as_draws(fit_1, variable = "^b_", regex = TRUE)
posterior::summarise_draws(draws, "mean", "sd", "mcse_mean", "mcse_sd")
#> # A tibble: 2 × 5
#> variable mean sd mcse_mean mcse_sd
#> <chr> <dbl> <dbl> <dbl> <dbl>
#> 1 b_Intercept 154.59 0.27 0.00 0.00
#> 2 b_weight_c 0.91 0.04 0.00 0.00Utilizziamo la funzione as_draws() che trasforma un oggetto R in un formato compatibile con posterior. Gli argomenti variable = "^b_" e regex = TRUEconsentono di selezionare solo i parametri il cui nome inizia con b_: nel nostro caso saranno l’intercetta e la pendenza del modello di regressione lineare.
Successivamente usiamo la funzione summarise_draws() con gli argomenti specificati per un sommario della distribuzione a posteriori dei parametri prescelti.
62.4.1 Spiegazione di mcse_mean e mcse_sd
I valori mcse_mean e mcse_sd sono le Monte Carlo Standard Errors (errori standard Monte Carlo) per la stima della media (mean) e della deviazione standard (sd), rispettivamente. Questi valori quantificano l’incertezza associata al processo di campionamento effettuato durante l’analisi bayesiana, in particolare quando si utilizzano algoritmi Monte Carlo come MCMC (Markov Chain Monte Carlo).
62.4.1.1 mcse_mean
- Rappresenta l’errore standard Monte Carlo per la stima della media.
- Indica quanto la media stimata (\(\text{mean}\)) potrebbe variare a causa della finitezza dei campioni generati dall’algoritmo MCMC.
- Un valore di
mcse_meanbasso rispetto alla deviazione standard (\(\text{sd}\)) suggerisce che il numero di campioni generati è sufficiente per ottenere una stima accurata della media.
62.4.1.2 mcse_sd
- Rappresenta l’errore standard Monte Carlo per la stima della deviazione standard.
- Indica quanto potrebbe variare la stima della deviazione standard (\(\text{sd}\)) a causa del numero finito di campioni generati.
- Anche qui, un valore basso di
mcse_sdrispetto allasdsuggerisce che l’incertezza introdotta dal campionamento è trascurabile.
62.4.2 Come interpretarli?
-
Proporzione rispetto alla
sd:-
mcse_meanemcse_sddovrebbero essere molto più piccoli rispetto ai rispettivi parametri (meanesd), idealmente almeno un ordine di grandezza inferiore. - Ad esempio, per
b_Intercept,mcse_mean= 0.0044 è molto più piccolo rispetto asd= 0.2695, indicando che la stima della media è robusta.
-
-
Indicazione della qualità del campionamento:
- Valori alti di
mcse_meanomcse_sdrispetto allasdpotrebbero indicare che il numero di iterazioni MCMC non è sufficiente, che le catene non sono ben mescolate o che ci sono problemi di convergenza.
- Valori alti di
In sintesi, mcse_mean e mcse_sd sono utili per valutare l’affidabilità delle stime derivate dal campionamento Monte Carlo. Se questi valori sono bassi, possiamo essere confidenti che il numero di campioni è sufficiente per rappresentare accuratamente la distribuzione a posteriori.
62.5 Specificare i priors
Se vogliamo personalizzare i priors, possiamo utilizzare la funzione get_prior per esplorare quelli predefiniti:
get_prior(height ~ 1 + weight_c, data = df)
#> prior class coef group resp dpar nlpar lb ub
#> student_t(3, 154.3, 8.5) Intercept
#> (flat) b
#> (flat) b weight_c
#> student_t(3, 0, 8.5) sigma 0
#> source
#> default
#> default
#> (vectorized)
#> default-
prior: descrive il prior predefinito assegnato a ciascun parametro del modello. Ad esempio:-
student_t(3, 154.3, 8.5): prior t di Student per l’intercetta con 3 gradi di libertà, una media di 154.3, e una scala di 8.5. -
(flat): prior piatto (non informativo) per i coefficienti delle variabili predittive, come \(b\) e \(b_{weight_c}\). -
student_t(3, 0, 8.5): prior t di Student per il parametro \(\sigma\) (deviazione standard residua), centrato su 0 con una scala di 8.5.
-
-
class: identifica la classe di parametro a cui il prior si applica:-
Intercept: prior per l’intercetta (\(\alpha\)). -
b: prior per i coefficienti delle variabili predittive (\(\beta\)). -
sigma: prior per il parametro della deviazione standard residua (\(\sigma\)).
-
-
coef: specifica a quale predittore si riferisce il prior, se applicabile. Ad esempio:- Vuoto per l’intercetta (poiché non dipende da un predittore specifico).
-
weight_cper il coefficiente relativo al predittoreweight_c.
-
lbeub: rappresentano rispettivamente i limiti inferiori (lower bound) e superiori (upper bound) per il prior, se specificati. Ad esempio:- Per
sigma, il limite inferiore è \(0\), dato che la deviazione standard non può essere negativa.
- Per
-
source: indica l’origine del prior. Se il prior è predefinito (default), il valore saràdefault. Se un prior è specificato manualmente dall’utente, sarà indicato come tale.
Ora impostiamo priors espliciti e adattiamo un nuovo modello:
Si noti l’uso di brms::prior(). Questa notazione specifica esplicitamente che stiamo utilizzando la funzione prior() propria del pacchetto brms, evitando potenziali conflitti con funzioni omonime presenti in altre librerie caricate.
fit_2 = brm(
bf(height ~ 1 + weight_c, center = FALSE),
prior = prior_guassian,
data = df,
backend = "cmdstanr",
silent = 0
)Otteniamo un sommario numerico dei parametri stimati:
draws <- posterior::as_draws(fit_2, variable = "^b_", regex = TRUE)
posterior::summarise_draws(draws, "mean", "sd", "mcse_mean", "mcse_sd")
#> # A tibble: 2 × 5
#> variable mean sd mcse_mean mcse_sd
#> <chr> <dbl> <dbl> <dbl> <dbl>
#> 1 b_Intercept 154.61 0.27 0.00 0.00
#> 2 b_weight_c 0.91 0.04 0.00 0.00I prior che abbiamo specificato non cambiano in maniera rilevante la soluzione a posteriori.
62.6 Predizioni predittive a posteriori
Un aspetto fondamentale nella valutazione di un modello statistico, sia frequentista che bayesiano, è verificare quanto bene i dati osservati siano rappresentati dalle predizioni del modello. Tuttavia, l’approccio e l’interpretazione differiscono tra i due paradigmi.
62.6.1 Confronto frequentista
Nel caso frequentista, si confrontano i valori predetti dal modello, \(\hat{y} = \hat{\alpha} + \hat{\beta}x\), con i dati osservati. Questo confronto si basa sull’analisi di:
- la vicinanza della retta di regressione stimata ai dati osservati;
- l’eventuale presenza di pattern nei dati che si discostano da un andamento lineare;
- la variazione della dispersione dei valori di \(y\) rispetto a \(x\) (ad esempio, per verificare l’ipotesi di omoschedasticità).
62.6.2 Approccio bayesiano
Nell’approccio bayesiano, si eseguono le stesse verifiche di base, ma l’analisi si arricchisce attraverso l’uso delle Predizioni Predittive a Posteriori (Posterior Predictive Checks, PPCs). Questo metodo consente di confrontare i dati osservati con dati simulati dal modello, utilizzando l’incertezza stimata nelle distribuzioni a posteriori dei parametri.
62.6.3 Costruzione delle predizioni predittive posteriori
Nel caso di un modello bivariato, il processo per generare un grafico delle Predizioni Predittive a Posteriori è il seguente:
Dati osservati: Si parte dall’istogramma lisciato dei dati osservati, che rappresenta la distribuzione empirica di \(y\).
-
Simulazione di dati predetti:
- Si estrae un campione casuale di valori \(\alpha'\), \(\beta'\), e \(\sigma'\) dalle distribuzioni a posteriori dei parametri (\(\alpha\), \(\beta\), e \(\sigma\)).
- Usando questi valori, si calcolano dati simulati da una distribuzione normale: \[ y_{\text{sim}} \sim \mathcal{N}(\alpha' + \beta'x, \sigma') \] dove \(x\) sono i valori predittori osservati.
Creazione di istogrammi: Per ogni campione simulato, si costruisce un istogramma lisciato che rappresenta la distribuzione predetta dal modello.
Ripetizione: Il processo viene ripetuto più volte, generando molti istogrammi lisciati.
Confronto: Tutti gli istogrammi predetti vengono sovrapposti all’istogramma dei dati osservati. Questo consente di confrontare visivamente la capacità del modello di rappresentare la distribuzione dei dati.
62.6.4 Interpretazione
- Buona corrispondenza: Se gli istogrammi lisciati dei dati simulati si sovrappongono bene all’istogramma dei dati osservati, significa che il modello è in grado di rappresentare adeguatamente il campione corrente.
- Discrepanze: Se vi sono discrepanze sistematiche (ad esempio, picchi o code mancanti nei dati predetti rispetto agli osservati), ciò indica che il modello potrebbe non essere adeguato o che vi sono aspetti dei dati non catturati dal modello.
L’approccio delle Predizioni Predittive a Posteriori è particolarmente potente perché:
- integra l’incertezza nei parametri del modello;
- permette di verificare non solo la bontà di adattamento complessiva, ma anche specifici aspetti delle distribuzioni predette;
- è visivo e intuitivo, facilitando l’identificazione di discrepanze tra modello e dati.
In conclusione, le Predizioni Predittive a Posteriori forniscono un modo robusto per valutare l’adeguatezza di un modello bayesiano rispetto ai dati osservati. Se il modello riproduce bene la distribuzione dei dati osservati, si può concludere che è adatto almeno per il campione corrente. In caso contrario, potrebbe essere necessario rivedere le specifiche del modello, come i priors o la struttura delle variabili.
Verifichiamo dunque le predizioni del modello confrontandole con i dati osservati del campione corrente:
pp_check(fit_2)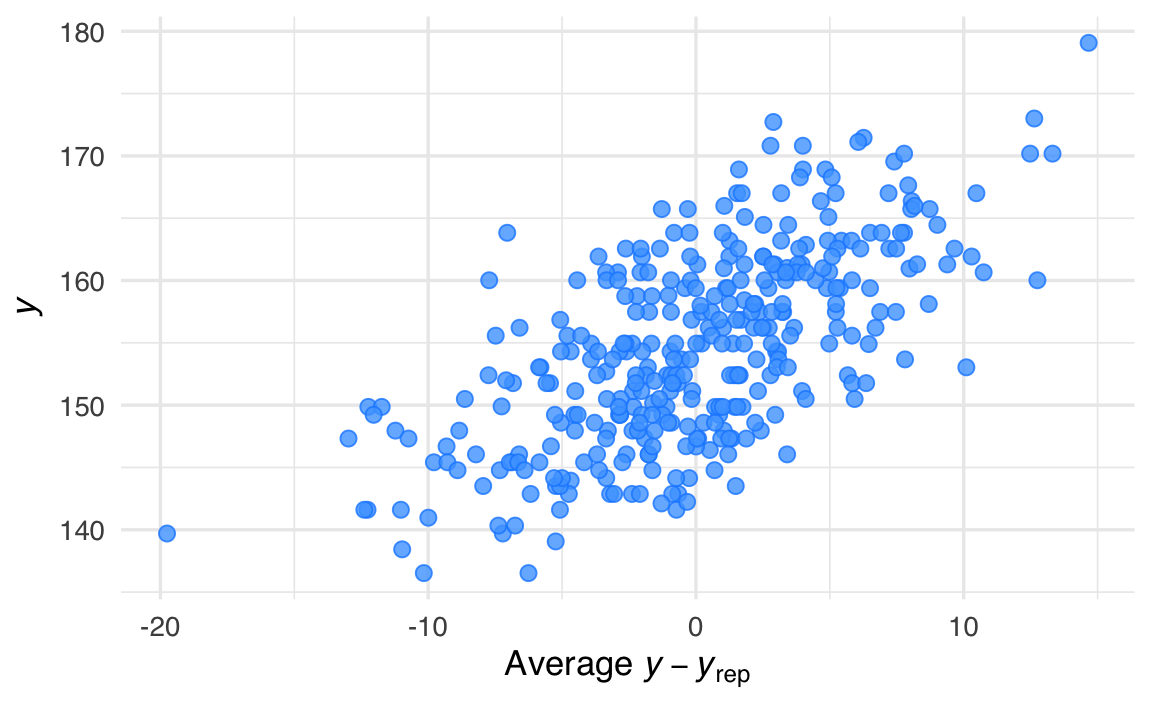
Nel caso presente, vi è una buona corrispondenza tra i dati simulati dal modello e i dati osservati.
Il grafico seguente analizza gli errori del modello rispetto alla retta di regressione stimata.
pp_check(fit_1, type = "error_scatter_avg")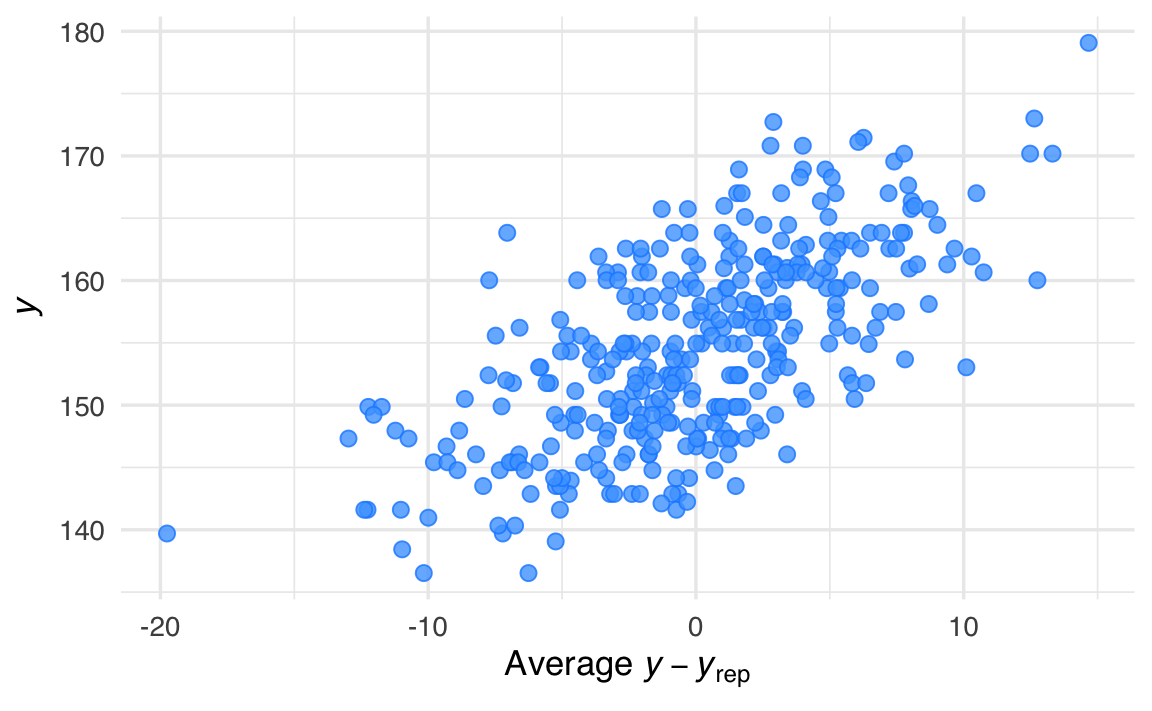
Questo comando utilizza la funzione pp_check() per produrre un grafico che mostra i residui bayesiani, ovvero le differenze tra i dati osservati e quelli predetti dal modello. Nel tipo specifico di grafico scelto ("error_scatter_avg"), i residui sono rappresentati rispetto ai valori predetti, consentendo di valutare visivamente se sono distribuiti in modo uniforme. Dal grafico, si osserva che i residui bayesiani appaiono distribuiti in modo omogeneo rispetto alla retta di regressione (che non è direttamente mostrata nel grafico). Questo suggerisce che:
- Il modello cattura correttamente la relazione tra la variabile predittiva e la variabile di risposta.
- Non ci sono pattern sistematici nei residui, come deviazioni non lineari o variazioni della dispersione (eteroschedasticità).
Se fossero presenti pattern evidenti nei residui (ad esempio, una struttura curva o una variazione sistematica della dispersione), ciò indicherebbe che il modello potrebbe non essere adeguato, richiedendo una rivalutazione della sua struttura (ad esempio, aggiungendo termini non lineari o trasformando le variabili).
62.7 Regressione robusta
In questa sezione introduciamo la regressione robusta. Lo scopo è quello di mostrare quanto sia facile modificare il modello definito da brm per specificare una diversa distribuzione degli errori. Questo non è possibile nel caso dell’approccio frequentista.
I modelli robusti sono utili in presenza di outlier. Ad esempio, introduciamo un outlier nei dati:
df_outlier <- df
df_outlier$height[1] <- 400
df_outlier$weight_c[1] <- -25df_outlier |>
ggplot(aes(x = weight_c, y = height)) +
geom_point() +
labs(x = "Weight", y = "Height") 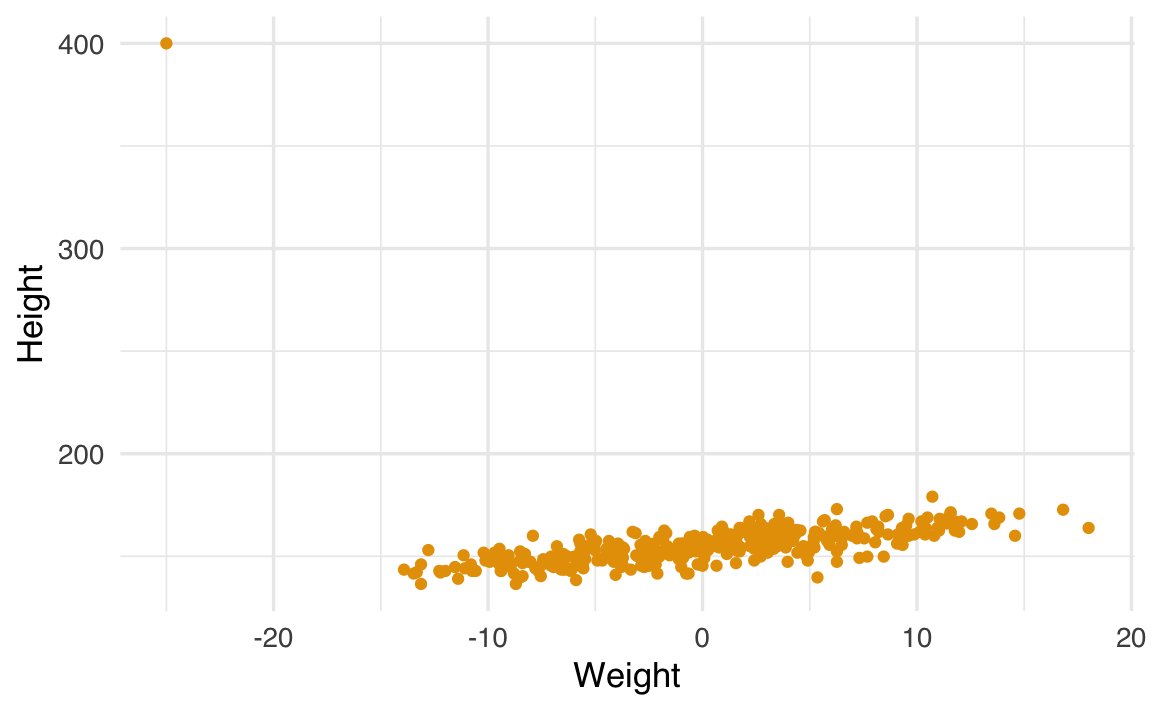
Notiamo come la presenza di un solo outlier introduce una distorsione nei risultati:
fit_3 = brm(
bf(height ~ 1 + weight_c, center = FALSE),
prior = prior_guassian,
data = df_outlier,
backend = "cmdstanr",
silent = 0
)draws <- posterior::as_draws(fit_3, variable = "^b_", regex = TRUE)
posterior::summarise_draws(draws, "mean", "sd", "mcse_mean", "mcse_sd")
#> # A tibble: 2 × 5
#> variable mean sd mcse_mean mcse_sd
#> <chr> <dbl> <dbl> <dbl> <dbl>
#> 1 b_Intercept 155.38 0.82 0.01 0.01
#> 2 b_weight_c 0.47 0.12 0.00 0.00Senza il valore outlier, la stima di beta è circa 0.9.
Adattiamo ora un modello robusto utilizzando una distribuzione \(t\) di Student:
fit_4 = brm(
bf(height ~ 1 + weight_c, center = FALSE),
prior = prior_guassian,
family = student(),
data = df_outlier,
backend = "cmdstanr",
silent = 0
)I risultati mostrano che il modello \(t\) è meno influenzato dagli outlier rispetto al modello gaussiano.
draws <- posterior::as_draws(fit_4, variable = "^b_", regex = TRUE)
posterior::summarise_draws(draws, "mean", "sd", "mcse_mean", "mcse_sd")
#> # A tibble: 2 × 5
#> variable mean sd mcse_mean mcse_sd
#> <chr> <dbl> <dbl> <dbl> <dbl>
#> 1 b_Intercept 154.71 0.27 0.00 0.00
#> 2 b_weight_c 0.93 0.04 0.00 0.00Il parametro \(\nu\) della \(t\) di Student viene stimato dal modello. Nel caso presente
draws <- posterior::as_draws(fit_4, variable = "nu")
posterior::summarise_draws(draws, "mean", "sd", "mcse_mean", "mcse_sd")
#> # A tibble: 1 × 5
#> variable mean sd mcse_mean mcse_sd
#> <chr> <dbl> <dbl> <dbl> <dbl>
#> 1 nu 4.23 0.80 0.01 0.01Con un parametro \(\nu=4\), la distribuzione \(t\) di Student presenta code molto più pesanti rispetto a una gaussiana. Questo la rende più robusta nel gestire la presenza di outliers rispetto al modello gaussiano.
62.8 Indice di determinazione bayesiano
Con il pacchetto brms, possiamo calcolare il Bayes \(R^2\), che rappresenta l’equivalente bayesiano del classico indice di determinazione \(R^2\). Questo indice quantifica la proporzione di varianza spiegata dal modello, tenendo conto dell’incertezza intrinseca delle stime bayesiane.
Il comando per calcolarlo è:
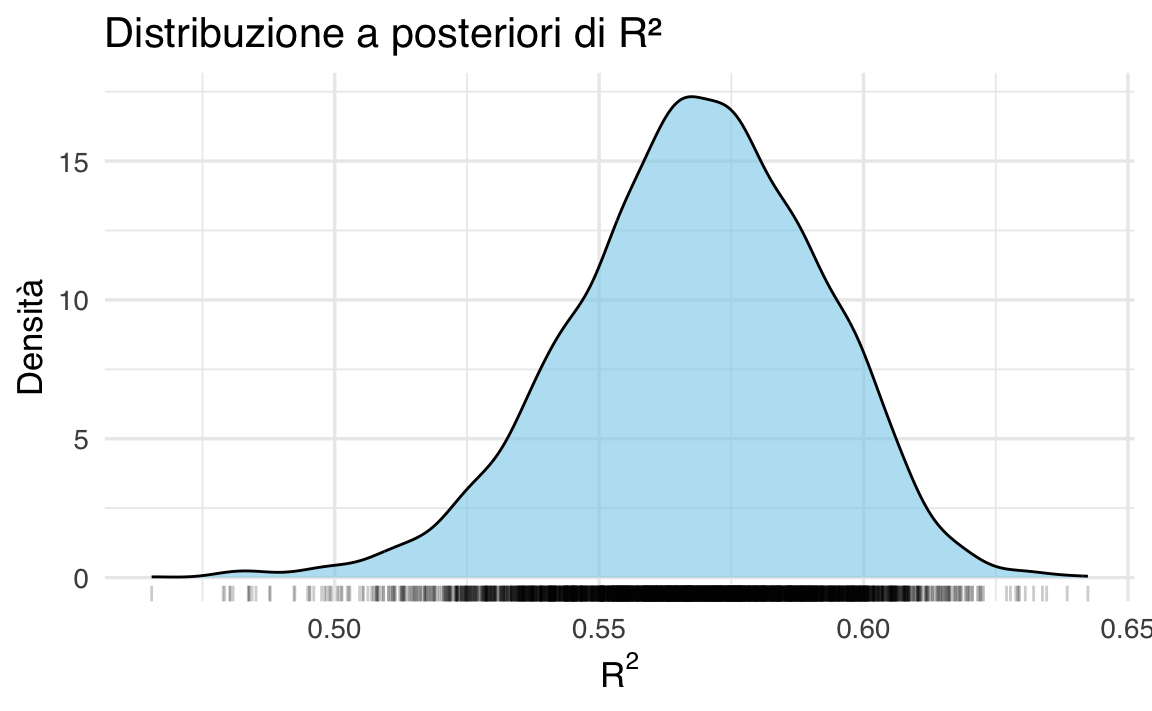
bayes_R2(fit_2)
#> Estimate Est.Error Q2.5 Q97.5
#> R2 0.5686 0.0237 0.519 0.6107Il comando restituisce un tibble (una tabella ordinata) con le seguenti informazioni:
- Estimate: La stima media del Bayes \(R^2\), cioè la proporzione di varianza spiegata dal modello, basata sulle distribuzioni a posteriori dei parametri.
- Est.Error: L’errore standard associato alla stima del \(R^2\).
- Q2.5 e Q97.5: I limiti inferiore e superiore dell’intervallo di credibilità al 95% per il Bayes \(R^2\). Questi valori indicano l’incertezza sul \(R^2\), riflettendo la distribuzione a posteriori.
Nel caso presente
- Stima del \(R^2\): Il modello spiega in media circa il 57% della varianza osservata nella variabile dipendente.
- Errore Standard: L’incertezza sulla stima è relativamente bassa (±0.02).
- Intervallo di Credibilità: C’è un 95% di probabilità che il vero valore del \(R^2\) si trovi tra 0.52 e 0.61.
62.8.1 Differenze rispetto all’indice di determinazione frequentista
- Incertezza: Il Bayes \(R^2\) include un’intera distribuzione a posteriori, permettendo di rappresentare l’incertezza attraverso l’intervallo di credibilità. Questo non è possibile con il \(R^2\) frequentista, che fornisce una stima puntuale.
- Priors: Il Bayes \(R^2\) è influenzato dai priors scelti per i parametri del modello, il che consente una maggiore flessibilità e incorpora conoscenze preesistenti.
In conclusione, il Bayes \(R^2\) è uno strumento potente per valutare l’adattamento di un modello bayesiano, permettendo di quantificare non solo la proporzione di varianza spiegata, ma anche l’incertezza associata alla stima.
r2_draws <- bayes_R2(fit_2, summary = FALSE)r2_df <- data.frame(R2 = as.numeric(r2_draws))ggplot(r2_df, aes(x = R2)) +
geom_density(fill = "skyblue", alpha = 0.6) +
geom_rug(alpha = 0.2) +
labs(
title = "Distribuzione a posteriori di R²",
x = expression(R^2),
y = "Densità"
) 
Lo stesso risultato si ottiene con la funzione mcmc_areas() di bayesplot:
mcmc_areas(r2_draws, prob = 0.95) +
ggtitle("Posterior di R² (area = 95% CI)")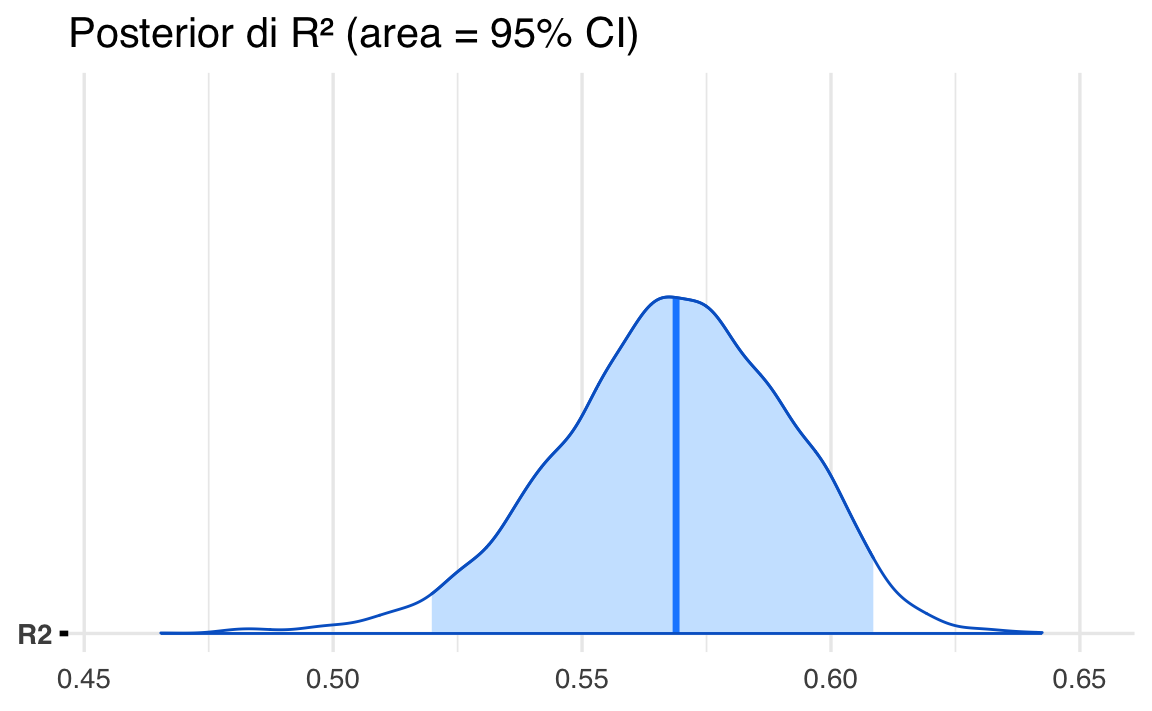
62.9 Approfondimento sulla manipolazione della distribuzione a posteriori con brms
Di seguito illustriamo come accedere e manipolare i campioni generati dal modello bayesiano in brms. Supponiamo di aver costruito un modello lineare semplice, dove vogliamo predire la variabile height in funzione di weight_c:
fit_2 = brm(
bf(height ~ 1 + weight_c, center = FALSE),
prior = prior_guassian,
data = df,
backend = "cmdstanr",
silent = 0
)Una volta che il modello è stato adattato e abbiamo ottenuto l’oggetto fit_2, possiamo estrarre le draws (ossia i campioni) della distribuzione a posteriori tramite la funzione as_draws():
posterior_2 <- as_draws(fit_2)L’oggetto posterior_2 ottenuto è di tipo draws (una struttura definita dal pacchetto posterior), che internamente può essere rappresentato come una lista o un array in cui sono memorizzati i campioni MCMC:
str(posterior_2)
#> List of 4
#> $ 1:List of 5
#> ..$ b_Intercept: num [1:1000] 155 154 154 155 154 ...
#> ..$ b_weight_c : num [1:1000] 0.87 0.922 0.884 0.943 0.906 ...
#> ..$ sigma : num [1:1000] 4.74 5.57 5.25 5.18 5.08 ...
#> ..$ lprior : num [1:1000] -8.61 -8.8 -8.76 -8.69 -8.73 ...
#> ..$ lp__ : num [1:1000] -1080 -1082 -1082 -1079 -1083 ...
#> $ 2:List of 5
#> ..$ b_Intercept: num [1:1000] 154 155 155 155 155 ...
#> ..$ b_weight_c : num [1:1000] 0.999 0.992 0.871 0.933 0.942 ...
#> ..$ sigma : num [1:1000] 4.97 4.9 5.26 4.82 4.94 ...
#> ..$ lprior : num [1:1000] -8.67 -8.64 -8.71 -8.63 -8.63 ...
#> ..$ lp__ : num [1:1000] -1081 -1081 -1079 -1079 -1080 ...
#> $ 3:List of 5
#> ..$ b_Intercept: num [1:1000] 155 155 154 154 154 ...
#> ..$ b_weight_c : num [1:1000] 0.909 0.851 0.953 0.93 0.877 ...
#> ..$ sigma : num [1:1000] 5.53 5.29 5.2 5.25 5.14 ...
#> ..$ lprior : num [1:1000] -8.76 -8.72 -8.74 -8.73 -8.72 ...
#> ..$ lp__ : num [1:1000] -1081 -1079 -1081 -1079 -1080 ...
#> $ 4:List of 5
#> ..$ b_Intercept: num [1:1000] 154 155 155 155 154 ...
#> ..$ b_weight_c : num [1:1000] 0.934 0.93 0.977 0.882 0.923 ...
#> ..$ sigma : num [1:1000] 4.81 5.18 4.87 5 4.97 ...
#> ..$ lprior : num [1:1000] -8.63 -8.67 -8.61 -8.64 -8.7 ...
#> ..$ lp__ : num [1:1000] -1079 -1081 -1083 -1079 -1082 ...
#> - attr(*, "class")= chr [1:3] "draws_list" "draws" "list"Questa ispezione ci permette di vedere che l’oggetto contiene le catene e i parametri campionati. Possiamo estrarre i nomi dei parametri del modello dall’oggetto creato da brm nel modo seguente:
variables(fit_2)
#> [1] "b_Intercept" "b_weight_c" "sigma" "lprior" "lp__"Nel nostro esempio, siamo interessati al coefficiente di regressione associato a weight_c, che in brms è etichettato come b_weight_c. Per semplificare la manipolazione e l’analisi, possiamo servirci di tidybayes, un pacchetto che fornisce funzioni utili per trasformare i campioni in formati “tidy”.
b_slope_draws <- posterior_2 |>
spread_draws(b_weight_c)La funzione spread_draws() estrae e “srotola” le draw dei parametri in un tibble, che risulta più comodo da esplorare:
head(b_slope_draws)
#> # A tibble: 6 × 4
#> .chain .iteration .draw b_weight_c
#> <int> <int> <int> <dbl>
#> 1 1 1 1 0.870
#> 2 1 2 2 0.922
#> 3 1 3 3 0.884
#> 4 1 4 4 0.943
#> 5 1 5 5 0.906
#> 6 1 6 6 0.946In questo modo, ogni riga corrisponde a un singolo campione della catena MCMC, per quel parametro. Una volta estratti i campioni del parametro di interesse (qui b_weight_c), possiamo calcolare facilmente statistiche come quantili e medie:
mean(b_slope_draws$b_weight_c)
#> [1] 0.9059- I quantili a 0.03, 0.50 e 0.97 forniscono, rispettivamente, un limite inferiore al 94% (0.03–0.97), la mediana a posteriori (0.50) e un limite superiore.
- La funzione
mean()restituisce la media a posteriori del coefficiente, un’altra statistica utile per la stima puntuale.
Per comprendere meglio la forma della distribuzione a posteriori, è buona prassi tracciarne la densità. Con tidyverse e tidybayes, possiamo creare un grafico elegante in poche righe:
tibble(beta = b_slope_draws$b_weight_c) %>%
ggplot(aes(x = beta)) +
stat_halfeye(fill = "skyblue", alpha = 0.6) +
labs(
title = "Distribuzione a posteriori di β (b_weight_c)",
x = "Valore di β",
y = "Densità a posteriori"
)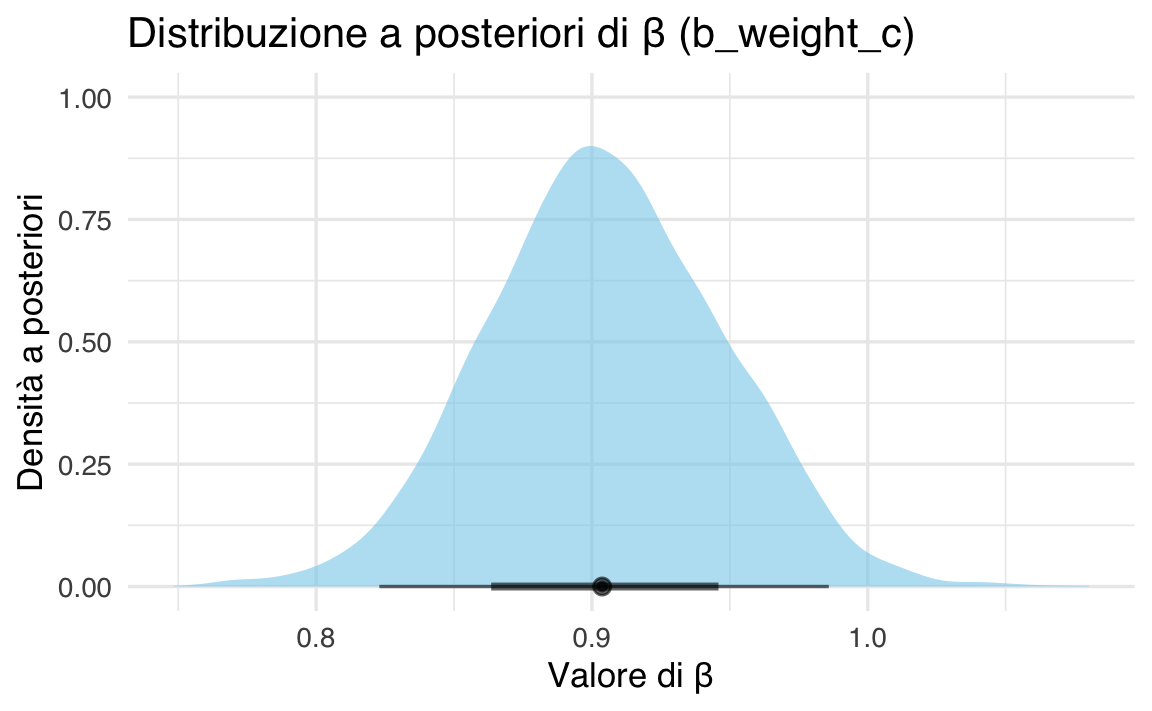
-
stat_halfeye()mostra la densità stimata dei campioni, evidenziando in modo intuitivo i valori più probabili.
- Il colore e l’alpha permettono di personalizzare l’aspetto del grafico.
In sintesi, utilizzando l’oggetto restituito da brm() e la funzione as_draws(), possiamo estrarre i campioni della distribuzione a posteriori e analizzare:
- statistiche di sintesi, come media, mediana e quantili;
- distribuzioni di densità, per una visualizzazione più immediata della variabilità e della forma della distribuzione a posteriori di un parametro.
La combinazione di posterior, tidybayes e tidyverse rende l’intero flusso di lavoro — dall’estrazione dei campioni fino alla creazione di grafici e statistiche — semplice e flessibile.
Riflessioni conclusive
Questo capitolo ha mostrato come utilizzare brms per costruire e interpretare modelli lineari, evidenziando le sue capacità di gestione dei priors, diagnostica e modellizzazione robusta. Grazie alla sua semplicità e flessibilità, brms rappresenta un potente strumento per l’inferenza bayesiana.