here::here("code", "_common.R") |>
source()
# Load packages
if (!requireNamespace("pacman")) install.packages("pacman")
pacman::p_load(mice)46 Distribuzioni coniugate (2)
“The great advantage of conjugate families is that they make Bayesian updating almost automatic.”
– Dennis V. Lindley, Bayesian Statistics: A Review (1972)
Introduzione
Nel capitolo precedente abbiamo esplorato il modello Beta-Binomiale, un esempio paradigmatico di coniugazione nel contesto dei dati discreti. Ora, ci addentriamo nel dominio continuo con il modello Normale-Normale. Grazie alla sua eleganza analitica e alla sua trattabilità, questo modello rappresenta uno strumento cardine nell’inferenza bayesiana.
Consideriamo di avere \(n\) osservazioni \(y_1, \dots, y_n\) che assumiamo essere indipendenti e identicamente distribuite (iid) secondo una distribuzione Normale (o Gaussiana) con media \(\mu\) e varianza \(\sigma^2\), ossia \(y_i \stackrel{\text{iid}}{\sim} \mathcal{N}(\mu, \sigma^2)\). Nell’approccio bayesiano, esprimiamo le nostre conoscenze preliminari su \(\mu\) (e, se necessario, su \(\sigma^2\)) attraverso una distribuzione a priori, tipicamente anch’essa Gaussiana. La magia di questo modello risiede nella coniugazione: se la distribuzione a priori è Normale e la verosimiglianza è Normale, anche la distribuzione a posteriori per \(\mu\) sarà Normale.
L’inferenza bayesiana è un processo iterativo di apprendimento che combina in modo sistematico:
- Convinzioni Iniziali (Distribuzione a Priori): Definiamo una distribuzione a priori per il parametro di nostro interesse, in questo caso la media \(\mu\). Questa riflette la nostra incertezza o conoscenza su \(\mu\) prima di osservare i dati.
- Evidenza Empirica (Funzione di Verosimiglianza): Osserviamo i dati e costruiamo la funzione di verosimiglianza, che ci dice quanto sono probabili i dati osservati per diversi valori possibili di \(\mu\).
- Aggiornamento della Conoscenza (Distribuzione a Posteriori): Applichiamo il teorema di Bayes per combinare la prior e la verosimiglianza. Il risultato è la distribuzione a posteriori, che rappresenta la nostra conoscenza aggiornata su \(\mu\) dopo aver considerato i dati.
Un beneficio chiave di questo approccio è la progressiva riduzione dell’incertezza. All’aumentare del numero di osservazioni, la varianza della distribuzione a posteriori tende a diminuire, fornendo una stima sempre più precisa (cioè con un intervallo di plausibilità più ristretto) per \(\mu\).
In questo capitolo, ci concentreremo sulle famiglie coniugate (per approfondimenti, si veda Capitolo 45), con un focus specifico sul modello Normale-Normale. Vedremo come, partendo da una prior Normale e una verosimiglianza Normale, la distribuzione a posteriori per la media \(\mu\) risulti anch’essa Normale, semplificando notevolmente i calcoli.
Panoramica del capitolo
- introdurre il modello Normale-Normale come esempio di famiglia coniugata;
- mostrare come combinare prior e verosimiglianza per ottenere la distribuzione a posteriori;
- calcolare media e varianza a posteriori in forma chiusa;
- interpretare il ruolo relativo di prior e dati nell’aggiornamento bayesiano;
- applicare il modello a casi concreti (tempi di reazione, punteggi di QI).
46.1 Perché Scegliere una Distribuzione Normale?
La scelta di una distribuzione a priori (e di una verosimiglianza) Normale offre numerosi vantaggi, sia dal punto di vista teorico che pratico:
Simmetria e Adattabilità: La caratteristica forma “a campana” e simmetrica della distribuzione Normale ben si adatta a descrivere molti fenomeni naturali, psicologici e cognitivi, come i tempi di reazione, i punteggi di abilità, o gli errori di misurazione. Questa simmetria facilita l’interpretazione della media \(\mu\) come misura di tendenza centrale e della varianza \(\sigma^2\) come misura della dispersione o incertezza.
Efficienza Parametrica: Nel modello Normale-Normale con varianza nota, l’incertezza sulla media \(\mu\) nella distribuzione a priori è descritta dal singolo parametro \(\sigma_0^2\) (la varianza della prior). Analogamente, la variabilità dei dati è descritta da \(\sigma^2\). Questa parsimonia parametrica semplifica sia la fase di modellizzazione sia la comunicazione dei risultati.
Convergenza con l’Inferenza Classica: Per campioni di dati sufficientemente ampi, le stime bayesiane ottenute con il modello Normale tendono a convergere verso quelle dell’inferenza frequentista. Questa proprietà, legata al teorema di Bernstein-von Mises, è talvolta indicata come calibrazione asintotica e fa sì che il modello Normale-Normale possa agire da ponte tra i due paradigmi inferenziali.
Semplicità Computazionale: Le operazioni matematiche tra distribuzioni Normali (come il prodotto richiesto dal teorema di Bayes) risultano in un’altra distribuzione Normale. Questo permette di ottenere soluzioni analitiche in forma chiusa per i parametri della distribuzione a posteriori, evitando la necessità di ricorrere a metodi di approssimazione numerica complessi, come le simulazioni Monte Carlo Markov Chain (MCMC), almeno nei casi più semplici.
In sintesi: se le nostre conoscenze preliminari suggeriscono una distribuzione unimodale e simmetrica per il parametro di interesse, o se ci aspettiamo che la distribuzione a posteriori abbia tali caratteristiche (cosa spesso favorita dal Teorema del Limite Centrale quando si ha un campione ampio), la distribuzione Normale rappresenta una scelta robusta, elegante e computazionalmente vantaggiosa per condurre un’inferenza rigorosa.
46.2 Inferenza Bayesiana per la Media di una Popolazione Normale (Varianza Nota)
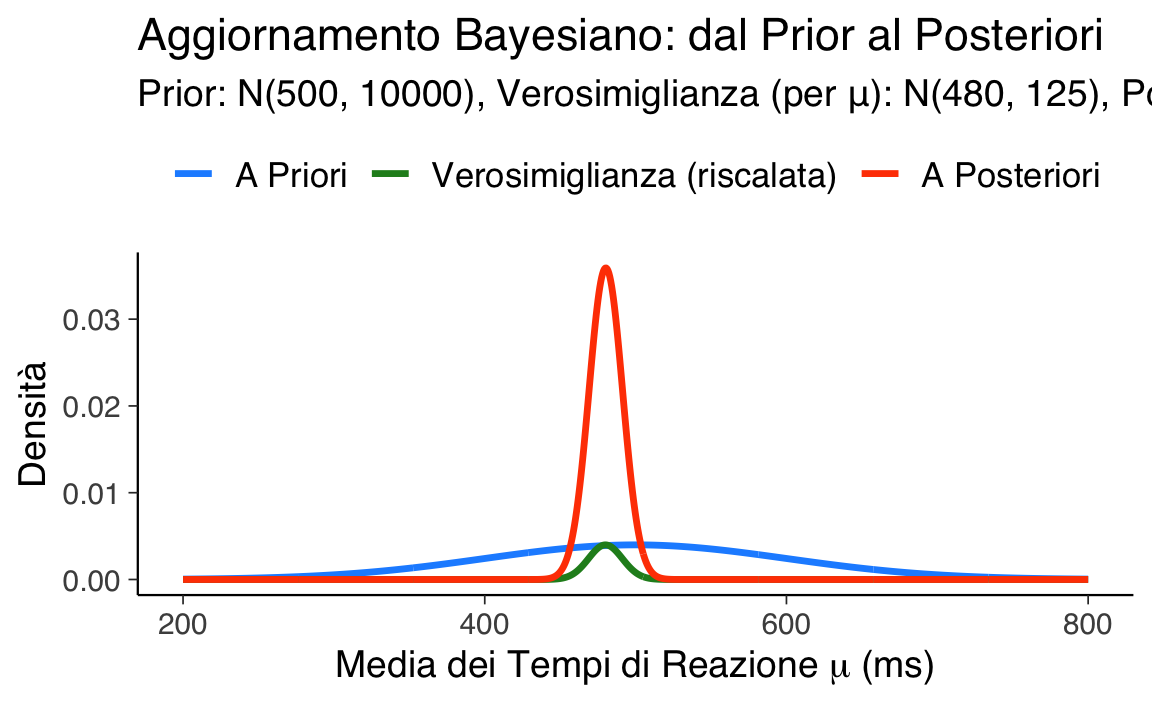
Immaginiamo di voler stimare il tempo medio di reazione \(\mu\) (in millisecondi, ms) di una popolazione di studenti impegnati in un compito Stroop. Supponiamo di aver raccolto i tempi di reazione \(y_1, \dots, y_n\) da un campione di \(n\) studenti. Assumiamo che questi dati provengano da una distribuzione Normale \(y_i \sim \mathcal{N}(\mu, \sigma^2)\) e, per semplificare inizialmente il modello, assumiamo che la varianza \(\sigma^2\) della popolazione sia nota (ad esempio, da studi precedenti o dalla natura standardizzata del compito). Sia \(\sigma = 50\) ms la deviazione standard nota.
46.2.1 I Tre Passi Fondamentali dell’Inferenza Bayesiana
Il processo di inferenza bayesiana si articola nei seguenti passaggi chiave:
| Passo | Significato Intuitivo | Formalizzazione Matematica (Modello Normale-Normale) |
|---|---|---|
| A. Distribuzione a Priori | Le nostre convinzioni iniziali sulla media \(\mu\). | \(\mu \sim \mathcal{N}(\mu_0, \sigma_0^2)\) |
| B. Verosimiglianza dei Dati | L’informazione su \(\mu\) contenuta nei dati osservati. | \(y_i \stackrel{\text{iid}}{\sim} \mathcal{N}(\mu, \sigma^2)\) |
| C. Distribuzione a Posteriori | Le nostre convinzioni aggiornate su \(\mu\) dopo i dati. | \(\mu \mid \mathbf{y} \sim \mathcal{N}(\mu_p, \sigma_p^2)\) |
Quando la varianza \(\sigma^2\) dei dati è nota e la prior per \(\mu\) è Normale, la distribuzione Normale è coniugata per la media \(\mu\). Ciò significa che la distribuzione a posteriori per \(\mu\) sarà anch’essa Normale, mantenendo la stessa forma funzionale attraverso l’aggiornamento bayesiano.
46.2.2 Distribuzione A priori
\(\mu \sim \mathcal{N}(\mu_0,\sigma_0^2)\): descrive dove crediamo sia \(\mu\) e quanta incertezza abbiamo, una varianza grande significa poca informazione.
46.2.3 Verosimiglianza
\[ p(y\mid\mu,\sigma)=\prod_{i=1}^{n}\frac{1}{\sigma\sqrt{2\pi}} \exp\!\Bigl[-\tfrac{(y_i-\mu)^2}{2\sigma^2}\Bigr]. \]
46.2.4 Teorema di Bayes
Il teorema di Bayes combina prior e verosimiglianza attraverso un prodotto ponderato:
\[ p(\mu\mid y)=\frac{p(y\mid\mu)\,p(\mu)}{p(y)} \;\; \propto\;\; \underbrace{\mathcal{N}(\mu_0,\sigma_0^2)}_{\text{prior}} \; \times \; \underbrace{\mathcal{N}(\bar y,\sigma^2/n)}_{\text{verosimiglianza}} . \]
Il prodotto di due distribuzioni gaussiane è una distribuzione gaussiana: basta aggiornare media e varianza.
46.2.5 Media a posteriori
\[ \mu_p=\frac{\tfrac{1}{\sigma_0^2}\,\mu_0 + \tfrac{n}{\sigma^2}\,\bar y} {\tfrac{1}{\sigma_0^2} + \tfrac{n}{\sigma^2}}, \qquad \bar y=\frac{1}{n}\sum_{i=1}^{n}y_i. \tag{46.1}\]
\(\mu_0\): l’idea iniziale.
\(\sigma_0^2\): la fiducia in quell’idea.
\(\bar y\): ciò che dicono i dati.
\(n/\sigma^2\): la quantità di informazione empirica, aumenta con più casi e diminuisce con misure rumorose.
-
Interpretazione: Il peso relativo di prior e dati dipende dalla loro credibilità:
- La prior è influente se ha alta precisione, ovvero 1/\(\sigma_0^2\) è grande, o se ci sono pochi dati, \(n\) piccolo.
- I dati sono dominanti se la prior ha bassa precisione o se c’è un ampio campione.
46.2.6 Varianza a posteriori
\[ \sigma_p^2=\frac{1}{\tfrac{1}{\sigma_0^2}+\tfrac{n}{\sigma^2}}. \tag{46.2}\]
- Proprietà Chiave: \(\sigma_p^2 \le \min(\sigma_0^2, \sigma^2/n)\). L’incertezza diminuisce monotonicamente all’aumentare di \(n\).
- Dialogo Costruttivo: L’inferenza bayesiana è un processo dinamico di dialogo tra le nostre ipotesi iniziali (prior) e l’evidenza empirica (dati/verosimiglianza).
- Calcoli Trasparenti: Con la varianza della popolazione \(\sigma^2\) nota, i calcoli per la media e la varianza a posteriori sono diretti e possono essere eseguiti analiticamente (anche “a mano” per esempi semplici).
- Riduzione Garantita dell’Incertezza: Dopo aver osservato i dati, l’incertezza sul parametro (misurata dalla varianza a posteriori) non può che diminuire o, al limite, rimanere uguale (caso teorico di dati non informativi), rispetto alla varianza a priori.
- Peso dell’Evidenza: Con pochi dati o dati molto “rumorosi” (alta \(\sigma^2\)), la distribuzione a priori esercita un’influenza maggiore sulla stima finale. Con molti dati o dati molto precisi (bassa \(\sigma^2\)), l’informazione proveniente dai dati tende a dominare, e l’influenza della prior sulla stima a posteriori diminuisce.
- Applicabilità Vasta: Lo schema concettuale e matematico del modello Normale-Normale si applica a una vasta gamma di problemi in diverse discipline, inclusa la psicologia sperimentale (es. tempi di reazione, punteggi a test, ampiezze di segnali EEG), l’ingegneria, l’economia, e molte altre aree dove si misurano quantità continue. :::
Riflessioni conclusive
In questo capitolo, abbiamo esplorato il meccanismo dell’aggiornamento bayesiano attraverso l’applicazione del modello Normale-Normale, specificamente nel caso in cui la varianza \(\sigma^2\) della popolazione da cui provengono i dati sia considerata nota.
Il processo inizia con la definizione di una distribuzione a priori per la media incognita \(\mu\). Questa prior, \(\mu \sim \mathcal{N}(\mu_0, \sigma_0^2)\), quantifica le nostre conoscenze o ipotesi iniziali su \(\mu\) attraverso la sua media \(\mu_0\) e la sua varianza \(\sigma_0^2\).
Successivamente, acquisiamo nuovi dati \(y_1, \dots, y_n\), che assumiamo essere campionati da una distribuzione Normale \(\mathcal{N}(\mu, \sigma^2)\). L’informazione contenuta nei dati viene sintetizzata dalla media campionaria \(\bar{y}\) e dalla dimensione del campione \(n\), che, insieme alla varianza nota \(\sigma^2\), determinano la forma della funzione di verosimiglianza (proporzionale a \(\mathcal{N}(\bar{y}, \sigma^2/n)\) come funzione di \(\mu\)).
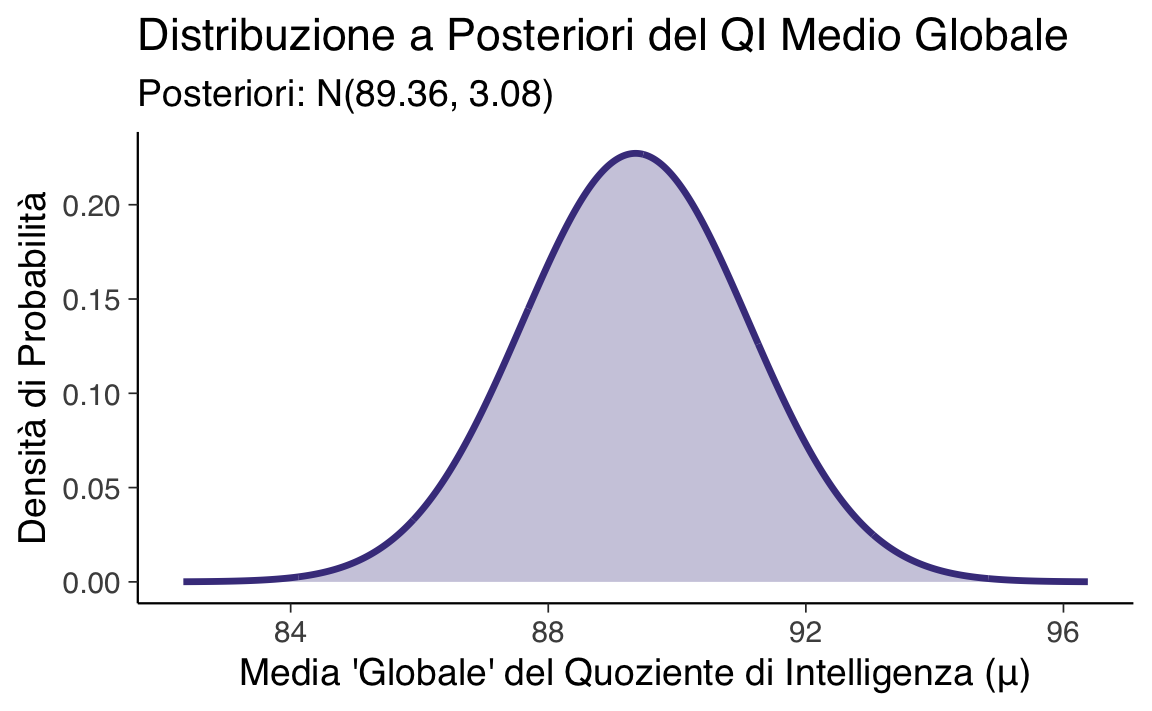
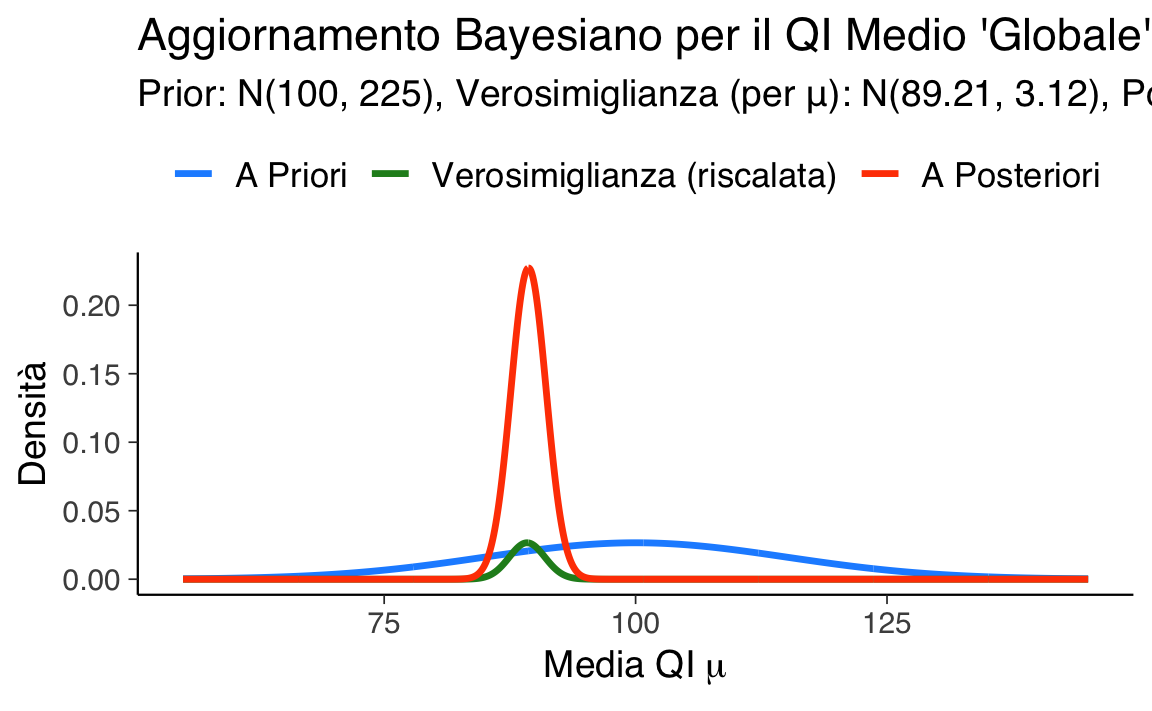
Applicando il Teorema di Bayes, combiniamo la prior e la verosimiglianza. Grazie alla proprietà di coniugatezza tra la distribuzione Normale (per la prior) e la verosimiglianza Normale (per i dati), la distribuzione a posteriori per \(\mu\) risulta anch’essa Normale: \(\mu \mid \mathbf{y} \sim \mathcal{N}(\mu_n, \sigma_n^2)\).
La media a posteriori \(\mu_n\) è una media ponderata della media a priori \(\mu_0\) e della media campionaria \(\bar{y}\). I pesi sono determinati dalle rispettive precisioni (il reciproco delle varianze):
\[ \mu_n = \frac{\left(\frac{1}{\sigma_0^2}\right)\mu_0 + \left(\frac{n}{\sigma^2}\right)\bar{y}}{\frac{1}{\sigma_0^2} + \frac{n}{\sigma^2}} \]
La varianza a posteriori \(\sigma_n^2\) è calcolata in modo che la sua precisione sia la somma delle precisioni della prior e dei dati:
\[ \frac{1}{\sigma_n^2} = \frac{1}{\sigma_0^2} + \frac{n}{\sigma^2} \quad \implies \quad \sigma_n^2 = \left(\frac{1}{\sigma_0^2} + \frac{n}{\sigma^2}\right)^{-1} \]
Un aspetto fondamentale è che la varianza a posteriori \(\sigma_n^2\) è sempre minore o uguale sia a \(\sigma_0^2\) sia a \(\sigma^2/n\), indicando che l’incorporazione dei dati porta a una riduzione (o al più, a nessuna variazione) dell’incertezza su \(\mu\).
In sintesi, il modello Normale-Normale con varianza nota offre un quadro analiticamente trattabile e intuitivo per l’aggiornamento bayesiano. La coniugatezza semplifica i calcoli, permettendo di concentrarsi sull’interpretazione di come le credenze iniziali vengono modificate dall’evidenza empirica. Sebbene l’assunzione di varianza nota sia una semplificazione, questo modello getta le basi per comprendere modelli più complessi in cui anche la varianza è incognita e stimata.