here::here("code", "_common.R") |>
source()
# Load packages
if (!requireNamespace("pacman")) install.packages("pacman")
pacman::p_load(mice)45 Distribuzioni coniugate (1)
“Conjugate families are not chosen because they are realistic, but because they allow us to see the Bayesian machinery at work in its simplest form.”
– Morris H. DeGrootx, Optimal Statistical Decisions (1970)
Introduzione
In questo capitolo, esploriamo il concetto di distribuzioni a priori coniugate e il loro ruolo nell’inferenza bayesiana. Utilizzando il modello beta-binomiale come esempio paradigmatico, dimostreremo come queste distribuzioni semplificano l’analisi attraverso calcoli analitici diretti. L’uso di una distribuzione a priori coniugata non solo rende l’inferenza più agevole, ma fornisce anche una chiara visione del modo in cui le credenze a priori influenzano le conclusioni.
Panoramica del capitolo
- introduzione del modello beta-binomiale,
- analisi della distribuzione Beta e del suo ruolo come distribuzione a priori,
- descrizione del processo di aggiornamento bayesiano e dei vantaggi derivanti dall’uso di distribuzioni coniugate.
45.1 Il modello Beta-Binomiale
Il modello beta-binomiale è un esempio classico per analizzare una proporzione \(\theta\), ossia la probabilità di successo in una sequenza di prove binarie (ad esempio, successo/fallimento). Supponiamo di osservare \(y\) successi su \(n\) prove, dove ogni prova è indipendente e con la stessa probabilità di successo \(\theta\), che appartiene all’intervallo \([0,1]\).
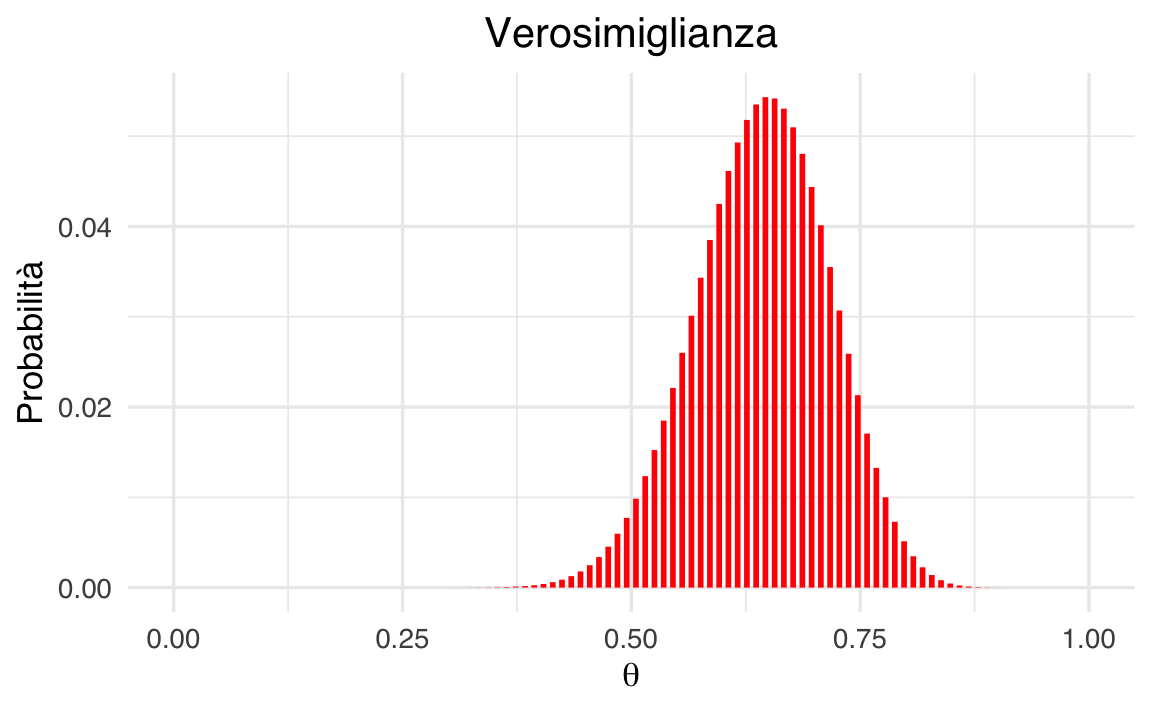
La funzione di verosimiglianza, basata sulla distribuzione binomiale, è espressa come:
\[ \mathcal{Binomial}(y \mid n, \theta) = \binom{n}{y} \theta^y (1 - \theta)^{n - y}, \]
dove \(\binom{n}{y}\) è il coefficiente binomiale che conta il numero di modi in cui \(y\) successi possono verificarsi in \(n\) prove.
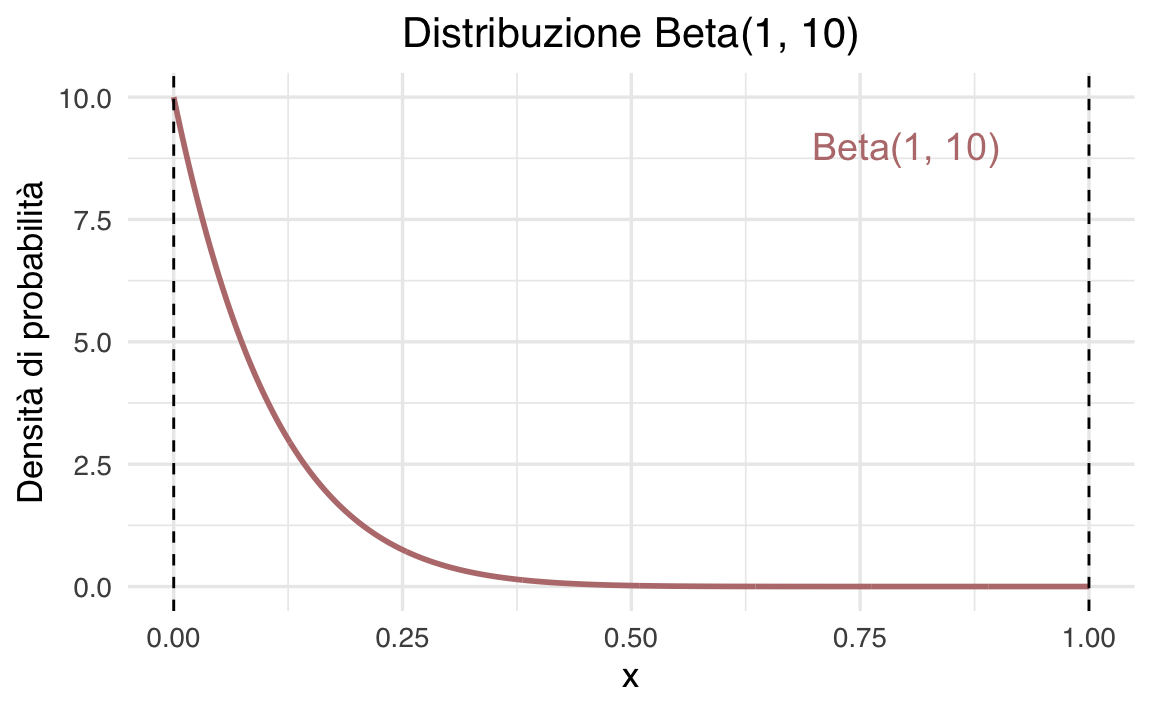
Per modellare la nostra conoscenza preliminare su \(\theta\), scegliamo una distribuzione a priori Beta, che rappresenta un’ampia gamma di credenze iniziali con parametri flessibili.
45.2 La distribuzione Beta
La distribuzione Beta è definita come:
\[ \mathcal{Beta}(\theta \mid \alpha, \beta) = \frac{1}{B(\alpha, \beta)} \theta^{\alpha - 1} (1 - \theta)^{\beta - 1}, \quad \text{con } \theta \in (0, 1), \]
dove:
\(\alpha > 0\) e \(\beta > 0\) sono i parametri che determinano la forma della distribuzione,
-
\(B(\alpha, \beta)\) è la funzione Beta di Eulero, calcolata come:
\[ B(\alpha, \beta) = \frac{\Gamma(\alpha)\Gamma(\beta)}{\Gamma(\alpha + \beta)}, \]
dove \(\Gamma(x)\) è la funzione Gamma, una generalizzazione del fattoriale.
In termini bayesiani, possiamo pensare a questi parametri nel modo seguente:
- \(\alpha -1\) rappresenta il numero ipotetico di “successi” a priori,
- \(\beta -1\) rappresenta il numero ipotetico di “fallimenti” a priori.
Ad esempio:
- una distribuzione Beta(1, 1) è uniforme (0 successi a priori e 0 fallimenti), indicando totale incertezza iniziale (assenza di credenze informate);
- una distribuzione Beta(10, 20) rappresenta una conoscenza a priori basata su 9 successi e 19 fallimenti ipotizzati, indicando una convinzione iniziale relativamente solida, poiché deriva da un totale di 28 osservazioni virtuali che riflettono le nostre credenze precedenti.
Questa interpretazione consente di calibrare le credenze a priori in base all’evidenza disponibile o alla fiducia nella stima.
La distribuzione Beta è estremamente versatile:
- valori diversi di \(\alpha\) e \(\beta\) producono distribuzioni simmetriche, asimmetriche o uniformi;
- valori elevati di \(\alpha\) e \(\beta\) riducono la varianza, riflettendo credenze più forti.
Questa flessibilità rende la distribuzione Beta una scelta ideale per rappresentare credenze iniziali sulle proporzioni.
45.3 Aggiornamento bayesiano
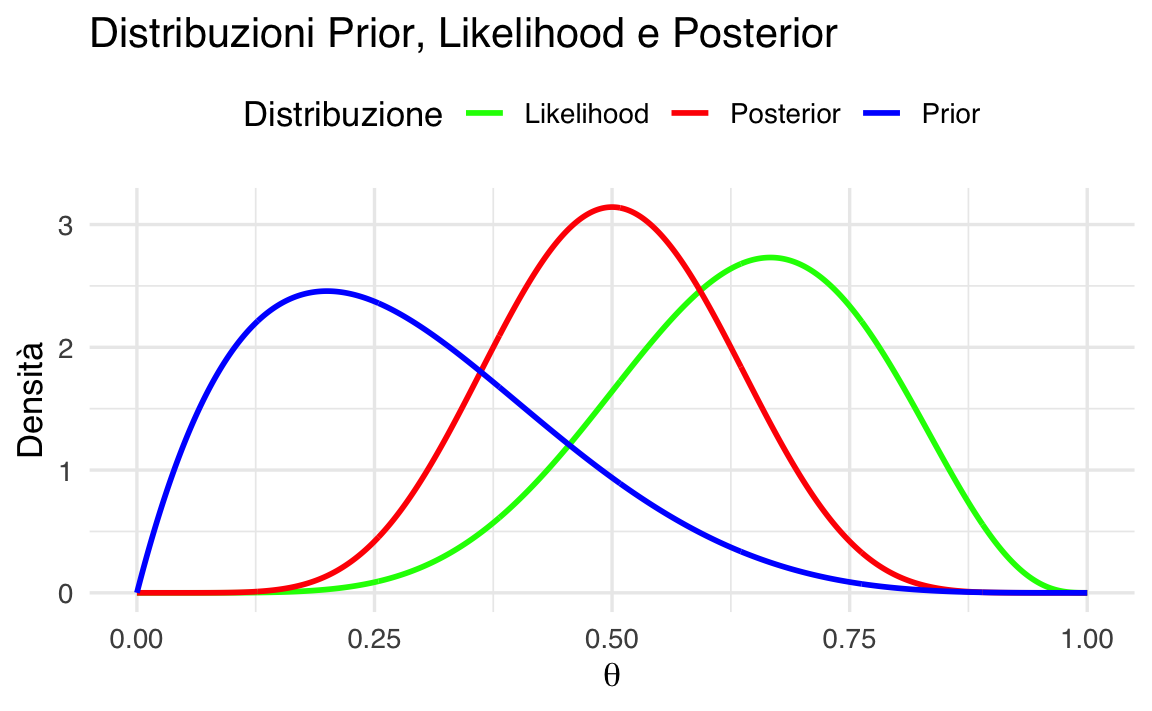
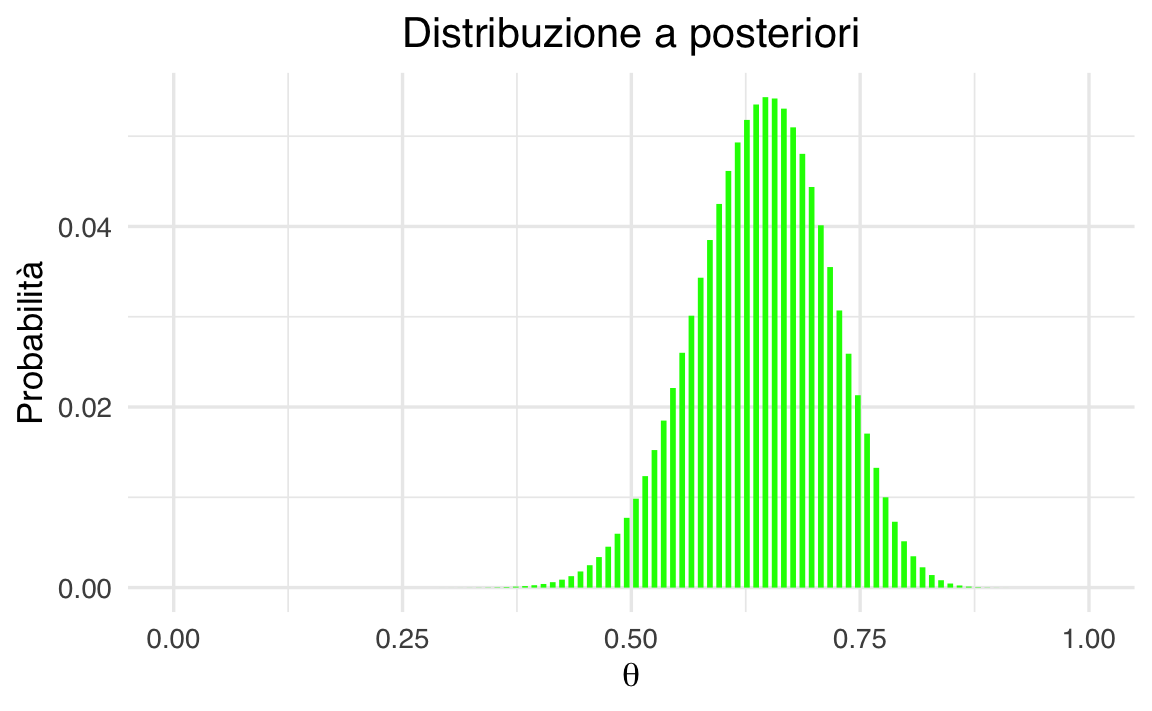
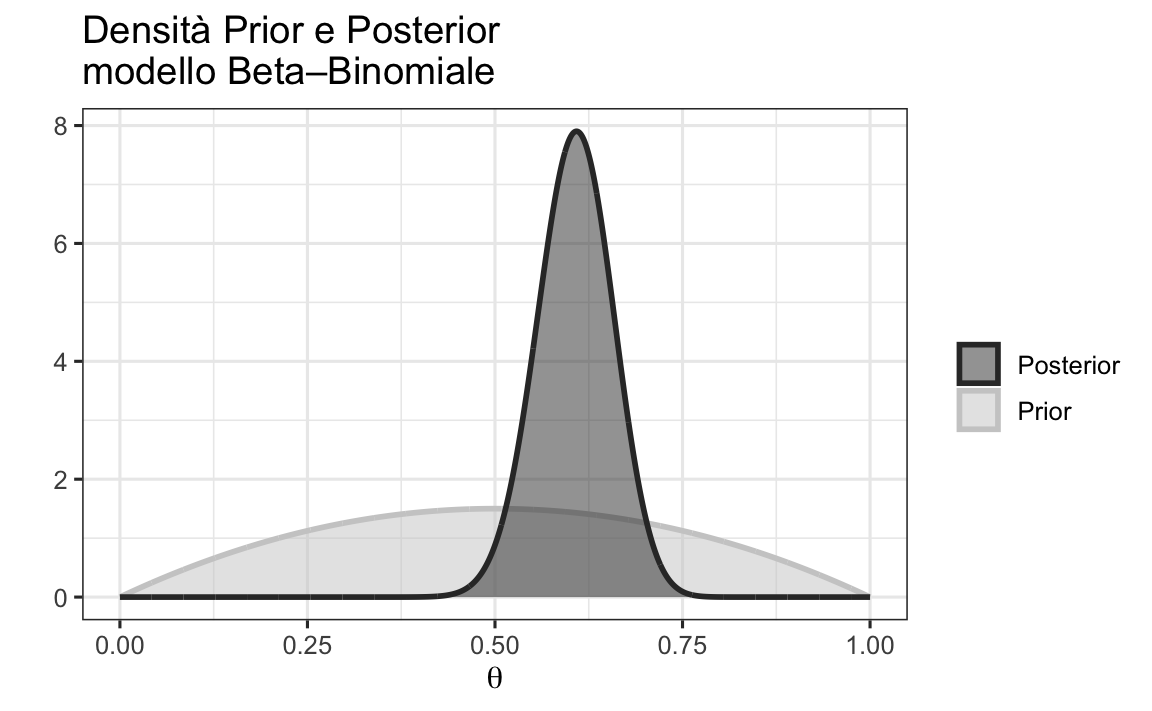
L’aggiornamento bayesiano combina le informazioni iniziali (distribuzione a priori) con i dati osservati (verosimiglianza) per produrre una nuova distribuzione delle nostre credenze (distribuzione a posteriori). Nel caso del modello beta-binomiale, questo processo è particolarmente semplice grazie alla “coniugazione”: il prior Beta e la verosimiglianza Binomiale producono una distribuzione a posteriori che appartiene ancora alla famiglia Beta.
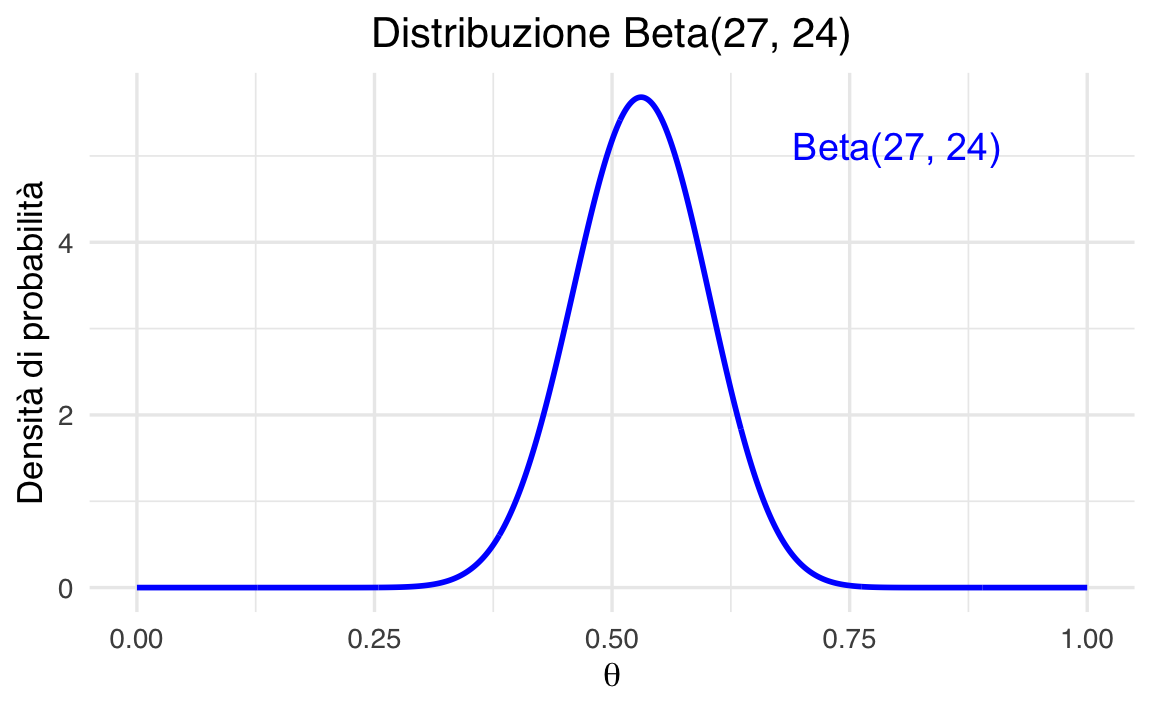
Teorema 45.1 Sia \(Y\sim\mathrm{Binomial}(n,\theta)\) il numero di successi \(y\) in \(n\) prove indipendenti con probabilità di successo \(\theta\), e sia la nostra distribuzione a priori su \(\theta\) una Beta\(\bigl(\alpha,\beta\bigr).\) Allora la distribuzione a posteriori di \(\theta\) dato l’osservazione \(Y=y\) è
\[ \theta \mid Y=y \;\sim\; \mathrm{Beta}\bigl(\alpha + y,\;\beta + (n - y)\bigr), \tag{45.1}\]
ovvero i parametri si aggiornano come
\[ \alpha' = \alpha + y, \quad \beta' = \beta + n - y. \tag{45.2}\]
45.3.1 Vantaggi del modello Beta-Binomiale
- Semplicità analitica: la coniugatezza della distribuzione Beta-Binomiale semplifica i calcoli, rendendo immediato l’aggiornamento dei parametri.
- Interpretazione intuitiva: l’aggiornamento dei parametri \(\alpha\) e \(\beta\) mostra in modo trasparente come i dati influenzino le credenze.
In sintesi, il modello Beta-Binomiale è un esempio didattico fondamentale per comprendere l’inferenza bayesiana e rappresenta un punto di partenza ideale per approcci più avanzati.
45.4 Principali distribuzioni coniugate
Esistono altre combinazioni di verosimiglianza e distribuzione a priori che producono una distribuzione a posteriori con la stessa forma della distribuzione a priori. Ecco alcune delle più note coniugazioni tra modelli statistici e distribuzioni a priori:
Nel modello Normale-Normale \(\mathcal{N}(\mu, \sigma^2_0)\), la distribuzione a priori è \(\mathcal{N}(\mu_0, \tau^2)\) e la distribuzione a posteriori è \(\mathcal{N}\left(\frac{\mu_0\sigma^2 + \bar{y}n\tau^2}{\sigma^2 + n\tau^2}, \frac{\sigma^2\tau^2}{\sigma^2 + n\tau^2} \right)\).
Nel modello Poisson-gamma \(\text{Po}(\theta)\), la distribuzione a priori è \(\Gamma(\lambda, \delta)\) e la distribuzione a posteriori è \(\Gamma(\lambda + n \bar{y}, \delta +n)\).
Nel modello Esponenziale \(\text{Exp}(\theta)\), la distribuzione a priori è \(\Gamma(\lambda, \delta)\) e la distribuzione a posteriori è \(\Gamma(\lambda + n, \delta +n\bar{y})\).
Nel modello Uniforme-Pareto \(\text{U}(0, \theta)\), la distribuzione a priori è \(\text{Pa}(\alpha, \varepsilon)\) e la distribuzione a posteriori è \(\text{Pa}(\alpha + n, \max(y_{(n)}, \varepsilon))\).
Riflessioni conclusive
In conclusione, l’utilizzo di priori coniugati presenta vantaggi e svantaggi. Cominciamo con i vantaggi principali. Il principale vantaggio dell’adozione di distribuzioni a priori coniugate risiede nella loro capacità di rendere l’analisi della distribuzione a posteriori trattabile da un punto di vista analitico. Ad esempio, nel corso di questo capitolo abbiamo esaminato come sia possibile formulare la distribuzione a posteriori in seguito a un esperimento composto da una serie di prove di Bernoulli (con una verosimiglianza binomiale), utilizzando una distribuzione Beta sia per la prior che per il posteriore.
Tuttavia, è cruciale riconoscere che i modelli basati sul concetto di famiglie coniugate presentano delle limitazioni intrinseche. Le distribuzioni coniugate a priori sono disponibili solamente per distribuzioni di verosimiglianza di base e relativamente semplici. Per modelli complessi e più realistici, la ricerca di priori coniugati diventa spesso un compito estremamente arduo, limitando quindi la loro utilità. Inoltre, anche quando le distribuzioni a priori coniugate sono disponibili, un modello che ne fa uso potrebbe non essere sufficientemente flessibile per adattarsi alle nostre credenze iniziali. Ad esempio, un modello basato su una distribuzione normale è sempre unimodale e simmetrico rispetto alla media \(\mu\). Tuttavia, se le nostre conoscenze iniziali non sono simmetriche o non seguono una distribuzione unimodale, la scelta di una distribuzione a priori normale potrebbe non risultare la più adeguata (Johnson et al., 2022).