here::here("code", "_common.R") |>
source()
# Load packages
if (!requireNamespace("pacman")) install.packages("pacman")
pacman::p_load(tidyr)45 Simulazioni
Prerequisiti
- Leggere Regression and Other Stories. Focalizzati sul capitolo 5.
- Leggere il capitolo Simulation (Schervish & DeGroot, 2014).
Concetti e Competenze Chiave
Preparazione del Notebook
45.1 Introduzione
La simulazione permette di sfruttare la potenza di calcolo dei moderni computer per sostituire i calcoli analitici, talvolta complessi o impossibili da risolvere. Attraverso la simulazione, non solo possiamo ottenere approssimazioni numeriche dei risultati attesi, ma possiamo anche studiare la distribuzione campionaria degli stimatori e delle funzioni complesse (come curve di regressione o istogrammi) quando le semplificazioni teoriche non sono più applicabili.
In questo capitolo, discuteremo alcuni esercizi di simulazione presentati da Schervish & DeGroot (2014) e da Gelman et al. (2021). Simulare variabili casuali è essenziale nelle statistiche applicate per diversi motivi:
Comprensione della variazione casuale: i modelli di probabilità imitano la variabilità del mondo reale. La simulazione aiuta a sviluppare intuizioni sulle oscillazioni casuali nel breve termine e sui loro effetti nel lungo termine.
Distribuzione campionaria: simulare dati consente di approssimare la distribuzione campionaria, trasferendo questa approssimazione alle stime e alle procedure statistiche.
Previsioni probabilistiche. i modelli di regressione producono previsioni probabilistiche. La simulazione è il metodo più generale per rappresentare l’incertezza nelle previsioni.
Inoltre, il processo di simulazione si basa sul seguente ragionamento fondamentale: un modello matematico è una narrazione di come i dati potrebbero essere stati generati. Simulare il modello significa seguirne i passaggi, compresi quelli casuali, per produrre dati sintetici (o surrogate data) che approssimino quelli reali. Questo approccio, noto come metodo Monte Carlo, sfrutta il Teorema dei Grandi Numeri per ottenere stime attendibili delle proprietà del modello.
45.2 Calcolare il valore medio di una distribuzione
Il teorema dei grandi numeri ci garantisce che, osservando un grande campione di variabili casuali i.i.d. (indipendenti e identicamente distribuite) con media finita, la media campionaria sarà vicina alla media della distribuzione. Utilizzando un computer per generare un ampio campione, possiamo calcolare la media delle variabili casuali al posto di affrontare calcoli analitici.
Per utilizzare la simulazione, occorre:
- Identificare il tipo di variabili casuali necessarie.
- Capire come farle generare al computer, sfruttando le funzioni built-in (ad esempio,
rnorm,runif,rbinom, ecc.). - Determinare il numero di osservazioni necessarie per avere fiducia nei risultati numerici.
Di seguito, vedremo un esempio pratico che verifica il concetto di simulazione confrontando i risultati numerici con quelli analitici.
45.3 Esempio 1: Calcolo della media di una distribuzione uniforme
La distribuzione uniforme sull’intervallo
sarà vicina alla media teorica
Vediamo come questo concetto si applica attraverso una simulazione in R.
# Impostiamo il numero di simulazioni
set.seed(123) # Per riproducibilità
sample_sizes <- c(100, 1000, 10000, 100000) # Dimensioni del campione
results <- data.frame(Sample_Size = sample_sizes, Sample_Mean = NA)
# Calcoliamo la media campionaria per ciascun campione
for (i in seq_along(sample_sizes)) {
n <- sample_sizes[i]
sample <- runif(n, min = 0, max = 1) # Generazione delle variabili uniformi
results$Sample_Mean[i] <- mean(sample) # Media campionaria
}
# Stampiamo i risultati
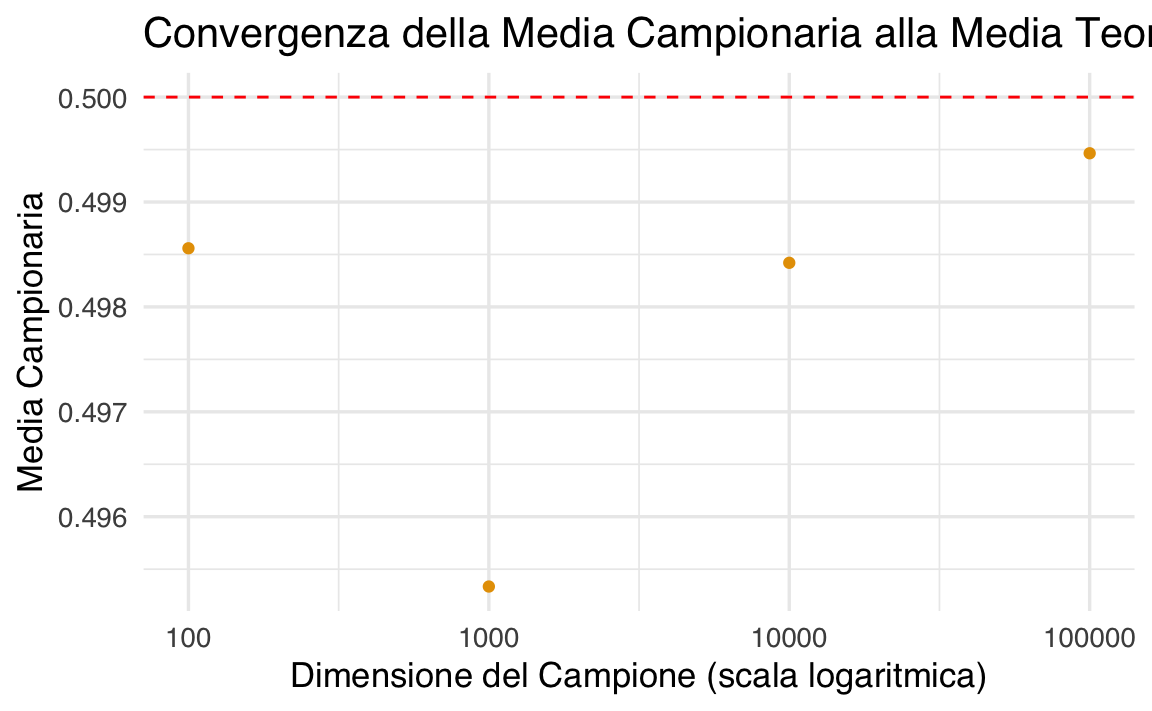
print(results)
#> Sample_Size Sample_Mean
#> 1 100 0.4986
#> 2 1000 0.4953
#> 3 10000 0.4984
#> 4 100000 0.4995# Visualizzazione grafica delle medie campionarie
ggplot(results, aes(x = Sample_Size, y = Sample_Mean)) +
geom_point() +
geom_hline(yintercept = 0.5, linetype = "dashed", color = "red") +
scale_x_log10() +
labs(
title = "Convergenza della Media Campionaria alla Media Teorica",
x = "Dimensione del Campione (scala logaritmica)",
y = "Media Campionaria"
) 
-
Generazione di variabili casuali: La funzione
runif(n, min = 0, max = 1)genera -
Media campionaria: Per ogni dimensione del campione (
mean(sample). -
Risultati: Presentiamo i risultati in una tabella e li visualizziamo graficamente, mostrando la convergenza della media campionaria (
L’output mostrerà una tabella con le medie campionarie per diverse dimensioni del campione. Il grafico evidenzierà come la media campionaria si avvicini al valore teorico di
45.4 Esempio 2: Probabilità di una Normale Standard
La probabilità che una variabile casuale standard normale sia almeno
Il teorema dei grandi numeri afferma che la proporzione:
dovrebbe essere vicina alla media teorica di
# Impostiamo il seme per la riproducibilità
set.seed(123)
# Dimensioni dei campioni da analizzare
sample_sizes <- c(100, 1000, 10000, 100000)
# Inizializziamo un data frame per salvare i risultati
results <- data.frame(
Sample_Size = sample_sizes,
Proportion = NA
)
# Calcolo delle proporzioni per ciascun campione
for (i in seq_along(sample_sizes)) {
n <- sample_sizes[i]
sample <- rnorm(n) # Generazione di variabili standard normali
Y <- ifelse(sample >= 1.0, 1, 0) # Variabili Bernoulli
results$Proportion[i] <- mean(Y) # Proporzione di Y_i = 1
}
# Stampiamo i risultati
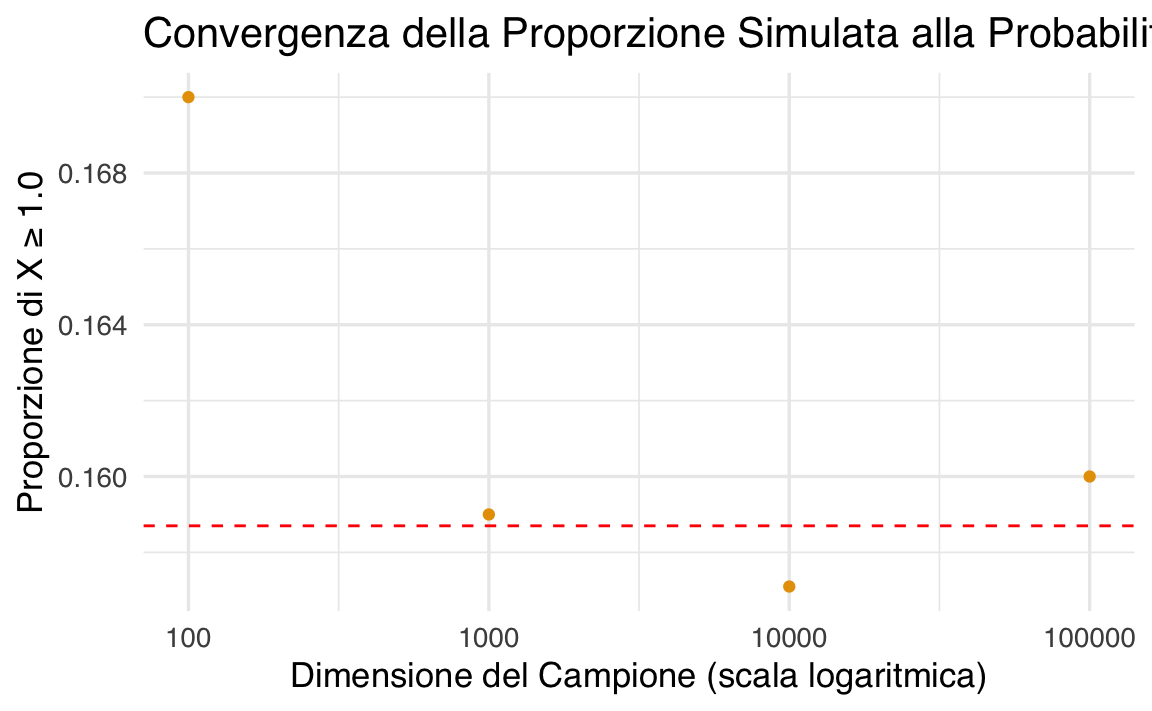
print(results)
#> Sample_Size Proportion
#> 1 100 0.1700
#> 2 1000 0.1590
#> 3 10000 0.1571
#> 4 100000 0.1600# Grafico della proporzione rispetto alla dimensione del campione
ggplot(results, aes(x = Sample_Size, y = Proportion)) +
geom_point() +
geom_hline(yintercept = 0.1587, linetype = "dashed", color = "red") +
scale_x_log10() +
labs(
title = "Convergenza della Proporzione Simulata alla Probabilità Teorica",
x = "Dimensione del Campione (scala logaritmica)",
y = "Proporzione di X ≥ 1.0"
) 
-
Generazione di variabili normali standard: La funzione
rnorm(n)genera -
Creazione delle variabili Bernoulli: Tramite
ifelse(sample >= 1.0, 1, 0)trasformiamo le variabili normali standard in variabili Bernoulli. -
Calcolo della proporzione: La proporzione di
-
Confronto visivo: Il grafico mostra la convergenza della proporzione simulata al valore teorico (
Il codice dimostra che, con l’aumentare della dimensione del campione
45.5 Esempio 3: Attesa per il Cambiamento dell’Umore
Immaginiamo un esperimento psicologico in cui due partecipanti completano un compito che induce stress, al termine del quale misurano il loro livello di umore su una scala da 1 a 10. Entrambi devono raggiungere un punteggio di umore pari a 7 prima di fermarsi. Si vuole stimare, in media, quanto tempo uno dei due dovrà attendere affinché l’altro raggiunga il punteggio stabilito.
Supponiamo che il cambiamento dell’umore per ciascun partecipante sia modellato come una variabile casuale gamma con parametri
Anziché affrontare calcoli analitici complessi, utilizzeremo una simulazione per stimare questa media.
# Impostiamo il seme per la riproducibilità
set.seed(123)
# Parametri della distribuzione gamma
k <- 10 # Numero di eventi
lambda <- 0.3 # Frequenza degli eventi
# Numero di simulazioni
n_sim <- 10000
# Simuliamo i tempi di completamento per A e B
X <- rgamma(n_sim, shape = k, rate = lambda)
Y <- rgamma(n_sim, shape = k, rate = lambda)
# Calcoliamo la differenza assoluta
Z <- abs(X - Y)
# Media della differenza assoluta
mean_Z <- mean(Z)
# Stampa dei risultati
cat("Media della differenza assoluta nei tempi di completamento:", mean_Z, "minuti\n")
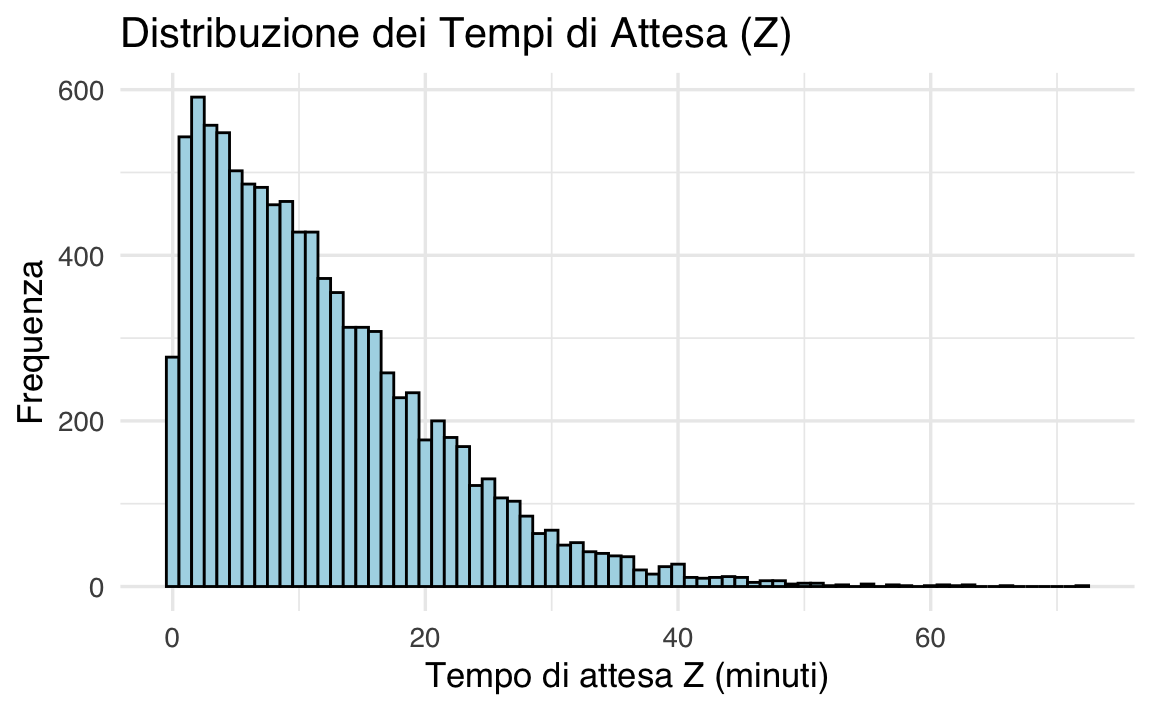
#> Media della differenza assoluta nei tempi di completamento: 11.76 minuti# Istogramma della distribuzione di Z
library(ggplot2)
ggplot(data.frame(Z), aes(x = Z)) +
geom_histogram(binwidth = 1, color = "black", fill = "lightblue") +
labs(
title = "Distribuzione dei Tempi di Attesa (Z)",
x = "Tempo di attesa Z (minuti)",
y = "Frequenza"
) 
-
Generazione dei tempi gamma: Usiamo la funzione
rgammaper generare -
Calcolo della differenza assoluta: La differenza assoluta
-
Stima della media: Utilizziamo la funzione
meanper calcolare la media di -
Istogramma: Il grafico visualizza la distribuzione dei tempi di attesa
Supponendo i parametri specificati, il codice produrrà una stima della media del tempo di attesa e un istogramma che mostra la distribuzione di
Questo esperimento simulato fornisce una stima del tempo medio di attesa tra due partecipanti in un contesto di regolazione emotiva. La variabilità nei tempi di completamento evidenzia come i processi individuali (ad esempio, il recupero emotivo) possano differire, un aspetto rilevante per la progettazione di interventi psicologici.
45.6 Esempio 4: Probabilità di Sequenze Lunghe in un Lancio di Monete
In questo esempio, esploriamo la probabilità di ottenere una sequenza di 12 teste consecutive in 100 lanci di una moneta. Anche se la probabilità di ottenere 12 teste in 12 lanci consecutivi è estremamente bassa, la situazione cambia quando consideriamo un numero maggiore di lanci, come 100. Attraverso una simulazione, possiamo osservare come la probabilità di tale evento aumenti significativamente in un contesto di 100 lanci.
Immagina di sentire qualcuno affermare di aver ottenuto 12 teste consecutive lanciando una moneta equa. La probabilità di ottenere esattamente 12 teste in 12 lanci indipendenti è data da
Per rispondere a questa domanda, utilizziamo una simulazione. Definiamo una variabile aleatoria
# Impostiamo il seme per la riproducibilità
set.seed(123)
# Parametri della simulazione
n_sim <- 10000 # Numero di simulazioni
n_flips <- 100 # Numero di lanci per simulazione
sequence_length <- 12 # Lunghezza della sequenza cercata
# Funzione per verificare la presenza di una sequenza di almeno 'sequence_length' teste
check_run <- function(flips, sequence_length) {
# Trasformiamo i risultati in una stringa per contare sequenze consecutive
flip_string <- paste(rbinom(flips, 1, 0.5), collapse = "")
return(any(grepl(paste(rep(1, sequence_length), collapse = ""), flip_string)))
}
# Eseguiamo la simulazione
results <- replicate(n_sim, check_run(n_flips, sequence_length))
# Calcoliamo la probabilità stimata
estimated_probability <- mean(results)
# Stampiamo il risultato
cat("Probabilità stimata di ottenere una sequenza di almeno", sequence_length,
"teste consecutive in", n_flips, "lanci:", estimated_probability, "\n")
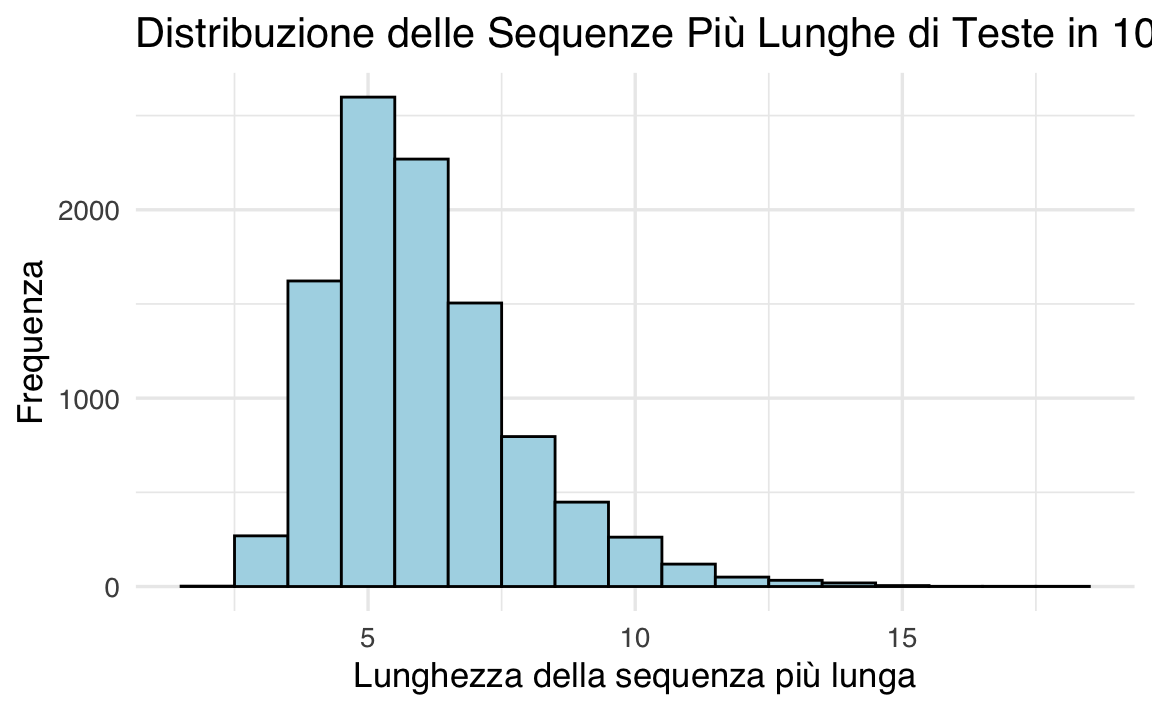
#> Probabilità stimata di ottenere una sequenza di almeno 12 teste consecutive in 100 lanci: 0.0106# Distribuzione delle lunghezze massime delle sequenze
longest_runs <- replicate(n_sim, {
flips <- rbinom(n_flips, 1, 0.5)
max(rle(flips)$lengths[which(rle(flips)$values == 1)])
})
# Grafico della distribuzione delle sequenze più lunghe
ggplot(data.frame(Longest_Run = longest_runs), aes(x = Longest_Run)) +
geom_histogram(binwidth = 1, color = "black", fill = "lightblue") +
labs(
title = "Distribuzione delle Sequenze Più Lunghe di Teste in 100 Lanci",
x = "Lunghezza della sequenza più lunga",
y = "Frequenza"
) 
-
Generazione dei lanci di moneta: Ogni lancio è modellato come una variabile binaria con
rbinom. -
Verifica della sequenza: La funzione
check_runcerca se una sequenza di lunghezza specificata appare nei lanci simulati. Questo è realizzato trasformando i risultati dei lanci in una stringa e utilizzando la funzionegreplper rilevare la sequenza. - Stima della probabilità: La proporzione di simulazioni in cui appare una sequenza di almeno 12 teste fornisce una stima della probabilità.
- Distribuzione delle sequenze più lunghe: Calcoliamo la lunghezza massima di una sequenza di teste per ogni simulazione e la visualizziamo con un istogramma.
L’output fornisce:
- La probabilità stimata di ottenere una sequenza di almeno 12 teste consecutive in 100 lanci.
- Un grafico che mostra la distribuzione delle sequenze più lunghe osservate nei 10,000 esperimenti.
Questo approccio può essere utilizzato per modellare eventi rari ma possibili in psicologia, come la comparsa di lunghe sequenze di comportamenti simili in compiti ripetuti (ad esempio, risposte corrette consecutive in un test di memoria). La simulazione offre una stima della probabilità di osservare tali eventi, fornendo informazioni utili per interpretare i risultati sperimentali.
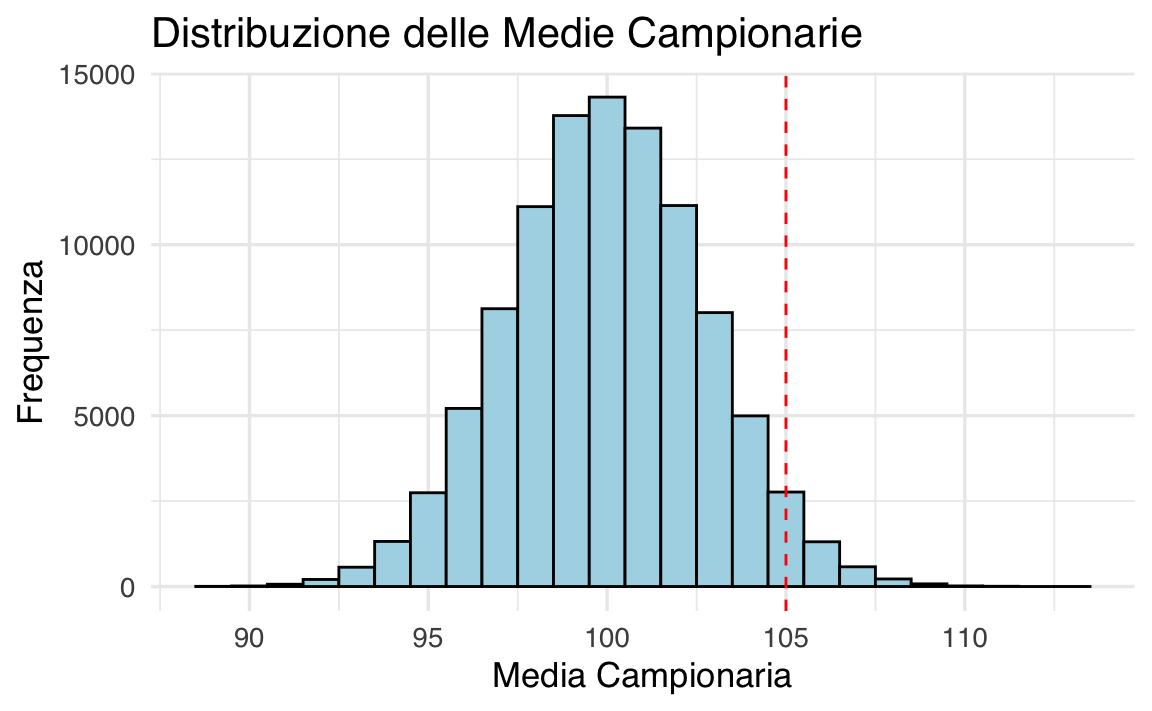
45.7 Esempio: Probabilità della Media Campionaria
Consideriamo un campione casuale di 30 persone estratte dalla popolazione generale, in cui il QI segue una distribuzione normale
La distribuzione della media campionaria è:
e la probabilità di superare 105 si calcola come:
# Calcolo analitico della probabilità
mu <- 100 # Media della popolazione
sigma <- 15 # Deviazione standard della popolazione
n <- 30 # Dimensione del campione
threshold <- 105 # Soglia per la media campionaria
# Probabilità teorica
prob_teorica <- 1 - pnorm(threshold, mean = mu, sd = sigma / sqrt(n))
cat("Probabilità teorica che la media campionaria sia maggiore di 105:", prob_teorica, "\n")
#> Probabilità teorica che la media campionaria sia maggiore di 105: 0.03394Procediamo ora ad una simulazione:
# Simulazione
set.seed(123) # Per riproducibilità
n_sim <- 100000 # Numero di simulazioni
# Generazione dei campioni e calcolo delle medie campionarie
sample_means <- replicate(n_sim, mean(rnorm(n, mean = mu, sd = sigma)))
# Probabilità stimata tramite simulazione
prob_simulata <- mean(sample_means > threshold)
cat("Probabilità stimata tramite simulazione:", prob_simulata, "\n")
#> Probabilità stimata tramite simulazione: 0.0339# Istogramma delle medie campionarie
ggplot(data.frame(Sample_Means = sample_means), aes(x = Sample_Means)) +
geom_histogram(binwidth = 1, color = "black", fill = "lightblue") +
geom_vline(xintercept = threshold, linetype = "dashed", color = "red") +
labs(
title = "Distribuzione delle Medie Campionarie",
x = "Media Campionaria",
y = "Frequenza"
) 
- La funzione
pnormcalcola la probabilità cumulativa della normale. Sottraendo tale valore da 1, otteniamo la probabilità di superare la soglia specificata. - La funzione
rnormgenerareplicate, calcolando la media campionaria ogni volta. - La proporzione di medie campionarie che superano 105 fornisce la stima simulata della probabilità.
La simulazione offre un metodo pratico per stimare probabilità anche in situazioni più complesse, dove il calcolo analitico potrebbe non essere semplice.
45.8 Esempio 5: Simulazione in un Trial Clinico
In questo esempio, vogliamo analizzare i risultati di un trial clinico con quattro gruppi di trattamento (
L’analisi si basa su una distribuzione a posteriori di
-
La verosimiglianza (likelihood): Derivata dai dati osservati
-
La distribuzione a priori: Una distribuzione Beta(
45.8.1 Derivazione della Distribuzione a Posteriori
Likelihood (Verosimiglianza):
La probabilità di osservareDistribuzione a priori:
Assumiamo cheDistribuzione a posteriori:
Combinando la verosimiglianza e la distribuzione a priori, otteniamo la distribuzione a posteriori (tramite il Teorema di Bayes):
45.8.2 Simulazione con R
Il nostro obiettivo è stimare:
-
- La probabilità che un trattamento sia il più efficace (
- La probabilità che tutti i
# Parametri della distribuzione a priori
alpha0 <- 2 # Parametro a priori per i successi
beta0 <- 2 # Parametro a priori per i fallimenti
# Dati osservati
n <- c(50, 50, 50, 50) # Numero totale di pazienti per ciascun gruppo
x <- c(35, 30, 25, 20) # Numero di pazienti che non hanno avuto una ricaduta
# Parametri posteriori
posterior_alpha <- alpha0 + x
posterior_beta <- beta0 + n - x
# Numero di simulazioni
n_sim <- 10000
# Simulazione delle distribuzioni posteriori
set.seed(123) # Per riproducibilità
P <- sapply(1:4, function(i) rbeta(n_sim, posterior_alpha[i], posterior_beta[i]))
# 1. Probabilità che ciascun trattamento sia migliore del placebo
probs_better_than_placebo <- colMeans(P[, 1:3] > P[, 4])
names(probs_better_than_placebo) <- paste0("P", 1:3, " > P4")
# print(probs_better_than_placebo)
# 2. Probabilità che un trattamento sia il migliore
probs_best <- colMeans(apply(P, 1, function(row) row == max(row)))
names(probs_best) <- paste0("P", 1:4, " è il migliore")
# print(probs_best)
# 3. Probabilità che tutti i Pi siano entro epsilon
epsilon <- 0.1
probs_within_epsilon <- mean(apply(P, 1, function(row) max(row) - min(row) <= epsilon))
cat("Probabilità che tutti i Pi siano entro epsilon =", epsilon, ":", probs_within_epsilon, "\n")
#> Probabilità che tutti i Pi siano entro epsilon = 0.1 : 0.0047
# Visualizzazione: Distribuzioni posteriori
P_df <- data.frame(
Simulazione = rep(1:n_sim, 4),
P = as.vector(P),
Gruppo = factor(rep(1:4, each = n_sim), labels = paste0("P", 1:4))
)
ggplot(P_df, aes(x = P, fill = Gruppo)) +
geom_density(alpha = 0.6) +
labs(
title = "Distribuzioni Posteriori dei Pi",
x = "Probabilità Pi",
y = "Densità"
) 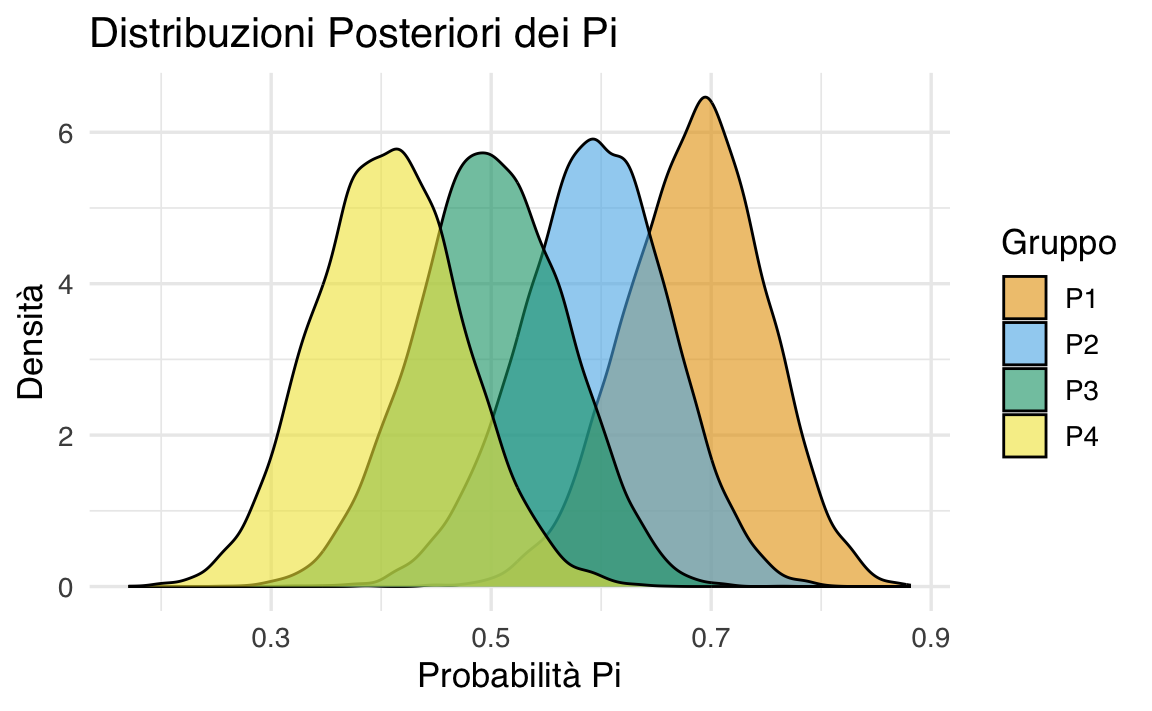
45.8.3 Risultati e Interpretazione
-
Probabilità
- Probabilità che un trattamento sia il migliore: Mostrano quale trattamento è più efficace in termini probabilistici.
-
Probabilità di vicinanza (
Questo approccio combina inferenza bayesiana e simulazione per rispondere a domande chiave in un trial clinico.
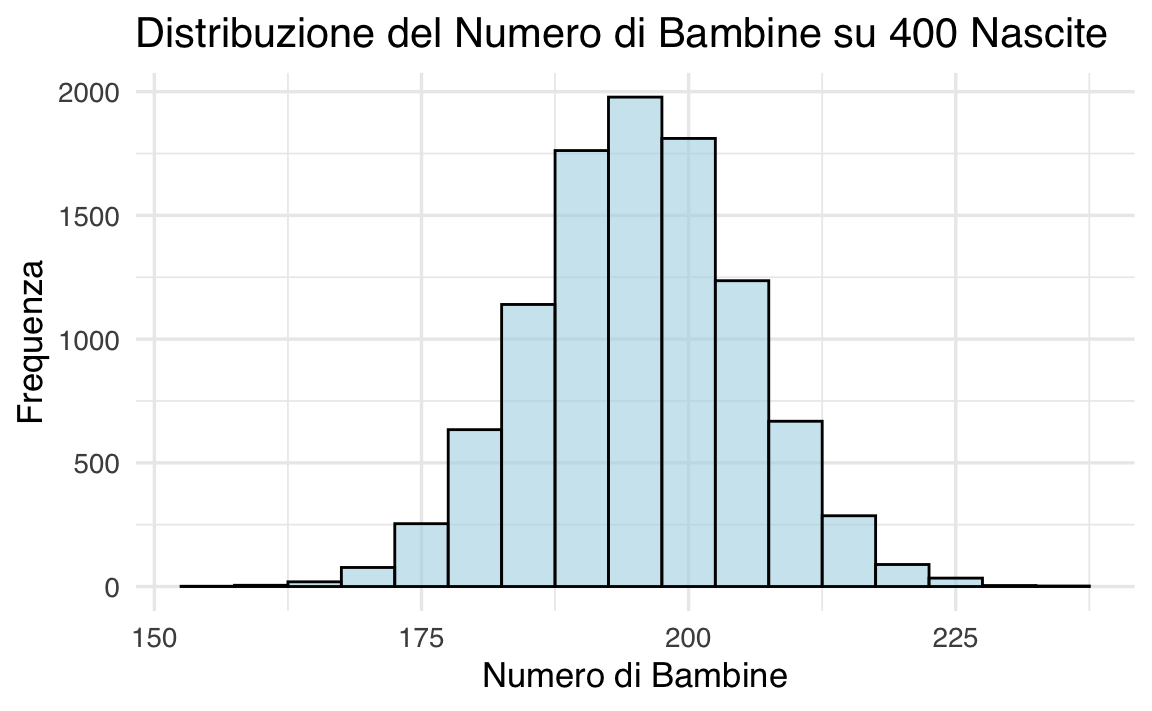
45.9 Esempio 6: Quante bambine su 400 nascite?
Supponiamo che la probabilità di nascita di una bambina sia
# Numero di simulazioni
n_sims <- 10000
# Probabilità di nascita di una bambina
p_girl <- 0.488
# Simulazione del numero di bambine
set.seed(123)
n_girls <- rbinom(n_sims, size = 400, prob = p_girl)
# Visualizzazione dell'istogramma
ggplot(data.frame(n_girls), aes(x = n_girls)) +
geom_histogram(binwidth = 5, fill = "lightblue", color = "black", alpha = 0.6) +
labs(
title = "Distribuzione del Numero di Bambine su 400 Nascite",
x = "Numero di Bambine",
y = "Frequenza"
)
45.10 Simulazione di probabilità continue
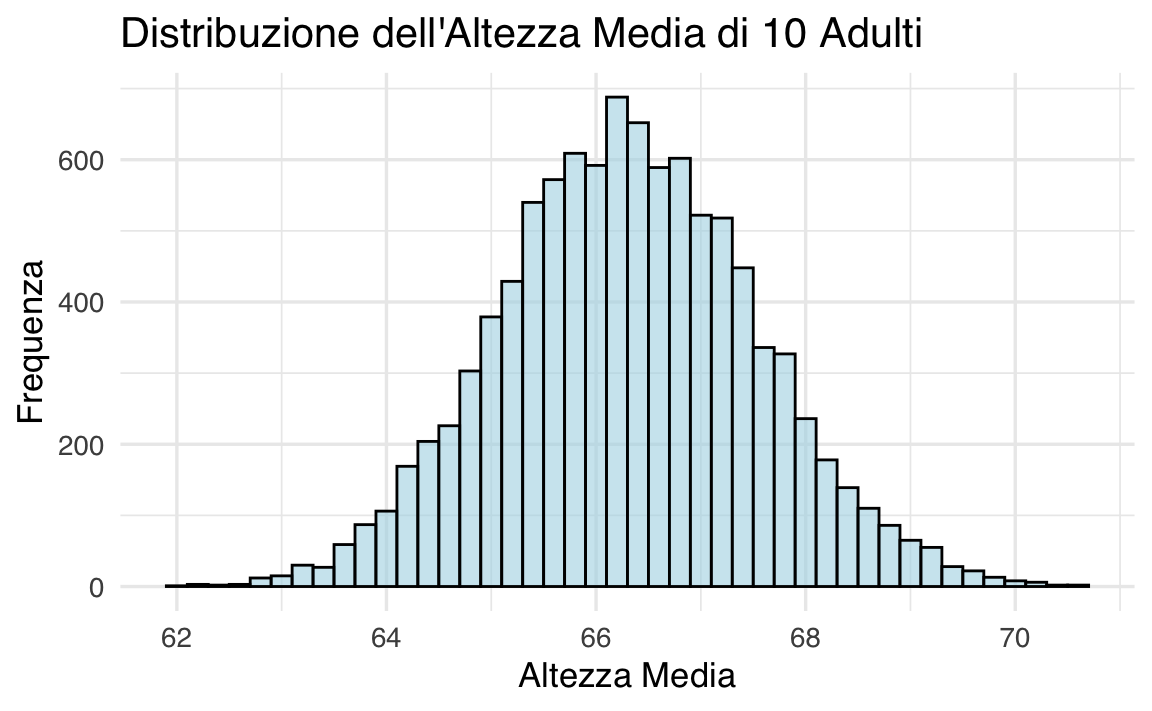
I metodi di simulazione non si limitano a variabili discrete: come dimostrato da Gelman et al. (2021), è possibile integrare anche distribuzioni continue. Ad esempio, per generare l’altezza di un adulto in base al genere – con il 52% di donne e il 48% di uomini – si utilizzano due distribuzioni normali distinte. L’altezza degli uomini segue approssimativamente una distribuzione normale con una media di 69.1 pollici e una deviazione standard di 2.9 pollici; per le donne, la media è 63.7 pollici e la deviazione standard è 2.7 pollici. Ecco il codice per generare l’altezza di un adulto scelto casualmente:
Supponiamo di selezionare 10 adulti a caso. Cosa possiamo dire della loro altezza media?
set.seed(123)
n_sims <- 10000
avg_heights <- replicate(n_sims, simulate_height(10))
# Visualizzazione dell'istogramma
ggplot(data.frame(avg_heights), aes(x = avg_heights)) +
geom_histogram(binwidth = 0.2, fill = "lightblue", color = "black", alpha = 0.6) +
labs(
title = "Distribuzione dell'Altezza Media di 10 Adulti",
x = "Altezza Media",
y = "Frequenza"
)
45.11 Sommario di una simulazione con media e mediana
Quando le nostre distribuzioni sono costruite come simulazioni al computer, può essere conveniente riassumerle in qualche modo. Tipicamente, riassumiamo la posizione di una distribuzione con la sua media o mediana.
La variazione nella distribuzione è tipicamente riassunta dalla deviazione standard, ma spesso preferiamo usare la deviazione mediana assoluta. Se la mediana di un insieme di simulazioni
Tuttavia, poiché siamo abituati a lavorare con le deviazioni standard, quando calcoliamo la deviazione mediana assoluta, la riscaliamo moltiplicandola per 1.483, il che riproduce la deviazione standard nel caso speciale della distribuzione normale:
Preferiamo tipicamente i riassunti basati sulla mediana perché sono più stabili computazionalmente, e riscaliamo il riassunto basato sulla mediana della variazione come descritto sopra in modo da essere comparabile alla deviazione standard, che sappiamo già interpretare nella pratica statistica usuale.
Ecco come implementare quanto sopra in R per i dati relativi all’altezza media di 10 adulti.
# Calcolo della media e mediana
mean_avg_height <- mean(avg_heights)
median_avg_height <- median(avg_heights)
# Calcolo della deviazione standard
sd_avg_height <- sd(avg_heights)
# Calcolo della MAD (Deviazione Mediana Assoluta)
mad_avg_height <- median(abs(avg_heights - median_avg_height)) * 1.483
# Risultati
cat("Mean:", mean_avg_height, "\n")
#> Mean: 66.3
cat("Median:", median_avg_height, "\n")
#> Median: 66.28
cat("Standard Deviation:", sd_avg_height, "\n")
#> Standard Deviation: 1.226
cat("MAD (scaled):", mad_avg_height, "\n")
#> MAD (scaled): 1.23545.11.1 Intervalli di Incertezza
Per rappresentare l’incertezza, si calcolano intervalli centrali al 50% e al 95%.
# Intervalli di incertezza
lower_50 <- quantile(avg_heights, 0.25)
upper_50 <- quantile(avg_heights, 0.75)
lower_95 <- quantile(avg_heights, 0.025)
upper_95 <- quantile(avg_heights, 0.975)
cat("50% Interval:", lower_50, "-", upper_50, "\n")
#> 50% Interval: 65.46 - 67.13
cat("95% Interval:", lower_95, "-", upper_95, "\n")
#> 95% Interval: 63.91 - 68.77Ecco come interpretarli.
Intervallo centrale al 50%.
- Contiene i valori centrali che coprono il 50% della distribuzione. Questo intervallo si estende dal primo quartile (25° percentile) al terzo quartile (75° percentile).
- Indica la fascia di valori in cui si trovano i risultati “più comuni” o tipici. È una misura di variabilità concentrata nella parte centrale della distribuzione, meno sensibile a valori estremi.
- Per le altezze medie simulate di 10 adulti, un intervallo centrale al 50% che va da 65 a 67 pollici indica che metà delle medie osservate si trova in questo intervallo.
Intervallo centrale al 95%.
- Contiene il 95% della distribuzione simulata, lasciando solo il 2.5% dei valori al di sotto e il 2.5% al di sopra dell’intervallo. Questo intervallo si calcola tra il 2.5° percentile e il 97.5° percentile.
- Indica una fascia più ampia che cattura quasi tutti i valori plausibili, inclusi quelli meno probabili ma comunque possibili.
- Per le altezze medie simulate, un intervallo al 95% che va da 64 a 68 pollici significa che, in quasi tutte le simulazioni, l’altezza media si trova in questo intervallo, con poche eccezioni.
45.12 Commenti e Considerazioni Finali
La simulazione di dati fittizi, al di là del semplice calcolo numerico, ci permette di comprendere in profondità come le proprietà dei metodi statistici si manifestino in scenari pratici. Una delle ragioni principali per cui si ricorre alla simulazione è la difficoltà – o l’impossibilità – di derivare in forma chiusa le distribuzioni campionarie per stimatori complessi. In questi casi, simulare il modello significa generare in successione variabili casuali e utilizzare il metodo Monte Carlo per approssimare aspettative, varianze e perfino l’intera distribuzione di funzioni complesse.
Simulare significa, in sostanza, seguire la “storia” di come i dati potrebbero essere stati generati, replicando le fasi di un modello stocastico – dalla generazione delle variabili “esogenee” (quelle indipendenti) a quelle che dipendono dalle precedenti. Questo approccio è fondamentale non solo per ottenere stime, ma anche per controllare e validare i modelli: se i dati simulati non assomigliano a quelli reali, il modello probabilmente necessita di revisione.
Inoltre, la simulazione offre un potente strumento per eseguire analisi di sensibilità: possiamo variare le assunzioni del modello e osservare come cambiano le inferenze, aiutandoci a valutare la robustezza delle conclusioni tratte.
Infine, la capacità di simulare distribuzioni complesse e approssimare le distribuzioni campionarie di stimatori o funzioni (come curve di regressione o istogrammi) rappresenta uno strumento essenziale soprattutto quando si lavora con dati reali che violano le semplificazioni delle teorie classiche.
Per questo motivo, molti autori raccomandano di eseguire simulazioni preliminari prima di raccogliere i dati, per valutare aspetti come la dimensione campionaria necessaria o il potere statistico, e successivamente utilizzarle per il controllo e la validazione del modello.
Come sintetizzato da Gelman & Brown (2024), le simulazioni non sono solo un supporto numerico, ma un vero e proprio strumento per comprendere e interpretare la complessità dei processi di generazione dei dati, elemento chiave in ambito psicologico e in numerose altre discipline.
45.13 Esercizi
Immagina il caso di 220 studenti che devono sostenere tre prove in itinere in un corso. Il voto finale è la media dei voti ottenuti in queste tre prove. Le distribuzioni dei voti per le prove sono descritte come segue:
- Prima prova: I voti sono distribuiti secondo una gaussiana con media 24. Il 15% degli studenti ottiene un voto inferiore a 18.
- Seconda prova: I voti sono distribuiti secondo una gaussiana con media 25. Il 10% degli studenti ottiene un voto inferiore a 18.
- Terza prova: I voti sono distribuiti secondo una gaussiana con media 26. Solo il 5% degli studenti ottiene un voto inferiore a 18.
Dei 220 studenti iniziali:
- Il 10% non partecipa alla prima prova.
- Un ulteriore 5% non partecipa alla seconda prova.
Per ottenere il voto finale, uno studente deve partecipare a tutte e tre le prove.
Utilizzando una simulazione, trova la media finale dei voti e calcola l’intervallo di incertezza al 90% per la stima della media.
Il codice R segente esegue una simulazione completa del problema ed è strutturato nei seguenti passi:
- Calcola le deviazioni standard per le tre prove usando la funzione qnorm
- Determina il numero di studenti che partecipano a tutte le prove
- Simula i voti per ogni prova per ogni studente secondo le distribuzioni normali specificate
- Calcola la media finale per ogni studente
- Ripete la simulazione 1000 volte per ottenere un intervallo di confidenza robusto
- Calcola e visualizza i risultati con un istogramma
Il codice calcola automaticamente:
- Il numero finale di studenti che partecipano a tutte e tre le prove
- La media dei voti finali
- L’intervallo di confidenza al 90% per questa media
# Impostazione del seme per la riproducibilità
set.seed(123)
# Numero iniziale di studenti
initial_students <- 220
# Calcolo delle deviazioni standard per le tre prove
sd1 <- (18 - 24) / qnorm(0.15)
sd2 <- (18 - 25) / qnorm(0.10)
sd3 <- (18 - 26) / qnorm(0.05)
# Parametri delle distribuzioni
mean1 <- 24
mean2 <- 25
mean3 <- 26
# Calcolo del numero di studenti che partecipano a tutte le prove
students_after_first <- initial_students * 0.9 # 10% non partecipa alla prima prova
students_after_second <- students_after_first * 0.95 # 5% non partecipa alla seconda prova
final_students <- round(students_after_second) # Numero di studenti che partecipano a tutte le prove
# Generiamo i voti per ogni prova per ogni studente
set.seed(123) # Per la riproducibilità
# Simulazione dei voti per la prima prova
grades_first <- rnorm(final_students, mean = mean1, sd = sd1)
# Simulazione dei voti per la seconda prova
grades_second <- rnorm(final_students, mean = mean2, sd = sd2)
# Simulazione dei voti per la terza prova
grades_third <- rnorm(final_students, mean = mean3, sd = sd3)
# Calcolo della media finale per ogni studente
final_grades <- (grades_first + grades_second + grades_third) / 3
# Calcolo della media complessiva
mean_final <- mean(final_grades)
# Ripetiamo la simulazione più volte per ottenere un intervallo di confidenza
n_simulations <- 1000
means <- numeric(n_simulations)
for (i in 1:n_simulations) {
# Simulazione dei voti per tutte e tre le prove
sim_grades_first <- rnorm(final_students, mean = mean1, sd = sd1)
sim_grades_second <- rnorm(final_students, mean = mean2, sd = sd2)
sim_grades_third <- rnorm(final_students, mean = mean3, sd = sd3)
# Calcolo della media finale per questa simulazione
sim_final_grades <- (sim_grades_first + sim_grades_second + sim_grades_third) / 3
means[i] <- mean(sim_final_grades)
}
# Calcolo dell'intervallo di confidenza al 90%
confidence_interval <- quantile(means, c(0.05, 0.95))
# Risultati
print(paste("Numero di studenti che partecipano a tutte le prove:", final_students))
#> [1] "Numero di studenti che partecipano a tutte le prove: 188"
print(paste("Deviazione standard per la prima prova:", round(sd1, 4)))
#> [1] "Deviazione standard per la prima prova: 5.7891"
print(paste("Deviazione standard per la seconda prova:", round(sd2, 4)))
#> [1] "Deviazione standard per la seconda prova: 5.4621"
print(paste("Deviazione standard per la terza prova:", round(sd3, 4)))
#> [1] "Deviazione standard per la terza prova: 4.8637"
print(paste("Media finale dei voti:", round(mean_final, 4)))
#> [1] "Media finale dei voti: 25.1772"
print(paste("Intervallo di confidenza al 90%:",
round(confidence_interval[1], 4), "-",
round(confidence_interval[2], 4)))
#> [1] "Intervallo di confidenza al 90%: 24.6456 - 25.3693"
# Visualizzazione dei risultati
hist(means, main = "Distribuzione delle medie finali",
xlab = "Media finale", col = "lightblue", border = "white")
abline(v = confidence_interval, col = "red", lwd = 2, lty = 2)
abline(v = mean_final, col = "blue", lwd = 2)
legend("topright", legend = c("Media stimata", "Intervallo di confidenza 90%"),
col = c("blue", "red"), lwd = 2, lty = c(1, 2))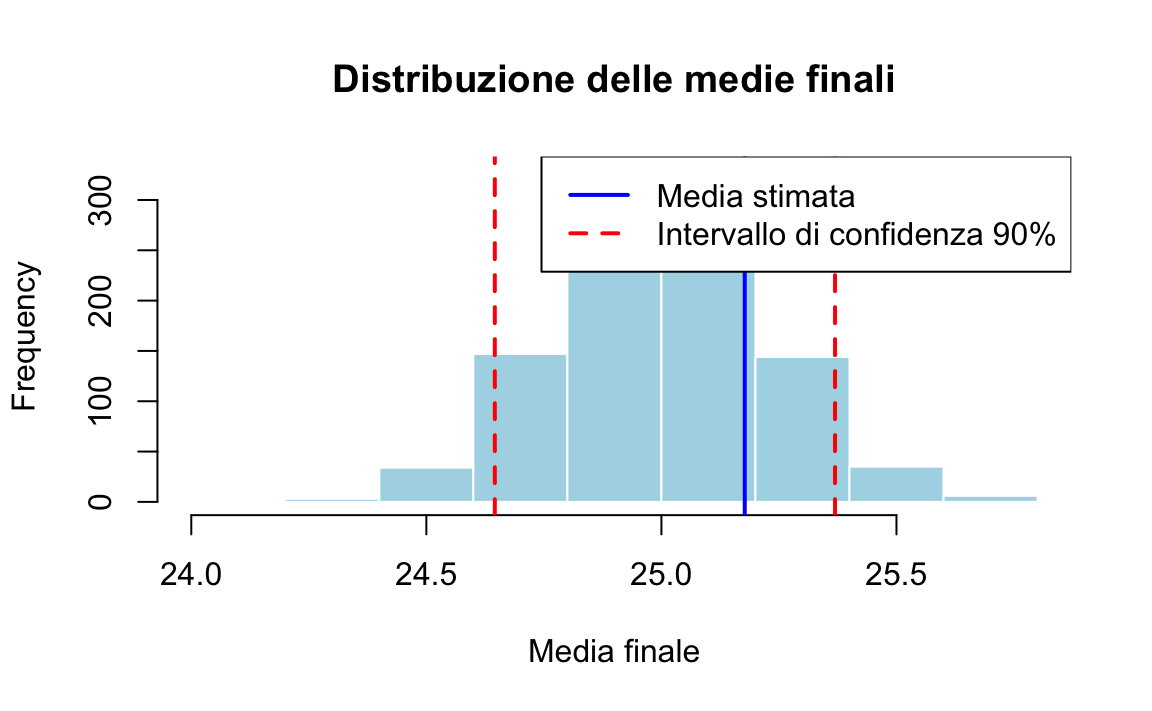
Informazioni sull’Ambiente di Sviluppo
sessionInfo()
#> R version 4.4.2 (2024-10-31)
#> Platform: aarch64-apple-darwin20
#> Running under: macOS Sequoia 15.3.2
#>
#> Matrix products: default
#> BLAS: /Library/Frameworks/R.framework/Versions/4.4-arm64/Resources/lib/libRblas.0.dylib
#> LAPACK: /Library/Frameworks/R.framework/Versions/4.4-arm64/Resources/lib/libRlapack.dylib; LAPACK version 3.12.0
#>
#> locale:
#> [1] C/UTF-8/C/C/C/C
#>
#> time zone: Europe/Rome
#> tzcode source: internal
#>
#> attached base packages:
#> [1] stats graphics grDevices utils datasets methods base
#>
#> other attached packages:
#> [1] thematic_0.1.6 MetBrewer_0.2.0 ggokabeito_0.1.0 see_0.11.0
#> [5] gridExtra_2.3 patchwork_1.3.0 bayesplot_1.11.1 psych_2.5.3
#> [9] scales_1.3.0 markdown_2.0 knitr_1.50 lubridate_1.9.4
#> [13] forcats_1.0.0 stringr_1.5.1 dplyr_1.1.4 purrr_1.0.4
#> [17] readr_2.1.5 tidyr_1.3.1 tibble_3.2.1 ggplot2_3.5.1
#> [21] tidyverse_2.0.0 rio_1.2.3 here_1.0.1
#>
#> loaded via a namespace (and not attached):
#> [1] generics_0.1.3 stringi_1.8.4 lattice_0.22-6 hms_1.1.3
#> [5] digest_0.6.37 magrittr_2.0.3 evaluate_1.0.3 grid_4.4.2
#> [9] timechange_0.3.0 fastmap_1.2.0 rprojroot_2.0.4 jsonlite_1.9.1
#> [13] mnormt_2.1.1 cli_3.6.4 rlang_1.1.5 munsell_0.5.1
#> [17] withr_3.0.2 tools_4.4.2 parallel_4.4.2 tzdb_0.5.0
#> [21] colorspace_2.1-1 pacman_0.5.1 vctrs_0.6.5 R6_2.6.1
#> [25] lifecycle_1.0.4 htmlwidgets_1.6.4 pkgconfig_2.0.3 pillar_1.10.1
#> [29] gtable_0.3.6 glue_1.8.0 xfun_0.51 tidyselect_1.2.1
#> [33] rstudioapi_0.17.1 farver_2.1.2 htmltools_0.5.8.1 nlme_3.1-167
#> [37] labeling_0.4.3 rmarkdown_2.29 compiler_4.4.2