here::here("code", "_common.R") |>
source()
# Load packages
if (!requireNamespace("pacman")) install.packages("pacman")
pacman::p_load(reshape2)50 Calcolo della distribuzione a posteriori gaussiana tramite metodo a griglia
- utilizzare il metodo basato su griglia per approssimare la distribuzione a posteriori gaussiana.
- Leggere il capitolo Grid approximation del testo di Johnson et al. (2022).
50.1 Introduzione
In questo capitolo, estenderemo la discussione precedente sul calcolo della distribuzione a posteriori utilizzando il metodo basato su griglia, applicandolo questa volta a un caso con verosimiglianza gaussiana. In particolare, ci concentreremo su come costruire un modello gaussiano per descrivere l’intelligenza.
Immaginiamo di condurre uno studio sulla plusdotazione, considerando l’approccio psicometrico. Secondo questo approccio, una persona è considerata plusdotata se ha un QI (Quoziente Intellettivo) di 130 o superiore (Robinson, Zigler, & Gallagher, 2000). Anche se l’uso di un QI di 130 come soglia è il criterio più comune, non è universalmente accettato. L’intelligenza nei bambini plusdotati non è solo superiore rispetto a quella dei loro pari, ma è qualitativamente diversa (Lubart & Zenasni, 2010). I bambini plusdotati tendono a mostrare caratteristiche come un vocabolario ampio, un linguaggio molto sviluppato, processi di ragionamento avanzati, eccellente memoria, vasti interessi, forte curiosità, empatia, capacità di leadership, abilità visive elevate, impegno in situazioni sfidanti e un forte senso di giustizia (Song & Porath, 2005).
Nella simulazione che seguirà, assumeremo che i dati provengano da una distribuzione normale. Per semplicità, considereremo che la deviazione standard sia nota e pari a 5. Il parametro della media sarà l’oggetto della nostra inferenza.
50.2 Dati
Supponiamo di avere un campione di 10 osservazioni. I dati saranno generati casualmente da una distribuzione normale con media 130 e deviazione standard 5.
set.seed(123) # Per la riproducibilità
vera_media <- 130 # Media vera
sigma_conosciuta <- 5 # Deviazione standard conosciuta
dimensione_campione <- 10 # Dimensione del campione
# Generare un campione
campione <- round(rnorm(n = dimensione_campione, mean = vera_media, sd = sigma_conosciuta))
campione
#> [1] 127 129 138 130 131 139 132 124 127 12850.3 Griglia
Creiamo ora una griglia di 100 valori compresi tra 110 e 150.
mu_griglia <- seq(110, 150, length.out = 100)
mu_griglia
#> [1] 110.0 110.4 110.8 111.2 111.6 112.0 112.4 112.8 113.2 113.6 114.0
#> [12] 114.4 114.8 115.3 115.7 116.1 116.5 116.9 117.3 117.7 118.1 118.5
#> [23] 118.9 119.3 119.7 120.1 120.5 120.9 121.3 121.7 122.1 122.5 122.9
#> [34] 123.3 123.7 124.1 124.5 124.9 125.4 125.8 126.2 126.6 127.0 127.4
#> [45] 127.8 128.2 128.6 129.0 129.4 129.8 130.2 130.6 131.0 131.4 131.8
#> [56] 132.2 132.6 133.0 133.4 133.8 134.2 134.6 135.1 135.5 135.9 136.3
#> [67] 136.7 137.1 137.5 137.9 138.3 138.7 139.1 139.5 139.9 140.3 140.7
#> [78] 141.1 141.5 141.9 142.3 142.7 143.1 143.5 143.9 144.3 144.7 145.2
#> [89] 145.6 146.0 146.4 146.8 147.2 147.6 148.0 148.4 148.8 149.2 149.6
#> [100] 150.050.4 Calcolo della Verosimiglianza
Per ogni valore della griglia, calcoliamo la verosimiglianza complessiva come prodotto delle densità di probabilità.
likelihood <- sapply(mu_griglia, function(mu) {
prod(dnorm(campione, mean = mu, sd = sigma_conosciuta))
})
likelihood
#> [1] 5.288e-50 1.406e-48 3.502e-47 8.172e-46 1.786e-44 3.658e-43 7.016e-42
#> [8] 1.261e-40 2.122e-39 3.347e-38 4.944e-37 6.842e-36 8.870e-35 1.077e-33
#> [15] 1.226e-32 1.306e-31 1.304e-30 1.220e-29 1.069e-28 8.772e-28 6.745e-27
#> [22] 4.858e-26 3.278e-25 2.072e-24 1.227e-23 6.807e-23 3.537e-22 1.722e-21
#> [29] 7.852e-21 3.355e-20 1.343e-19 5.033e-19 1.768e-18 5.816e-18 1.792e-17
#> [36] 5.175e-17 1.400e-16 3.547e-16 8.418e-16 1.872e-15 3.899e-15 7.608e-15
#> [43] 1.391e-14 2.381e-14 3.820e-14 5.741e-14 8.082e-14 1.066e-13 1.317e-13
#> [50] 1.524e-13 1.652e-13 1.678e-13 1.596e-13 1.423e-13 1.188e-13 9.293e-14
#> [57] 6.809e-14 4.674e-14 3.005e-14 1.810e-14 1.022e-14 5.400e-15 2.674e-15
#> [64] 1.240e-15 5.391e-16 2.195e-16 8.370e-17 2.990e-17 1.001e-17 3.138e-18
#> [71] 9.215e-19 2.535e-19 6.535e-20 1.578e-20 3.569e-21 7.563e-22 1.501e-22
#> [78] 2.791e-23 4.863e-24 7.935e-25 1.213e-25 1.737e-26 2.330e-27 2.929e-28
#> [85] 3.448e-29 3.802e-30 3.929e-31 3.802e-32 3.447e-33 2.928e-34 2.330e-35
#> [92] 1.736e-36 1.212e-37 7.931e-39 4.860e-40 2.790e-41 1.500e-42 7.557e-44
#> [99] 3.566e-45 1.576e-4650.5 Calcolo della Distribuzione a Posteriori
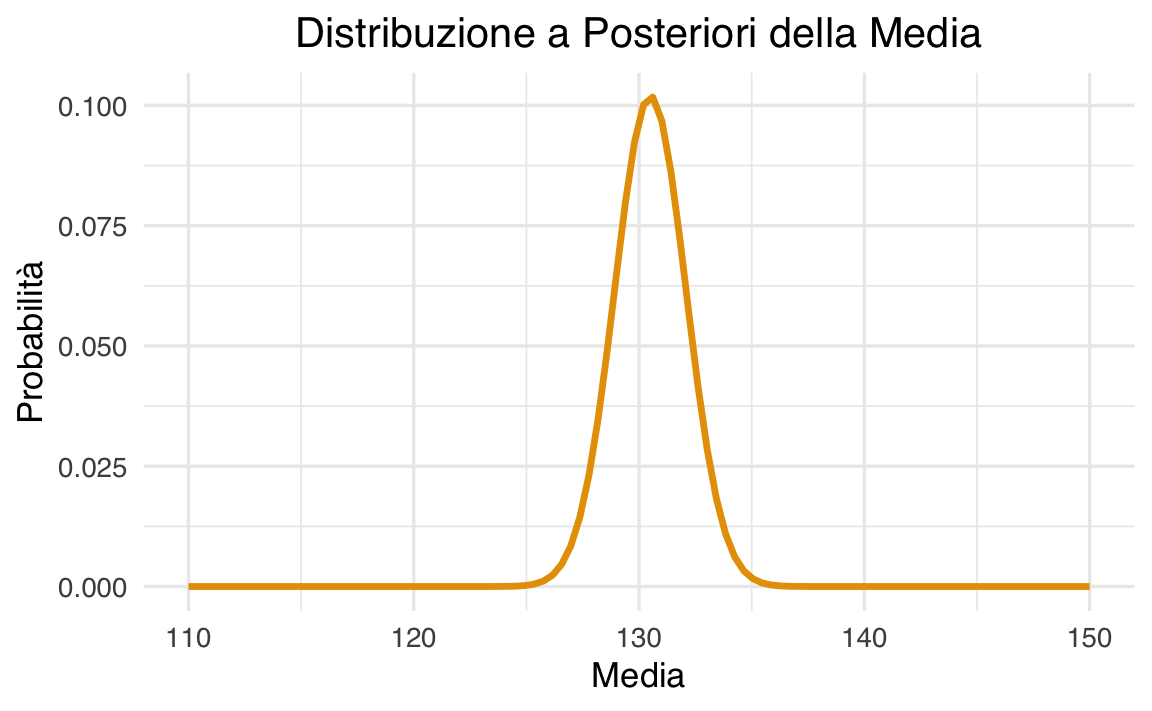
Impostiamo una prior uniforme e calcoliamo la distribuzione a posteriori normalizzata.
Visualizzazione:
# Creazione del dataframe per il plot
dat <- tibble(
mu_griglia = mu_griglia, # Ascissa
posterior = posterior # Ordinata
)
# Grafico con ggplot2
dat |>
ggplot(aes(x = mu_griglia, y = posterior)) +
geom_line(size = 1.2) +
labs(
title = "Distribuzione a Posteriori della Media",
x = "Media",
y = "Probabilità"
) +
theme(
plot.title = element_text(hjust = 0.5), # Centra il titolo
legend.position = "none"
)
50.5.1 Aggiunta di una Prior Informativa
Usiamo una prior gaussiana con media 140 e deviazione standard 3.
# Calcolo prior, posterior non normalizzato e posterior
prior <- dnorm(mu_griglia, mean = 140, sd = 3)
posterior_non_norm <- likelihood * prior
posterior <- posterior_non_norm / sum(posterior_non_norm)
# Creazione del dataframe per il grafico
dat <- tibble(
mu_griglia = mu_griglia,
prior = prior / sum(prior), # Normalizzazione del prior
posterior = posterior # Posterior già normalizzato
)
# Preparazione dei dati in formato lungo per ggplot2
long_data <- dat |>
pivot_longer(
cols = c(prior, posterior),
names_to = "distribution",
values_to = "density"
)
# Grafico con ggplot2
long_data |>
ggplot(aes(x = mu_griglia, y = density, color = distribution, linetype = distribution)) +
geom_line(size = 1.2) +
labs(
title = "Distribuzione a Posteriori e Prior della Media",
x = "Media",
y = "Densità",
color = "Distribuzione",
linetype = "Distribuzione"
) +
theme(
plot.title = element_text(hjust = 0.5),
legend.position = "bottom"
)
50.6 Campionamento dalla Posterior
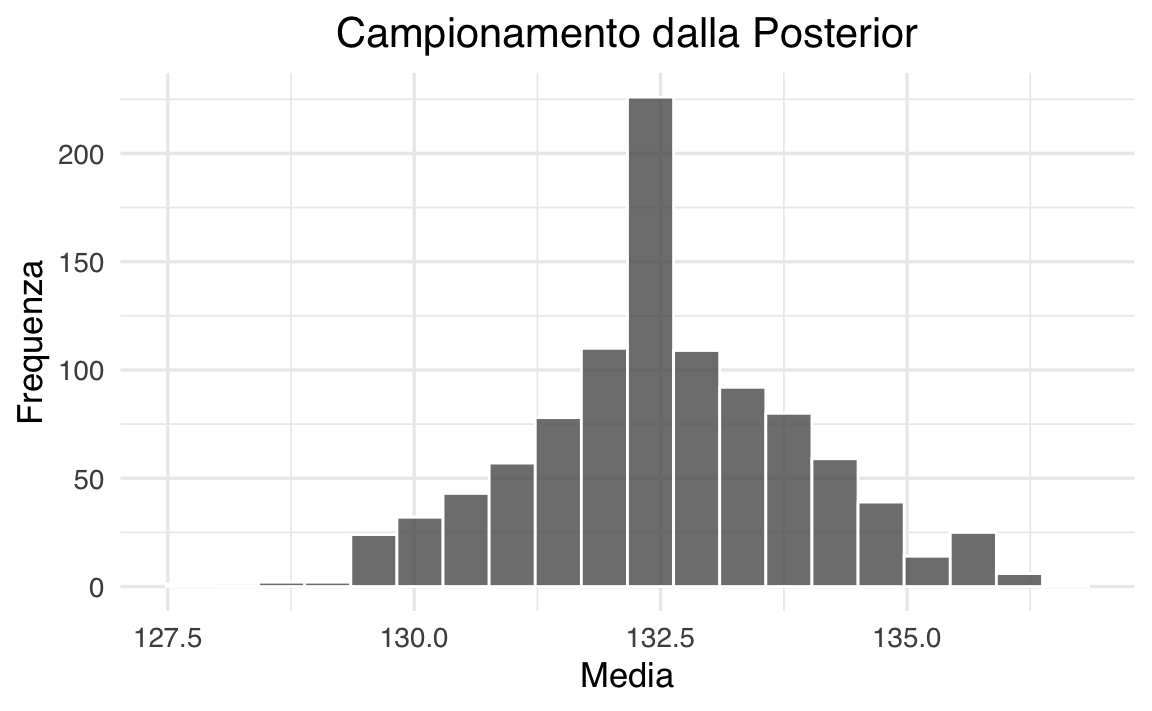
Generiamo un campione dalla distribuzione a posteriori.
# Set seed for reproducibility
set.seed(123)
# Sampling from the posterior distribution
indice_campionato <- sample(1:length(mu_griglia), size = 1000, replace = TRUE, prob = posterior)
media_campionata <- mu_griglia[indice_campionato]
# Create a dataframe for ggplot2
sample_df <- tibble(media_campionata = media_campionata)
# Histogram using ggplot2
ggplot(sample_df, aes(x = media_campionata)) +
geom_histogram(
bins = 20,
color = "white",
alpha = 0.8
) +
labs(
title = "Campionamento dalla Posterior",
x = "Media",
y = "Frequenza"
) +
theme(
plot.title = element_text(hjust = 0.5)
)
50.7 Calcolo della Log-Verosimiglianza
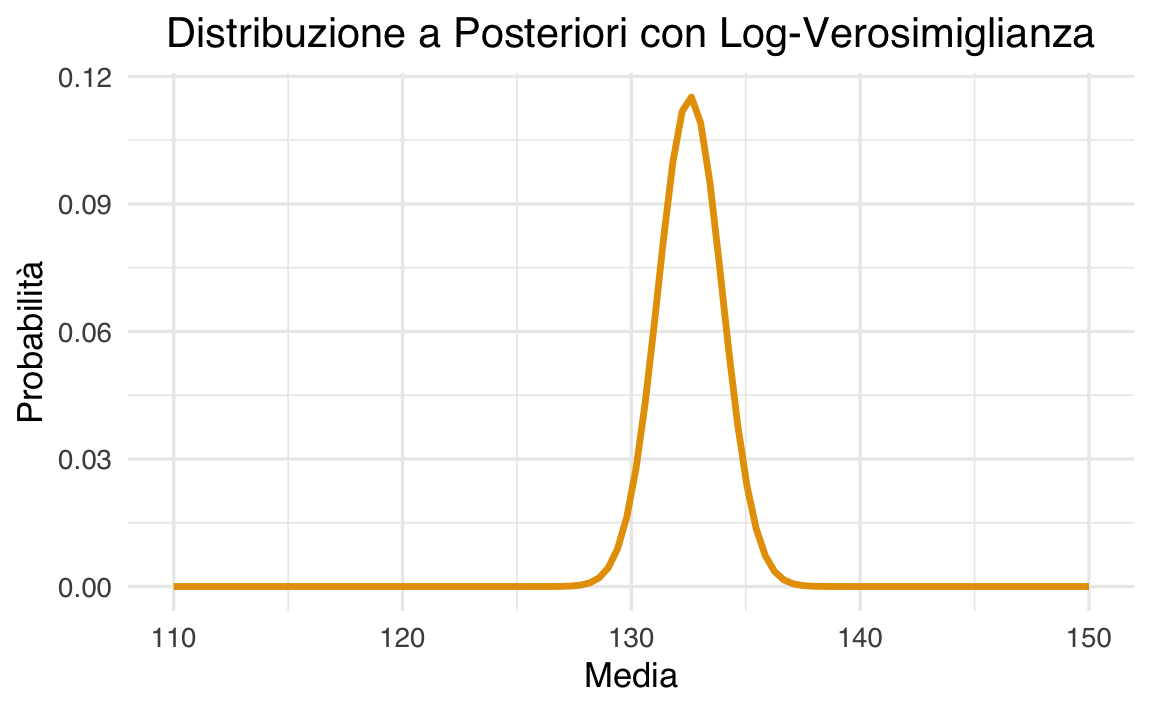
Utilizziamo i logaritmi per migliorare la stabilità numerica.
# Calcolo log-likelihood, log-prior e posterior
log_likelihood <- sapply(mu_griglia, function(mu) {
sum(dnorm(campione, mean = mu, sd = sigma_conosciuta, log = TRUE))
})
log_prior <- dnorm(mu_griglia, mean = 140, sd = 3, log = TRUE)
log_posterior_non_norm <- log_likelihood + log_prior
log_posterior <- log_posterior_non_norm - max(log_posterior_non_norm) # Stabilizzazione numerica
posterior <- exp(log_posterior) / sum(exp(log_posterior))
# Creazione del dataframe per il grafico
dat <- tibble(
mu_griglia = mu_griglia,
posterior = posterior
)
# Grafico con ggplot2
dat |>
ggplot(aes(x = mu_griglia, y = posterior)) +
geom_line(size = 1.2) +
labs(
title = "Distribuzione a Posteriori con Log-Verosimiglianza",
x = "Media",
y = "Probabilità"
) +
theme(
plot.title = element_text(hjust = 0.5), # Centra il titolo
legend.position = "none" # Rimuove la legenda per una linea singola
)
50.8 Estensione alla Deviazione Standard Ignota
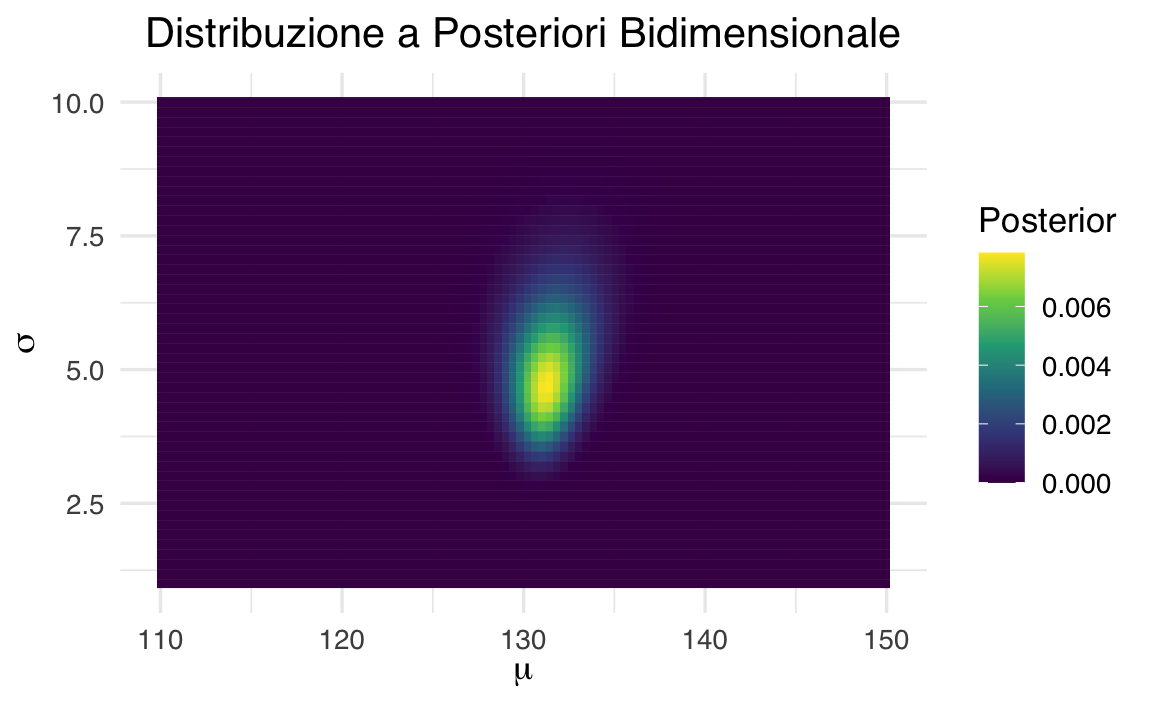
Per una griglia bidimensionale di valori di \(\mu\) e \(\sigma\):
mu_griglia <- seq(110, 150, length.out = 100)
sigma_griglia <- seq(1, 10, length.out = 50)
# Create combinations of mu and sigma using expand.grid
grid <- expand.grid(mu = mu_griglia, sigma = sigma_griglia)
# Compute the log-likelihood for each combination of mu and sigma
log_likelihood <- apply(grid, 1, function(params) {
mu <- params["mu"]
sigma <- params["sigma"]
sum(dnorm(campione, mean = mu, sd = sigma, log = TRUE))
})
# Reshape log-likelihood into a matrix
log_likelihood_2d <- matrix(log_likelihood, nrow = length(mu_griglia), ncol = length(sigma_griglia))
# Compute priors for mu and sigma
log_prior_mu <- dnorm(mu_griglia, mean = 140, sd = 5, log = TRUE)
log_prior_sigma <- dnorm(sigma_griglia, mean = 5, sd = 2, log = TRUE)
# Combine priors into a grid
log_prior_2d <- outer(log_prior_mu, log_prior_sigma, "+")
# Compute log-posterior
log_posterior_2d <- log_likelihood_2d + log_prior_2d
log_posterior_2d <- log_posterior_2d - max(log_posterior_2d) # Stabilize
posterior_2d <- exp(log_posterior_2d)
posterior_2d <- posterior_2d / sum(posterior_2d) # Normalize
# Convert posterior_2d to a data frame for visualization
posterior_df <- reshape2::melt(posterior_2d)
names(posterior_df) <- c("mu_idx", "sigma_idx", "posterior")
posterior_df$mu <- mu_griglia[posterior_df$mu_idx]
posterior_df$sigma <- sigma_griglia[posterior_df$sigma_idx]
# Plot the posterior distribution
ggplot(posterior_df, aes(x = mu, y = sigma, fill = posterior)) +
geom_tile() +
scale_fill_viridis_c(name = "Posterior") +
labs(
title = "Distribuzione a Posteriori Bidimensionale",
x = expression(mu),
y = expression(sigma)
) +
theme(
plot.title = element_text(hjust = 0.5),
legend.position = "right"
)
50.9 Riflessioni Conclusive
Quando si passa alla stima simultanea di più parametri, come la media (\(\mu\)) e la deviazione standard (\(\sigma\)), l’analisi diventa notevolmente più complessa. Questo perché occorre considerare un numero molto maggiore di combinazioni di parametri rispetto alla stima di un solo parametro, aumentando così il carico computazionale. Inoltre, la scelta delle priors per ciascun parametro richiede particolare attenzione, poiché queste influenzeranno in modo diretto le stime a posteriori.
In scenari dove lo spazio dei parametri è multidimensionale o quando l’esplorazione della griglia diventa impraticabile, l’uso di metodi avanzati come il campionamento di Markov Chain Monte Carlo (MCMC) diventa indispensabile. Questi metodi permettono di campionare in modo efficiente dalla distribuzione a posteriori, senza la necessità di esplorare esplicitamente ogni combinazione possibile di parametri, rendendo l’analisi più gestibile anche in contesti complessi.
In conclusione, l’estensione dell’approccio bayesiano a problemi con più parametri sconosciuti richiede un’attenzione ancora maggiore nella definizione dello spazio dei parametri, nella selezione delle priors appropriate e nel calcolo delle distribuzioni a posteriori. L’adozione di tecniche come l’MCMC può facilitare questo processo, permettendo di affrontare in modo efficiente problemi che altrimenti sarebbero proibitivi dal punto di vista computazionale.
Esercizi
Quali sono i vantaggi dell’uso del metodo della griglia per il calcolo della distribuzione a posteriori? Quali sono le principali limitazioni di questo approccio?
Qual è il ruolo della funzione di verosimiglianza nell’inferenza bayesiana e come si combina con la distribuzione a priori per ottenere la distribuzione a posteriori?
In che modo l’uso di una distribuzione a priori informativa può influenzare l’inferenza bayesiana rispetto a una a priori uniforme? Fai riferimento a un esempio pratico.
Perché in alcuni casi è preferibile lavorare con la log-verosimiglianza anziché con la verosimiglianza stessa? Spiega i vantaggi di questa trasformazione.
Qual è il significato del campionamento dalla distribuzione a posteriori e perché è utile in un’analisi bayesiana? Quali sono alcuni metodi alternativi per ottenere campioni dalla posterior quando il metodo della griglia non è praticabile?
Utilizzando i dati della Satisfaction With Life Scale (SWLS) raccolti dagli studenti, costruisci un modello bayesiano per stimare la media della soddisfazione di vita. Segui questi passaggi:
-
Carica i dati della SWLS e visualizza la distribuzione delle risposte.
-
Definisci una griglia di valori possibili per la media della soddisfazione di vita (ad esempio, da 1 a 7 se il punteggio della SWLS è su una scala Likert 1-7).
-
Assumi che la deviazione standard sia nota (puoi stimarla dai dati o usare un valore ragionevole, come 1).
-
Calcola la funzione di verosimiglianza per ogni valore della griglia assumendo una distribuzione normale.
-
Imposta una distribuzione a priori (uniforme o gaussiana centrata su un valore atteso, ad esempio 4).
-
Calcola la distribuzione a posteriori e normalizzala.
-
Visualizza la distribuzione a posteriori della media della soddisfazione di vita.
- Estrai campioni dalla distribuzione a posteriori e calcola un intervallo di credibilità al 94%.
Esegui il codice in R e commenta i risultati ottenuti.
Consegna: carica il file .qmd con le risposte, convertito in PDF, su Moodle.
-
Il metodo della griglia ha il vantaggio di essere intuitivo e facilmente implementabile, poiché calcola direttamente la distribuzione a posteriori valutando la funzione di verosimiglianza e il prior su una griglia di valori possibili per il parametro di interesse. Questo approccio permette una visualizzazione chiara della distribuzione a posteriori e facilita il confronto tra diversi prior.
Tuttavia, presenta alcune limitazioni:
-
Scalabilità: Diventa impraticabile quando il numero di parametri cresce, poiché il numero di combinazioni nella griglia aumenta esponenzialmente.
-
Risoluzione: La precisione dell’inferenza dipende dalla densità della griglia, e una griglia troppo fine può essere computazionalmente costosa.
- Difficoltà per modelli complessi: Non è adatto per modelli con parametri ad alta dimensionalità o con distribuzioni a posteriori complesse.
-
Scalabilità: Diventa impraticabile quando il numero di parametri cresce, poiché il numero di combinazioni nella griglia aumenta esponenzialmente.
-
La funzione di verosimiglianza rappresenta la probabilità di osservare i dati dati i valori del parametro di interesse. Nell’inferenza bayesiana, questa informazione viene combinata con la distribuzione a priori attraverso il teorema di Bayes, che permette di aggiornare la conoscenza pregressa con le nuove osservazioni.
Matematicamente, la distribuzione a posteriori è data da:
\[ p(\theta \mid D) = \frac{p(D \mid \theta) p(\theta)}{p(D)} \]
dove:
-
\(p(D \mid \theta)\) è la verosimiglianza, che misura quanto bene il parametro \(\theta\) spiega i dati \(D\).
-
\(p(\theta)\) è il prior, che rappresenta la conoscenza iniziale sul parametro.
- \(p(D)\) è la costante di normalizzazione.
L’aggiornamento bayesiano consente di affinare le stime dei parametri alla luce di nuove evidenze in modo sistematico e coerente.
-
\(p(D \mid \theta)\) è la verosimiglianza, che misura quanto bene il parametro \(\theta\) spiega i dati \(D\).
-
L’uso di un prior informativo consente di incorporare conoscenze pregresse nella stima dei parametri, riducendo l’incertezza quando i dati sono scarsi. Tuttavia, se il prior è troppo forte rispetto ai dati, potrebbe dominare la distribuzione a posteriori e introdurre un bias nelle stime.
Esempio pratico: Supponiamo di voler stimare il QI medio di una popolazione sulla base di un piccolo campione. Se usiamo un prior informativo centrato su 140 con una deviazione standard di 3, la distribuzione a posteriori sarà fortemente influenzata da questa assunzione. Se invece utilizziamo un prior uniforme, i dati avranno un impatto maggiore sulla stima a posteriori.
In generale, i prior informativi sono utili quando abbiamo conoscenze affidabili da incorporare, mentre i prior non informativi sono preferibili quando vogliamo lasciare che i dati guidino l’inferenza.
-
Lavorare con la log-verosimiglianza presenta diversi vantaggi:
-
Stabilità numerica: La moltiplicazione di molte probabilità può portare a valori molto piccoli che causano underflow numerico. Usare il logaritmo trasforma i prodotti in somme, evitando questi problemi.
-
Efficienza computazionale: Le somme sono più efficienti da calcolare rispetto ai prodotti, specialmente per modelli con molti dati.
- Interpretabilità: La log-verosimiglianza fornisce una misura più chiara della bontà di adattamento del modello, poiché la somma dei log-likelihood è direttamente proporzionale alla probabilità complessiva dei dati dato il parametro.
Per questi motivi, la log-verosimiglianza è ampiamente usata in applicazioni statistiche e machine learning.
-
Stabilità numerica: La moltiplicazione di molte probabilità può portare a valori molto piccoli che causano underflow numerico. Usare il logaritmo trasforma i prodotti in somme, evitando questi problemi.
-
Il campionamento dalla distribuzione a posteriori permette di ottenere stime dei parametri e di quantificare l’incertezza in modo efficace. Poiché la posterior rappresenta la nostra credenza aggiornata sul parametro dopo aver osservato i dati, il campionamento consente di generare simulazioni di possibili valori di \(\theta\).
Utilità del campionamento:
- Permette di calcolare intervalli di credibilità.
- Consente di effettuare inferenze basate sulla distribuzione completa, anziché su un singolo valore puntuale.
- È utile per simulare previsioni e testare ipotesi.
Metodi alternativi per il campionamento dalla posterior:
-
Metropolis-Hastings (MCMC): Un algoritmo di Markov Chain Monte Carlo che permette di esplorare distribuzioni complesse.
-
Gibbs Sampling: Un metodo MCMC particolarmente utile per modelli con più parametri condizionali noti.
- Hamiltonian Monte Carlo (HMC): Utilizza gradienti per esplorare lo spazio dei parametri in modo efficiente, come implementato in Stan.
Quando il metodo della griglia non è praticabile, questi metodi consentono di stimare la posterior in modo efficiente anche per modelli complessi e ad alta dimensionalità.
- Permette di calcolare intervalli di credibilità.
Ecco un codice in R che segue i passaggi richiesti.
# Caricamento librerie necessarie
library(ggplot2)
library(dplyr)
library(tibble)
# Dati SWLS
swls_data <- data.frame(
soddisfazione = c(4.2, 5.1, 4.7, 4.3, 5.5, 4.9, 4.8, 5.0, 4.6, 4.4)
)
# Usando la deviazione standard campionaria
sigma_conosciuta <- sd(swls_data$soddisfazione)
n <- nrow(swls_data)
mean_x <- mean(swls_data$soddisfazione)
cat("Deviazione standard campionaria:", sigma_conosciuta, "\n")
cat("Media campionaria:", mean_x, "\n")
# Definizione della griglia più fine e centrata intorno alla media campionaria
mu_griglia <- seq(mean_x - 3*sigma_conosciuta/sqrt(n),
mean_x + 3*sigma_conosciuta/sqrt(n),
length.out = 1000)
# Calcolo della verosimiglianza
log_likelihood <- numeric(length(mu_griglia))
for (i in seq_along(mu_griglia)) {
# Utilizzo della log-likelihood per evitare problemi numerici
log_likelihood[i] <- sum(dnorm(swls_data$soddisfazione,
mean = mu_griglia[i],
sd = sigma_conosciuta,
log = TRUE))
}
# Prior uniforme (in scala logaritmica)
log_prior <- rep(0, length(mu_griglia))
# Calcolo della posteriori
log_posterior <- log_likelihood + log_prior
posterior <- exp(log_posterior - max(log_posterior))
posterior <- posterior / sum(posterior)
# Campionamento e calcolo statistiche
samples_grid <- sample(mu_griglia, size = 10000, replace = TRUE, prob = posterior)
mean_post_grid <- mean(samples_grid)
sd_post_grid <- sd(samples_grid)
ci_grid <- quantile(samples_grid, c(0.03, 0.97))
results <- tibble(
`Media Posteriori` = c(mean_post_grid),
`Dev. Std. Posteriori` = c(sd_post_grid)
)
# Visualizzazione risultati
print(results)
# Plot della distribuzione a posteriori per entrambi i metodi
ggplot() +
geom_line(data = data.frame(mu = mu_griglia, density = posterior),
aes(x = mu, y = density, color = "Griglia")) +
labs(title = "Distribuzione a Posteriori",
x = "Media", y = "Densità")
# Stampa intervallo di credibilità al 94%
cat("\nIntervallo di credibilità al 94% (metodo griglia):\n")
print(ci_grid)Conclusione:
Il modello bayesiano ci fornisce una stima della media della soddisfazione di vita con un intervallo di credibilità, quantificando l’incertezza in modo rigoroso.
Informazioni sull’Ambiente di Sviluppo
sessionInfo()
#> R version 4.4.2 (2024-10-31)
#> Platform: aarch64-apple-darwin20
#> Running under: macOS Sequoia 15.3.2
#>
#> Matrix products: default
#> BLAS: /Library/Frameworks/R.framework/Versions/4.4-arm64/Resources/lib/libRblas.0.dylib
#> LAPACK: /Library/Frameworks/R.framework/Versions/4.4-arm64/Resources/lib/libRlapack.dylib; LAPACK version 3.12.0
#>
#> locale:
#> [1] C/UTF-8/C/C/C/C
#>
#> time zone: Europe/Rome
#> tzcode source: internal
#>
#> attached base packages:
#> [1] stats graphics grDevices utils datasets methods base
#>
#> other attached packages:
#> [1] reshape2_1.4.4 thematic_0.1.6 MetBrewer_0.2.0 ggokabeito_0.1.0
#> [5] see_0.11.0 gridExtra_2.3 patchwork_1.3.0 bayesplot_1.11.1
#> [9] psych_2.5.3 scales_1.3.0 markdown_2.0 knitr_1.50
#> [13] lubridate_1.9.4 forcats_1.0.0 stringr_1.5.1 dplyr_1.1.4
#> [17] purrr_1.0.4 readr_2.1.5 tidyr_1.3.1 tibble_3.2.1
#> [21] ggplot2_3.5.1 tidyverse_2.0.0 rio_1.2.3 here_1.0.1
#>
#> loaded via a namespace (and not attached):
#> [1] generics_0.1.3 stringi_1.8.4 lattice_0.22-6 hms_1.1.3
#> [5] digest_0.6.37 magrittr_2.0.3 evaluate_1.0.3 grid_4.4.2
#> [9] timechange_0.3.0 fastmap_1.2.0 plyr_1.8.9 rprojroot_2.0.4
#> [13] jsonlite_1.9.1 viridisLite_0.4.2 mnormt_2.1.1 cli_3.6.4
#> [17] rlang_1.1.5 munsell_0.5.1 withr_3.0.2 tools_4.4.2
#> [21] parallel_4.4.2 tzdb_0.5.0 colorspace_2.1-1 pacman_0.5.1
#> [25] vctrs_0.6.5 R6_2.6.1 lifecycle_1.0.4 htmlwidgets_1.6.4
#> [29] pkgconfig_2.0.3 pillar_1.10.1 gtable_0.3.6 Rcpp_1.0.14
#> [33] glue_1.8.0 xfun_0.51 tidyselect_1.2.1 rstudioapi_0.17.1
#> [37] farver_2.1.2 htmltools_0.5.8.1 nlme_3.1-167 labeling_0.4.3
#> [41] rmarkdown_2.29 compiler_4.4.2