54 Distribuzione predittiva a posteriori
54.1 Introduzione
Nel contesto dell’inferenza bayesiana, uno degli obiettivi principali è non solo stimare i parametri di un modello (ad esempio, la probabilità \(p\) di successo in un esperimento binomiale) ma anche fare previsioni su dati futuri basandosi su ciò che abbiamo osservato. La distribuzione predittiva a posteriori risponde proprio a questa esigenza, combinando:
- La nostra incertezza sui parametri descritta dalla distribuzione a posteriori.
- La variabilità intrinseca del processo che genera i dati futuri.
In termini semplici, la distribuzione predittiva a posteriori ci dice quali risultati futuri sono plausibili, dato ciò che sappiamo dai dati osservati e dal modello.
54.2 Definizione Formale
Supponiamo di avere un insieme di dati osservati \(y = \{y_1, y_2, \ldots, y_n\}\), generati da un modello che dipende da un parametro sconosciuto \(\theta\). Il parametro \(\theta\) può rappresentare qualsiasi caratteristica del modello, come una probabilità di successo, una media, o un coefficiente in un modello di regressione. Inizialmente, la nostra conoscenza su \(\theta\) è rappresentata dalla distribuzione a priori \(p(\theta)\), che riflette ciò che sappiamo (o non sappiamo) su \(\theta\) prima di osservare i dati.
Dopo aver osservato i dati \(y\), possiamo aggiornare la nostra conoscenza su \(\theta\) utilizzando la formula di Bayes per calcolare la distribuzione a posteriori \(p(\theta \mid y)\):
\[ p(\theta \mid y) = \frac{p(y \mid \theta) p(\theta)}{p(y)}, \]
dove:
\(p(\theta \mid y)\): la distribuzione a posteriori rappresenta la nostra conoscenza aggiornata su \(\theta\) dopo aver osservato i dati.
\(p(y \mid \theta)\): la verosimiglianza è la probabilità di osservare i dati dati i parametri del modello.
\(p(\theta)\): la distribuzione a priori rappresenta la conoscenza iniziale su \(\theta\).
-
\(p(y)\): l’evidenza è la probabilità totale dei dati osservati, calcolata come:
\[ p(y) = \int p(y \mid \theta) p(\theta) \, d\theta. \]
54.2.1 Previsione di Nuovi Dati
Quando vogliamo prevedere un nuovo dato, indicato con \(\tilde{y}\), la nostra attenzione si sposta sulla distribuzione predittiva a posteriori, \(p(\tilde{y} \mid y)\).
54.2.1.1 Cosa rappresenta \(\tilde{y}\)?
- \(\tilde{y}\) rappresenta un dato futuro o non osservato. Ad esempio, se i dati \(y\) rappresentano il numero di successi osservati in una serie di lanci di una moneta, \(\tilde{y}\) potrebbe rappresentare il numero di successi in una nuova serie di lanci.
- L’obiettivo è stimare \(p(\tilde{y} \mid y)\), cioè la probabilità del nuovo dato \(\tilde{y}\) dato ciò che abbiamo osservato in \(y\).
54.2.1.2 Cosa rappresenta \(p(\tilde{y} \mid \theta)\)?
- \(p(\tilde{y} \mid \theta)\) è la probabilità del nuovo dato \(\tilde{y}\) dato un particolare valore del parametro \(\theta\).
- Ad esempio, in un modello binomiale, \(p(\tilde{y} \mid \theta)\) corrisponde alla probabilità di ottenere \(\tilde{y}\) successi su \(n_{\text{new}}\) prove, data la probabilità di successo \(\theta\).
54.2.1.3 Combinazione di \(p(\tilde{y} \mid \theta)\) con \(p(\theta \mid y)\)
Poiché non conosciamo esattamente \(\theta\), dobbiamo considerare tutte le possibili ipotesi su \(\theta\), pesandole in base alla loro probabilità a posteriori \(p(\theta \mid y)\). Questo porta alla formula per la distribuzione predittiva a posteriori:
\[ p(\tilde{y} \mid y) = \int p(\tilde{y} \mid \theta) p(\theta \mid y) \, d\theta. \]
54.2.1.4 Interpretazione
- La distribuzione predittiva a posteriori, \(p(\tilde{y} \mid y)\), rappresenta la nostra miglior stima della probabilità del nuovo dato \(\tilde{y}\), tenendo conto sia dei dati osservati \(y\) sia dell’incertezza su \(\theta\).
54.2.2 Caso Discreto
Se il parametro \(\theta\) assume un numero finito di valori, l’integrale si semplifica in una somma:
\[ p(\tilde{y} \mid y) = \sum_{\theta} p(\tilde{y} \mid \theta) p(\theta \mid y). \]
Questo approccio è utile nei modelli discreti o quando si approssimano i parametri con un numero finito di valori campionati.
54.3 Il Caso Beta-Binomiale
Consideriamo un classico esperimento binomiale: lanciare una moneta \(n\) volte e osservare il numero di successi \(y\) (ad esempio, il numero di “teste”). In un contesto bayesiano, il processo di analisi si articola in tre fasi:
-
Distribuzione a Priori: Prima di osservare i dati, formuliamo una distribuzione a priori sulla probabilità \(p\) di successo, che riflette le nostre conoscenze iniziali (o la loro assenza). Una scelta comune è la distribuzione Beta(\(\alpha, \beta\)), perché è flessibile e ben definita per probabilità. Per esempio:
- \(\alpha\) rappresenta il numero “fittizio” di successi osservati.
- \(\beta\) rappresenta il numero “fittizio” di insuccessi osservati.
-
Distribuzione a Posteriori: Dopo aver osservato \(y\) successi su \(n\) prove, aggiorniamo la distribuzione a priori combinandola con i dati osservati, ottenendo la distribuzione a posteriori di \(p\):
\[ \alpha_{\text{post}} = \alpha_{\text{prior}} + y, \quad \beta_{\text{post}} = \beta_{\text{prior}} + (n - y). \]
Questa distribuzione rappresenta ciò che sappiamo di \(p\) dopo aver osservato i dati.
-
Distribuzione Predittiva a Posteriori: Per prevedere il numero di successi futuri \(y_{\text{new}}\) in un nuovo esperimento con \(n_{\text{new}}\) prove, combiniamo la distribuzione a posteriori di \(p\) con la variabilità intrinseca del processo binomiale. In pratica:
- Campioniamo \(p\) dalla distribuzione a posteriori (\(p \sim \text{Beta}(\alpha_{\text{post}}, \beta_{\text{post}})\)).
- Usiamo ciascun campione di \(p\) per simulare nuovi dati (\(y_{\text{new}} \sim \text{Binomiale}(n_{\text{new}}, p)\)).
Questo processo tiene conto sia dell’incertezza sui parametri (\(p\)) sia della variabilità intrinseca nei dati futuri.
54.4 Simulazione della Distribuzione Predittiva a Posteriori
54.4.1 Impostazione dei Parametri
- Supponiamo di aver osservato \(y = 70\) successi su \(n = 100\) prove.
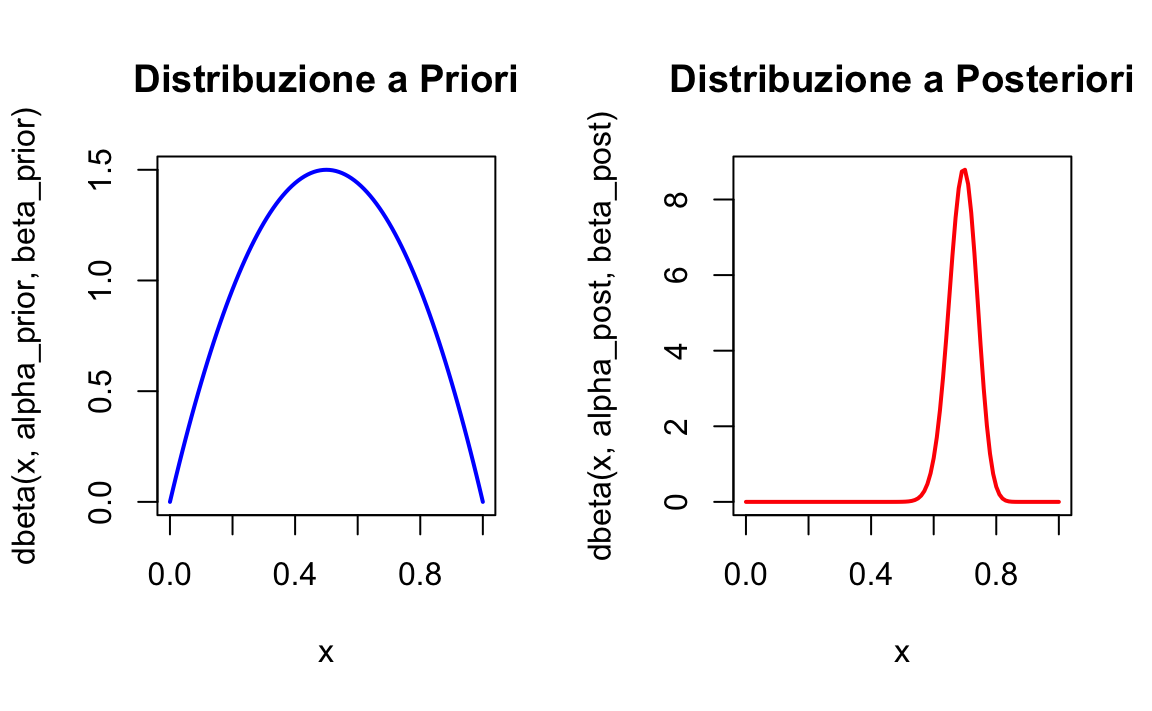
- Utilizziamo una distribuzione a priori Beta(\(2, 2\)), che rappresenta una conoscenza iniziale debolmente informativa, con una leggera preferenza per \(p \approx 0.5\).
54.4.2 Calcolo della Distribuzione a Posteriori
-
Aggiorniamo la distribuzione a priori con i dati osservati. I parametri aggiornati sono:
\[ \alpha_{\text{post}} = \alpha_{\text{prior}} + y = 2 + 70 = 72, \quad \beta_{\text{post}} = \beta_{\text{prior}} + (n - y) = 2 + 30 = 32. \]
La distribuzione a posteriori è quindi \(p \sim \text{Beta}(72, 32)\), che descrive la probabilità aggiornata di successo basata sui dati.
54.4.3 Simulazione dei Dati Futuri
Generiamo \(n_{\text{sim}} = 1000\) campioni di \(p\) dalla distribuzione a posteriori Beta(72, 32).
-
Per ogni campione di \(p\), simuliamo \(y_{\text{new}}\), il numero di successi futuri su \(n_{\text{new}} = 10\) prove, utilizzando la distribuzione binomiale:
\[ y_{\text{new}} \sim \text{Binomiale}(n_{\text{new}} = 10, p). \]
-
Infine, convertiamo \(y_{\text{new}}\) in proporzioni predette:
\[ \text{Proporzione predetta} = \frac{y_{\text{new}}}{n_{\text{new}}}. \]
# Impostazione del seed per riproducibilità
set.seed(123)
# Parametri osservati
y <- 70
n <- 100
# Parametri a priori
alpha_prior <- 2
beta_prior <- 2
# Calcolo dei parametri a posteriori
alpha_post <- alpha_prior + y
beta_post <- beta_prior + (n - y)
# Simulazione della distribuzione a posteriori
p_samples <- rbeta(1000, alpha_post, beta_post)
# Simulazione di nuovi dati per n_new = 10
y_preds <- sapply(p_samples, function(p) {
rbinom(1, 10, p)
})
# Calcolo delle proporzioni predette
prop_preds <- y_preds / 1054.4.4 Risultati Attesi
-
Distribuzione a Posteriori di \(p\):
- Centrata attorno a \(0.7\), con una varianza ridotta grazie alla dimensione del campione \(n = 100\).
-
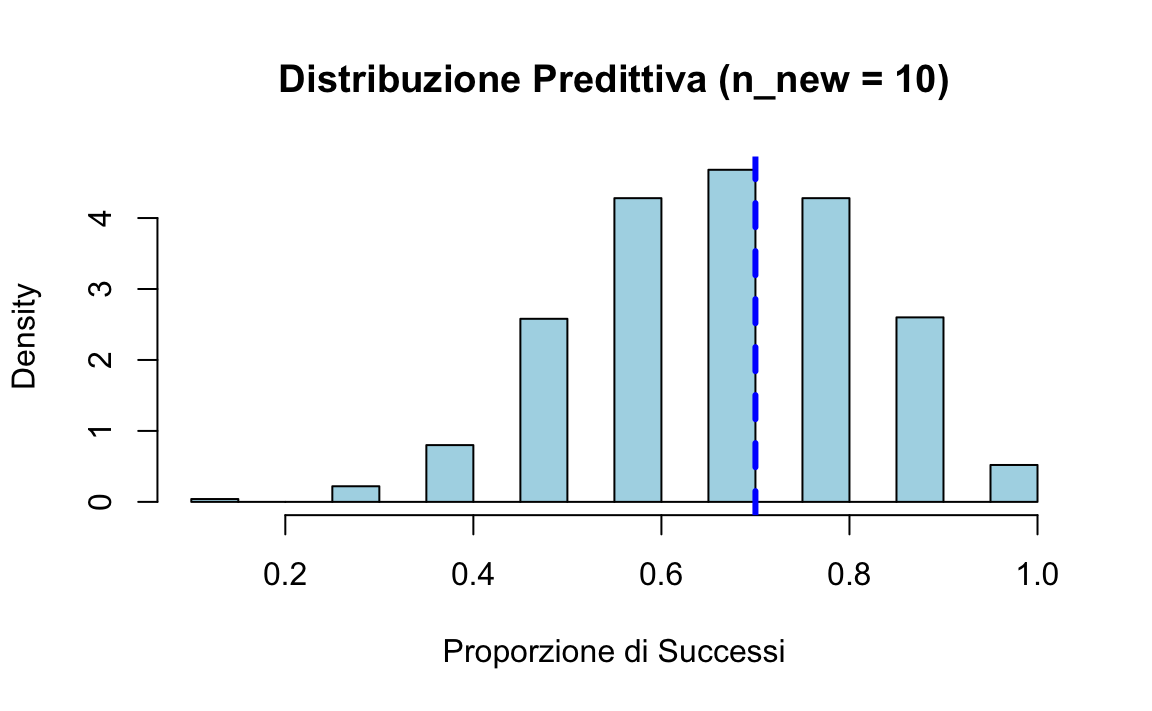
Distribuzione Predittiva per \(n_{\text{new}} = 10\):
- Variabilità più ampia rispetto a \(p\), dovuta al numero ridotto di prove (\(n_{\text{new}} = 10\)).
- Riflette sia l’incertezza su \(p\) sia la variabilità intrinseca dei dati futuri.
54.4.5 Spiegazione del Codice
Simulazione di nuovi dati per \(n_{\text{new}} = 10\):
-
p_samplescontiene \(1000\) valori di \(p\) simulati dalla distribuzione Beta(72, 32). - Per ciascun \(p\),
rbinom(1, 10, p)genera un valore di \(y_{\text{new}}\), simulando il numero di successi futuri su \(n_{\text{new}} = 10\) prove.
Risultato: un vettore di 1000 valori di \(y_{\text{new}}\), uno per ciascun campione di \(p\).
Calcolo delle proporzioni predette:
prop_preds <- y_preds / 10- Divide ciascun valore di \(y_{\text{new}}\) per \(n_{\text{new}} = 10\), ottenendo le proporzioni di successi predette.
Risultato: un vettore di 1000 proporzioni predette (\(y_{\text{new}} / n_{\text{new}}\)).
54.4.6 Interpretazione
- La distribuzione a posteriori di \(p\), Beta(72, 32), è centrata attorno a \(p \approx 0.7\), coerente con i dati osservati (\(y / n = 0.7\)).
- Le proporzioni predette mostrano la variabilità combinata dell’incertezza su \(p\) (dalla distribuzione a posteriori) e della variabilità binomiale per un campione futuro di 10 prove.
- L’istogramma delle proporzioni predette è più ampio rispetto alla distribuzione a posteriori di \(p\), riflettendo l’incertezza aggiuntiva derivante dal numero ridotto di prove (\(n_{\text{new}} = 10\)).
54.5 Posterior Predictive Check (PP-Check)
La distribuzione predittiva a posteriori ottenuta dalla simulazione (\(n_{\text{new}} = 10\)) può essere utilizzata per effettuare un Posterior Predictive Check (PP-Check). Questo controllo confronta i dati osservati con i dati simulati dal modello per verificare se il modello è in grado di riprodurre caratteristiche rilevanti dei dati osservati.
In questa simulazione, il Posterior Predictive-Check suggerisce che il modello è ben specificato per i dati osservati (\(y = 70\), \(n = 100\)): la proporzione osservata (\(0.7\)) è vicina al centro della distribuzione predittiva. Questo significa che il modello può essere considerato valido per fare previsioni sui dati futuri, almeno nel contesto specificato.
54.6 Riflessioni Conclusive
La distribuzione predittiva a posteriori è uno strumento centrale nell’inferenza bayesiana, poiché consente di fare previsioni sui dati futuri integrando l’incertezza sui parametri del modello con la variabilità intrinseca del processo generativo. Questa capacità va oltre la semplice stima dei parametri, permettendo di confrontare le previsioni del modello con i dati reali, un passaggio fondamentale per verificare la coerenza e l’utilità del modello stesso.
Nel flusso di lavoro bayesiano, la distribuzione predittiva a posteriori svolge un ruolo chiave nella valutazione del modello. Ad esempio, consente di effettuare controlli predittivi a posteriori per identificare discrepanze tra i dati osservati e quelli previsti. Tali controlli aiutano a diagnosticare problemi di specificazione del modello, a valutare l’adeguatezza delle scelte a priori e a guidare eventuali revisioni del modello.
Inoltre, il caso beta-binomiale utilizzato in questo capitolo rappresenta un esempio intuitivo e potente: evidenzia come l’incertezza sui parametri possa essere tradotta in previsioni probabilistiche robuste, senza la necessità di fare assunzioni rigide o non realistiche. Questo approccio non solo formalizza l’incertezza in modo rigoroso, ma permette anche di comunicare le previsioni in modo trasparente e interpretabile, caratteristiche essenziali in ambito decisionale e scientifico.
In sintesi, la distribuzione predittiva a posteriori è un elemento fondamentale della modellazione bayesiana, che lega l’inferenza paramatrica alla previsione empirica, contribuendo a rendere l’intero processo inferenziale più affidabile, interpretabile e applicabile a scenari complessi.
Esercizi
Informazioni sull’Ambiente di Sviluppo
sessionInfo()
#> R version 4.5.0 (2025-04-11)
#> Platform: aarch64-apple-darwin20
#> Running under: macOS Sequoia 15.5
#>
#> Matrix products: default
#> BLAS: /Library/Frameworks/R.framework/Versions/4.5-arm64/Resources/lib/libRblas.0.dylib
#> LAPACK: /Library/Frameworks/R.framework/Versions/4.5-arm64/Resources/lib/libRlapack.dylib; LAPACK version 3.12.1
#>
#> locale:
#> [1] C/UTF-8/C/C/C/C
#>
#> time zone: Europe/Rome
#> tzcode source: internal
#>
#> attached base packages:
#> [1] stats graphics grDevices utils datasets methods base
#>
#> other attached packages:
#> [1] thematic_0.1.7 MetBrewer_0.2.0 ggokabeito_0.1.0 see_0.11.0
#> [5] gridExtra_2.3 patchwork_1.3.0 bayesplot_1.13.0 psych_2.5.3
#> [9] scales_1.4.0 markdown_2.0 knitr_1.50 lubridate_1.9.4
#> [13] forcats_1.0.0 stringr_1.5.1 dplyr_1.1.4 purrr_1.0.4
#> [17] readr_2.1.5 tidyr_1.3.1 tibble_3.3.0 ggplot2_3.5.2
#> [21] tidyverse_2.0.0 rio_1.2.3 here_1.0.1
#>
#> loaded via a namespace (and not attached):
#> [1] generics_0.1.4 stringi_1.8.7 lattice_0.22-7
#> [4] hms_1.1.3 digest_0.6.37 magrittr_2.0.3
#> [7] evaluate_1.0.4 grid_4.5.0 timechange_0.3.0
#> [10] RColorBrewer_1.1-3 fastmap_1.2.0 rprojroot_2.0.4
#> [13] jsonlite_2.0.0 mnormt_2.1.1 cli_3.6.5
#> [16] rlang_1.1.6 withr_3.0.2 tools_4.5.0
#> [19] parallel_4.5.0 tzdb_0.5.0 pacman_0.5.1
#> [22] vctrs_0.6.5 R6_2.6.1 lifecycle_1.0.4
#> [25] htmlwidgets_1.6.4 pkgconfig_2.0.3 pillar_1.10.2
#> [28] gtable_0.3.6 glue_1.8.0 xfun_0.52
#> [31] tidyselect_1.2.1 rstudioapi_0.17.1 farver_2.1.2
#> [34] htmltools_0.5.8.1 nlme_3.1-168 rmarkdown_2.29
#> [37] compiler_4.5.0