43 La verosimiglianza gaussiana
Introduzione
La distribuzione gaussiana (o distribuzione normale) è una delle distribuzioni più utilizzate in statistica perché descrive molti fenomeni naturali e psicologici. In questo capitolo esploreremo come si calcola la verosimiglianza, ovvero la plausibilità dei parametri, nel caso della distribuzione normale.
43.1 Modello Gaussiano e Verosimiglianza
43.1.1 Caso di una Singola Osservazione
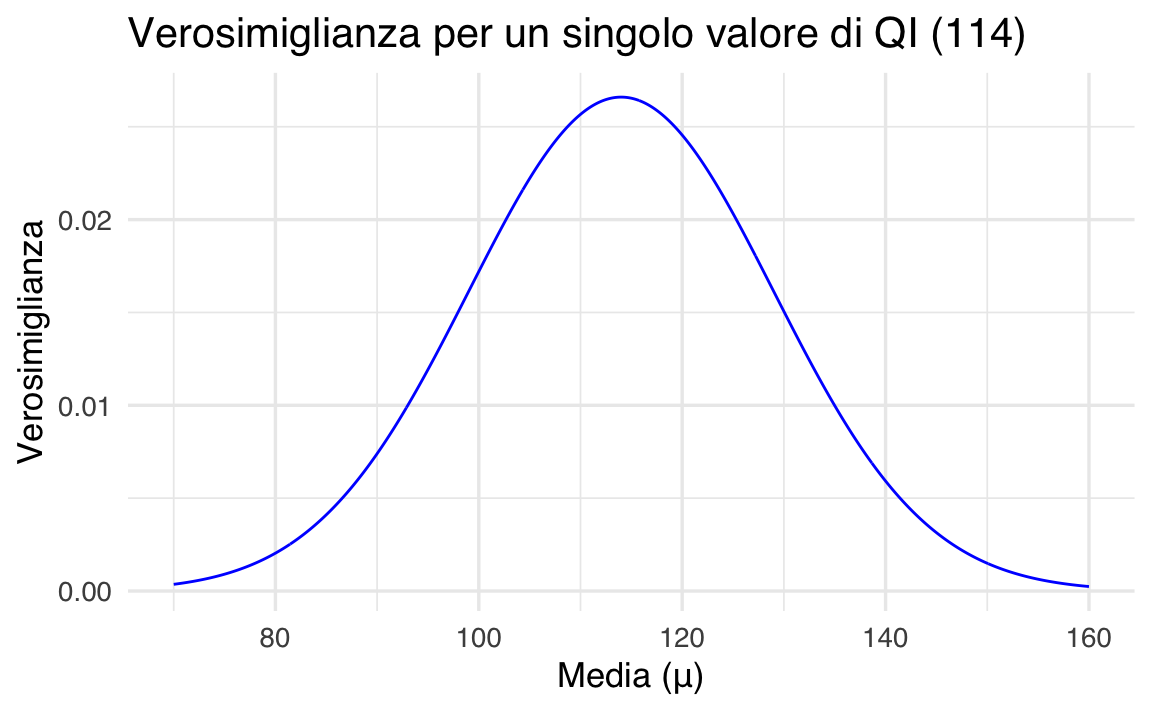
Immaginiamo di misurare il Quoziente Intellettivo (QI) di una persona e ottenere un valore specifico, ad esempio 114. Assumiamo che il QI segua una distribuzione normale con media \(\mu\) sconosciuta e deviazione standard \(\sigma\) nota (ad esempio \(\sigma = 15\)).
La funzione di densità di probabilità per una distribuzione normale è:
\[ f(y \mid \mu, \sigma) = \frac{1}{\sigma\sqrt{2\pi}} \exp\left(-\frac{(y-\mu)^2}{2\sigma^2}\right) , \]
dove:
- \(y\) è il valore osservato,
- \(\mu\) è la media (il parametro che vogliamo stimare),
- \(\sigma\) è la deviazione standard (conosciuta).
La verosimiglianza misura quanto diversi valori di \(\mu\) sono plausibili, dato il valore osservato (114).
Esempio pratico in R:
# Dati iniziali
y_obs <- 114
sigma <- 15
mu_values <- seq(70, 160, length.out = 1000)
# Calcolo della verosimiglianza
likelihood <- dnorm(y_obs, mean = mu_values, sd = sigma)
# Grafico della verosimiglianza
ggplot(data.frame(mu = mu_values, likelihood = likelihood), aes(x = mu, y = likelihood)) +
geom_line(color = okabe_ito_palette[2], linewidth = 1.2) +
labs(
title = "Verosimiglianza per un singolo valore di QI (114)",
x = "Media (μ)",
y = "Verosimiglianza"
)Qual è il valore migliore per \(\mu\)?
Il valore migliore di \(\mu\) sarà quello che rende massima la verosimiglianza. In questo semplice caso, è esattamente il valore osservato (114):
43.1.2 Log-Verosimiglianza
Spesso, per semplicità di calcolo, si usa la log-verosimiglianza, che trasforma i prodotti in somme, rendendo i calcoli più semplici e stabili:
\[ \log L(\mu \mid y, \sigma) = -\frac{1}{2}\log(2\pi) - \log(\sigma) - \frac{(y-\mu)^2}{2\sigma^2}. \]
Calcolo pratico con R:
# Funzione di log-verosimiglianza negativa usando dnorm()
negative_log_likelihood <- function(mu, y, sigma) {
# Ritorniamo il valore negativo della log-verosimiglianza
-dnorm(y, mean = mu, sd = sigma, log = TRUE)
}
result <- optim(
par = 100, # Valore iniziale
fn = negative_log_likelihood,
y = y_obs,
sigma = sigma,
method = "L-BFGS-B",
lower = 70,
upper = 160
)
mu_max_loglik <- result$par
cat("Il valore ottimale di μ dalla log-verosimiglianza è:", mu_max_loglik)
#> Il valore ottimale di μ dalla log-verosimiglianza è: 114In questo caso, otteniamo nuovamente \(\mu = 114\).
43.2 Campione di Osservazioni Indipendenti
Supponiamo di aver raccolto i punteggi alla scala BDI-II per 30 persone. Ciascun punteggio è un’osservazione indipendente da una distribuzione normale con media incognita \(\mu\) e deviazione standard nota \(\sigma = 6.5\).
# Dati osservati (punteggi BDI-II)
y <- c(
26, 35, 30, 25, 44, 30, 33, 43, 22, 43, 24, 19, 39, 31, 25,
28, 35, 30, 26, 31, 41, 36, 26, 35, 33, 28, 27, 34, 27, 22
)
sigma <- 6.543.2.1 Calcolo della Log-Verosimiglianza
Definiamo una funzione che calcola la log-verosimiglianza totale:
Esploriamo ora un intervallo di valori plausibili per \(\mu\) e calcoliamo la log-verosimiglianza per ciascun valore:
# Intervallo di valori possibili per μ
mu_range <- seq(mean(y) - 2 * sigma, mean(y) + 2 * sigma, length.out = 1000)
# Inizializza vettore dei risultati
log_lik_values <- numeric(length(mu_range))
# Ciclo esplicito per chiarezza didattica
for (i in seq_along(mu_range)) {
mu_val <- mu_range[i]
log_lik_values[i] <- log_likelihood(mu_val, y, sigma)
}43.2.2 Visualizzazione della Log-Verosimiglianza
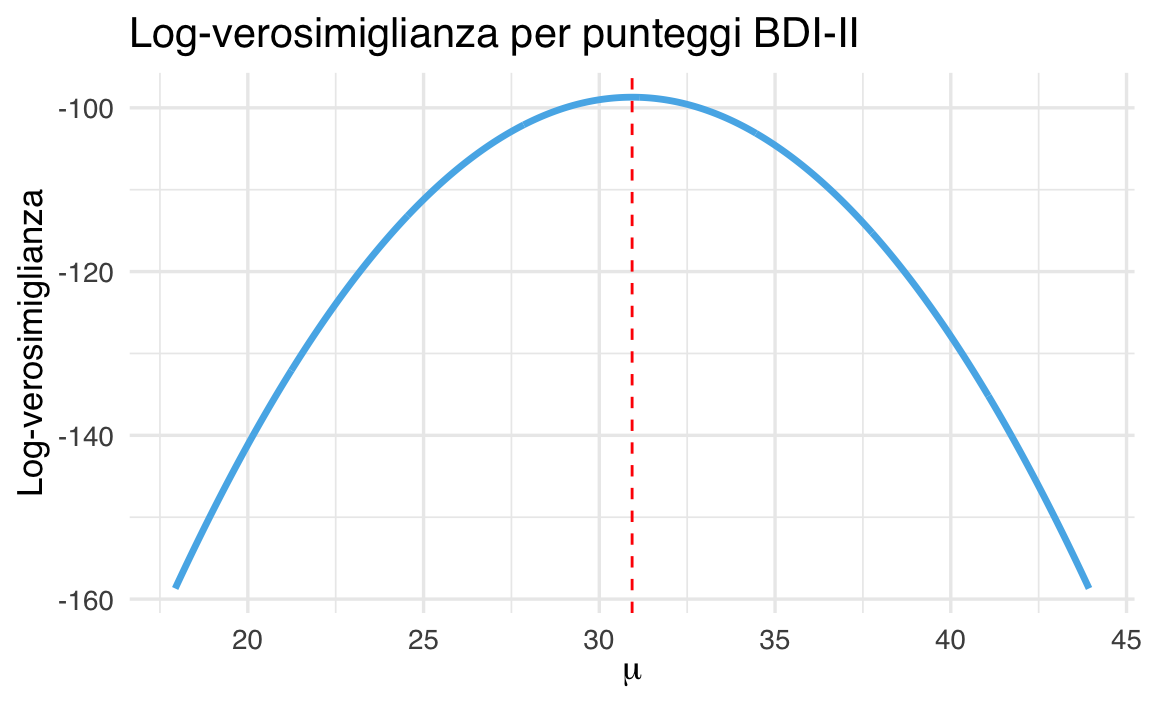
ggplot(
data.frame(mu = mu_range, log_likelihood = log_lik_values),
aes(x = mu, y = log_likelihood)
) +
geom_line(color = okabe_ito_palette[2], linewidth = 1.2) +
geom_vline(xintercept = mean(y), linetype = "dashed", color = "red") +
labs(
title = "Log-verosimiglianza per punteggi BDI-II",
x = expression(mu),
y = "Log-verosimiglianza"
)La linea tratteggiata rossa indica la media campionaria, che — come ci aspettiamo — è anche il valore che massimizza la log-verosimiglianza.
43.2.3 Ottimizzazione Numerica
Se volessimo calcolare il valore ottimale di \(\mu\) in modo automatico:
# Funzione negativa da minimizzare
negative_log_likelihood <- function(mu, y, sigma) {
-sum(dnorm(y, mean = mu, sd = sigma, log = TRUE))
}
# Ottimizzazione con limiti
result <- optim(
par = mean(y), # Valore iniziale
fn = negative_log_likelihood, # Funzione da minimizzare
y = y,
sigma = sigma,
method = "L-BFGS-B",
lower = min(mu_range),
upper = max(mu_range)
)
mu_optimal <- result$par
cat("Il valore ottimale di μ è:", mu_optimal)
#> Il valore ottimale di μ è: 30.93- Abbiamo utilizzato
dnorm(..., log = TRUE)per calcolare in modo semplice e numericamente stabile la log-verosimiglianza. - Il valore di \(\mu\) che massimizza la log-verosimiglianza corrisponde alla media campionaria.
- Questo è un caso in cui la stima di massima verosimiglianza ha una forma chiusa, ma l’approccio numerico resta utile e generalizzabile.
43.3 Riflessioni Conclusive
- Nel caso di una distribuzione normale con \(\sigma\) nota, la miglior stima di \(\mu\) (stima di massima verosimiglianza) è sempre la media delle osservazioni.
- Visualizzare la funzione di verosimiglianza aiuta a capire la plausibilità relativa dei diversi valori di \(\mu\).
- La log-verosimiglianza semplifica e rende più stabili i calcoli numerici.
Esercizi
Informazioni sull’Ambiente di Sviluppo
sessionInfo()
#> R version 4.5.0 (2025-04-11)
#> Platform: aarch64-apple-darwin20
#> Running under: macOS Sequoia 15.5
#>
#> Matrix products: default
#> BLAS: /Library/Frameworks/R.framework/Versions/4.5-arm64/Resources/lib/libRblas.0.dylib
#> LAPACK: /Library/Frameworks/R.framework/Versions/4.5-arm64/Resources/lib/libRlapack.dylib; LAPACK version 3.12.1
#>
#> locale:
#> [1] C/UTF-8/C/C/C/C
#>
#> time zone: Europe/Rome
#> tzcode source: internal
#>
#> attached base packages:
#> [1] stats graphics grDevices utils datasets methods base
#>
#> other attached packages:
#> [1] thematic_0.1.6 MetBrewer_0.2.0 ggokabeito_0.1.0 see_0.11.0
#> [5] gridExtra_2.3 patchwork_1.3.0 bayesplot_1.12.0 psych_2.5.3
#> [9] scales_1.4.0 markdown_2.0 knitr_1.50 lubridate_1.9.4
#> [13] forcats_1.0.0 stringr_1.5.1 dplyr_1.1.4 purrr_1.0.4
#> [17] readr_2.1.5 tidyr_1.3.1 tibble_3.2.1 ggplot2_3.5.2
#> [21] tidyverse_2.0.0 rio_1.2.3 here_1.0.1
#>
#> loaded via a namespace (and not attached):
#> [1] generics_0.1.4 stringi_1.8.7 lattice_0.22-7
#> [4] hms_1.1.3 digest_0.6.37 magrittr_2.0.3
#> [7] evaluate_1.0.3 grid_4.5.0 timechange_0.3.0
#> [10] RColorBrewer_1.1-3 fastmap_1.2.0 rprojroot_2.0.4
#> [13] jsonlite_2.0.0 mnormt_2.1.1 cli_3.6.5
#> [16] rlang_1.1.6 withr_3.0.2 tools_4.5.0
#> [19] parallel_4.5.0 tzdb_0.5.0 pacman_0.5.1
#> [22] vctrs_0.6.5 R6_2.6.1 lifecycle_1.0.4
#> [25] htmlwidgets_1.6.4 pkgconfig_2.0.3 pillar_1.10.2
#> [28] gtable_0.3.6 glue_1.8.0 xfun_0.52
#> [31] tidyselect_1.2.1 rstudioapi_0.17.1 farver_2.1.2
#> [34] htmltools_0.5.8.1 nlme_3.1-168 labeling_0.4.3
#> [37] rmarkdown_2.29 compiler_4.5.0