here::here("code", "_common.R") |>
source()
# Load packages
if (!requireNamespace("pacman")) install.packages("pacman")
pacman::p_load(readr)18 Analisi delle componenti principali
- eseguire la PCA usando l’algebra lineare;
- eseguire la PCA usando R.
- Leggere il capitolo Factor Analysis and Principal Component Analysis del testo di Petersen (2024).
18.1 Introduzione
L’Analisi delle Componenti Principali (PCA) è una tecnica statistica utilizzata per ridurre la dimensionalità di un insieme di dati. Il suo obiettivo è quello di semplificare analisi complesse conservando quanta più informazione possibile, cioè spiegando la maggior parte della varianza presente nei dati originali.
La PCA trasforma un insieme di variabili iniziali, spesso correlate tra loro, in un nuovo insieme di variabili non correlate dette componenti principali. Queste componenti sono combinazioni lineari delle variabili originali e sono ordinate in base alla quantità di varianza che riescono a spiegare.
18.2 Perché Usare la PCA in Psicologia?
In psicologia, ci troviamo spesso a lavorare con dati ad alta dimensionalità: questionari con decine di item, batterie di test, o set di dati raccolti tramite studi longitudinali. La PCA è utile perché:
- semplifica l’interpretazione dei dati, riducendo molte variabili a poche dimensioni latenti;
- elimina ridondanze: se due o più variabili sono altamente correlate, la PCA può rappresentarle con un’unica componente;
- favorisce la visualizzazione, soprattutto nei casi in cui si riescano a ridurre i dati a 2 o 3 componenti principali;
- prepara i dati per analisi successive come regressioni o modelli strutturali, evitando collinearità tra predittori.
🧩 Esempio tipico: un questionario su tratti di personalità con 50 item può essere ridotto a 5 componenti principali che riflettono le dimensioni dei Big Five.
18.3 Cos’è la Varianza Totale?
La varianza totale rappresenta la somma della variabilità presente in ciascuna variabile del dataset.
Se abbiamo tre variabili, la varianza totale sarà:
\[ \text{Varianza Totale} = \sigma_1^2 + \sigma_2^2 + \sigma_3^2 , \]
dove \(\sigma_i^2\) è la varianza della variabile \(i\)-esima.
Nella PCA:
- ogni autovalore (o eigenvalue) rappresenta la quantità di varianza spiegata da una componente principale;
- la somma degli autovalori è pari alla varianza totale del dataset (dopo la standardizzazione, essa sarà uguale al numero di variabili).
18.4 Un Nuovo Sistema di Coordinate
La PCA può essere intesa come una rotazione del sistema di riferimento nello spazio delle variabili:
- la prima componente principale (PC1) è la direzione lungo cui la varianza dei dati è massima;
- la seconda componente (PC2) è perpendicolare alla prima (cioè ortogonale) e spiega la massima varianza residua;
- le successive componenti seguono lo stesso principio.
Questo nuovo sistema è costruito in modo tale che le componenti siano non correlate tra loro (ortogonali) e spiegano, progressivamente, meno varianza.
💡 Nota importante: la PCA non elimina variabili, ma le riorganizza in modo da ridurre la complessità informativa.
18.5 Riduzione della Dimensionalità
La PCA consente di rappresentare i dati originali in un nuovo spazio, più compatto, ma informativamente ricco.
18.5.1 Cosa significa “ridurre la dimensionalità”?
- Riduzione: da p variabili iniziali (es. 30 item), possiamo ottenere k componenti principali (es. 3 o 5), dove k < p;
- Obiettivo: mantenere una soglia prefissata di varianza spiegata, ad esempio il 70% o l’80%.
🎯 Esempio concreto: In uno studio sui Big Five, la PCA può ridurre un set di 100 item a 5 componenti, ciascuna interpretabile come una delle dimensioni di personalità.
18.6 Interpretazione dei Risultati della PCA
La PCA produce due insiemi principali di risultati:
Punteggi delle componenti principali (scores)
Per ogni partecipante (o unità osservata), si ottiene un punteggio per ciascuna componente. Questi punteggi possono essere utilizzati come nuove variabili sintetiche.Varianza spiegata (eigenvalues)
L’importanza di ciascuna componente è misurata dalla proporzione di varianza che essa spiega:
\[ \text{Proporzione di varianza spiegata dalla PC}_i = \frac{\lambda_i}{\sum_{j=1}^{p} \lambda_j} , \]
dove \(\lambda_i\) è l’autovalore della componente i.
🔍 Interpretazione tipica: Se la PC1 spiega il 50% della varianza e la PC2 un ulteriore 30%, possiamo dire che le prime due componenti spiegano l’80% della variabilità totale.
18.7 Geometria della PCA: Assi, Autovalori e Autovettori
La PCA si basa su concetti fondamentali dell’algebra lineare:
- autovalori (eigenvalues): quantificano la varianza spiegata da ogni componente;
- autovettori (eigenvectors): definiscono le direzioni lungo cui si osserva la varianza massima nei dati.
Nel piano bidimensionale:
- ogni autovettore è un asse di una nuova base ortogonale;
- la lunghezza dell’asse è proporzionale alla radice quadrata dell’autovalore corrispondente;
- le osservazioni vengono proiettate ortogonalmente su questi assi per ottenere i punteggi delle componenti principali.
18.8 Visualizzazione: Scree Plot e Biplot
18.8.1 Scree Plot
Lo Scree Plot è un grafico che mostra gli autovalori ordinati per componente:
- permette di determinare quante componenti mantenere;
- un “gomito” nel grafico indica il punto oltre il quale le componenti aggiuntive spiegano poca varianza.
18.8.2 Biplot
Il biplot mostra simultaneamente:
- i punteggi delle osservazioni (punti);
- i contributi delle variabili originali (frecce).
🎓 Guida all’interpretazione del biplot:
- Correlazione positiva: variabili con frecce vicine tra loro (angolo piccolo tra i vettori);
- Correlazione negativa: variabili con frecce dirette in versi opposti (angolo ≈ 180°);
- Nessuna correlazione: variabili con frecce quasi perpendicolari (angolo ≈ 90°).Ogni angolo riflette l’intensità della relazione tra le variabili analizzate.
18.9 Tutorial in R
18.9.1 Obiettivo del tutorial
Applicare la PCA passo dopo passo su un dataset simulato con due variabili correlate, comprendendo ogni fase del processo:
- creazione dei dati e visualizzazione;
- standardizzazione;
- calcolo della PCA con algebra lineare;
- interpretazione geometrica (autovalori, autovettori);
- visualizzazioni: ellissi, componenti, scree plot;
- proiezione dei dati nello spazio delle componenti;
- verifica con
prcomp(); - costruzione del biplot.
18.9.2 Passo 1: Creare un dataset
Generiamo un dataset con due variabili correlate.
# Generiamo due variabili correlate
set.seed(123)
X <- data.frame(
x1 = rnorm(100, mean = 5, sd = 2),
x2 = rnorm(100, mean = 10, sd = 3)
)
# Aggiungiamo una correlazione lineare tra x1 e x2
X$x2 <- 0.8 * X$x1 + rnorm(100, mean = 0, sd = 1)18.9.3 Passo 2: Standardizzare i dati
La standardizzazione è essenziale nella PCA quando le variabili hanno scale diverse. Dopo la standardizzazione, ogni variabile ha media = 0 e deviazione standard = 1.
# Standardizziamo le variabili
X_scaled <- scale(X)Visualizziamo i dati standardizzati:
ggplot(X_scaled, aes(x = x1, y = x2)) +
geom_point(shape = 19) +
coord_fixed(ratio = 1) + # Imposta l'aspect ratio a 1
labs(
x = "x1 standardizzata",
y = "x2 standardizzata",
title = "Dati standardizzati (asp = 1)"
)
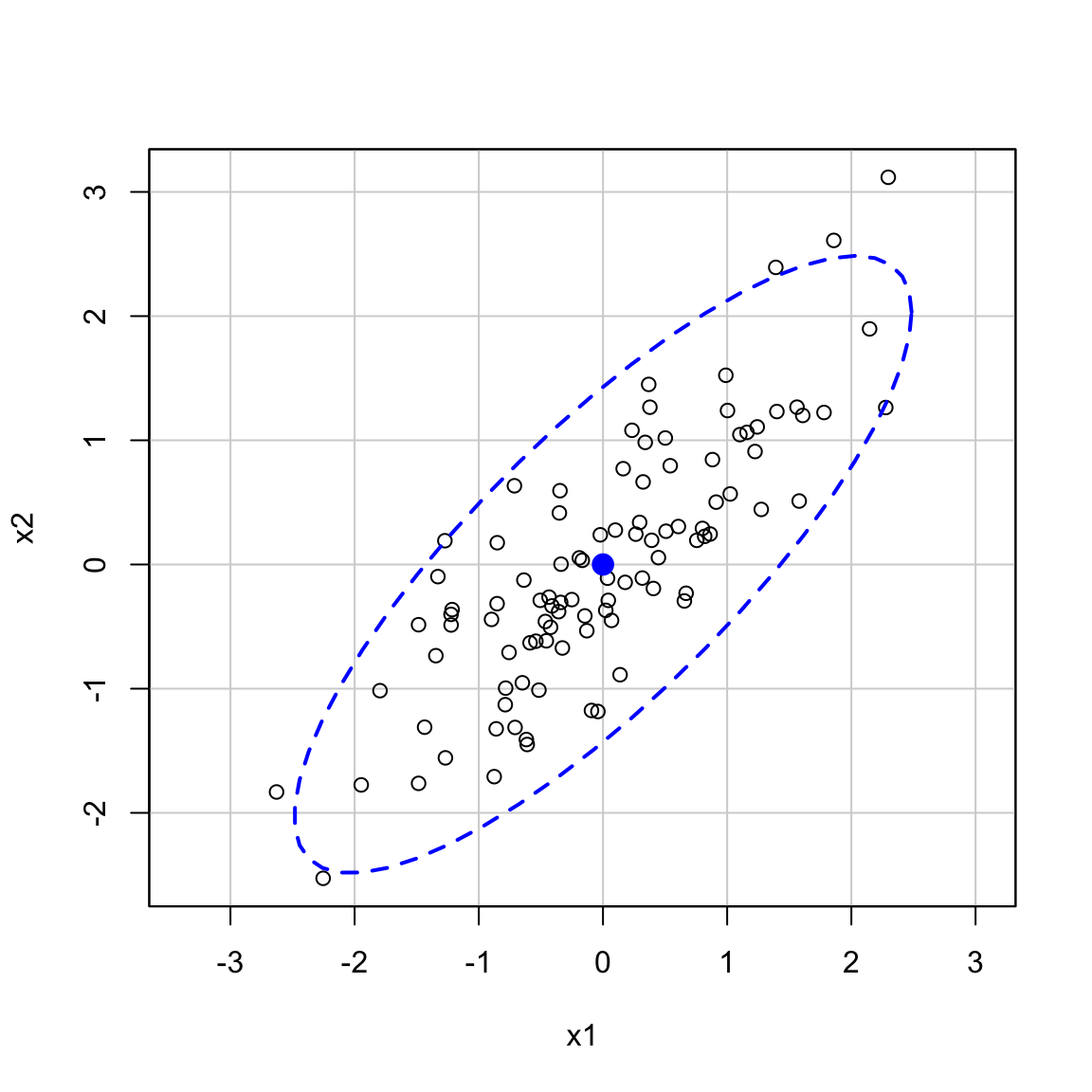
Aggiungiamo un’ellisse di confidenza per mostrare la distribuzione:
car::dataEllipse(
X_scaled[, 1], X_scaled[, 2],
levels = 0.95, lty = 2,
asp = 1,
xlab = "x1", ylab = "x2"
)
18.9.4 Passo 3: Calcolare la matrice di covarianza
cov_matrix <- cov(X_scaled)
cov_matrix
#> x1 x2
#> x1 1.0000 0.8177
#> x2 0.8177 1.000018.9.5 Passo 4: Calcolare autovalori e autovettori
eigen_decomp <- eigen(cov_matrix)
eigenvalues <- eigen_decomp$values # Varianza spiegata (autovalori)
eigenvectors <- eigen_decomp$vectors # Direzioni principali (autovettori)Stampiamo i risultati:
eigenvalues
#> [1] 1.8177 0.1823eigenvectors
#> [,1] [,2]
#> [1,] 0.7071 -0.7071
#> [2,] 0.7071 0.7071Verifichiamo che gli autovettori siano ortogonali (prodotto scalare = 0):
18.9.6 Visualizzare le direzioni principali
Calcoliamo le pendenze degli autovettori:
ev1_slope <- eigenvectors[2, 1] / eigenvectors[1, 1]
ev2_slope <- eigenvectors[2, 2] / eigenvectors[1, 2]Visualizziamo il grafico con gli autovettori sovrapposti:
data.frame(zx = X_scaled[, 1], zy = X_scaled[, 2]) |>
ggplot(aes(x = zx, y = zy)) +
geom_point(size = 2) +
geom_vline(xintercept = 0, linewidth = .5) +
geom_hline(yintercept = 0, linewidth = .5) +
geom_abline(slope = ev1_slope, color = "red", linewidth = 1.2) +
geom_abline(slope = ev2_slope, color = "blue", linewidth = 1.2) +
ggtitle("Autovettori: direzioni delle componenti principali") 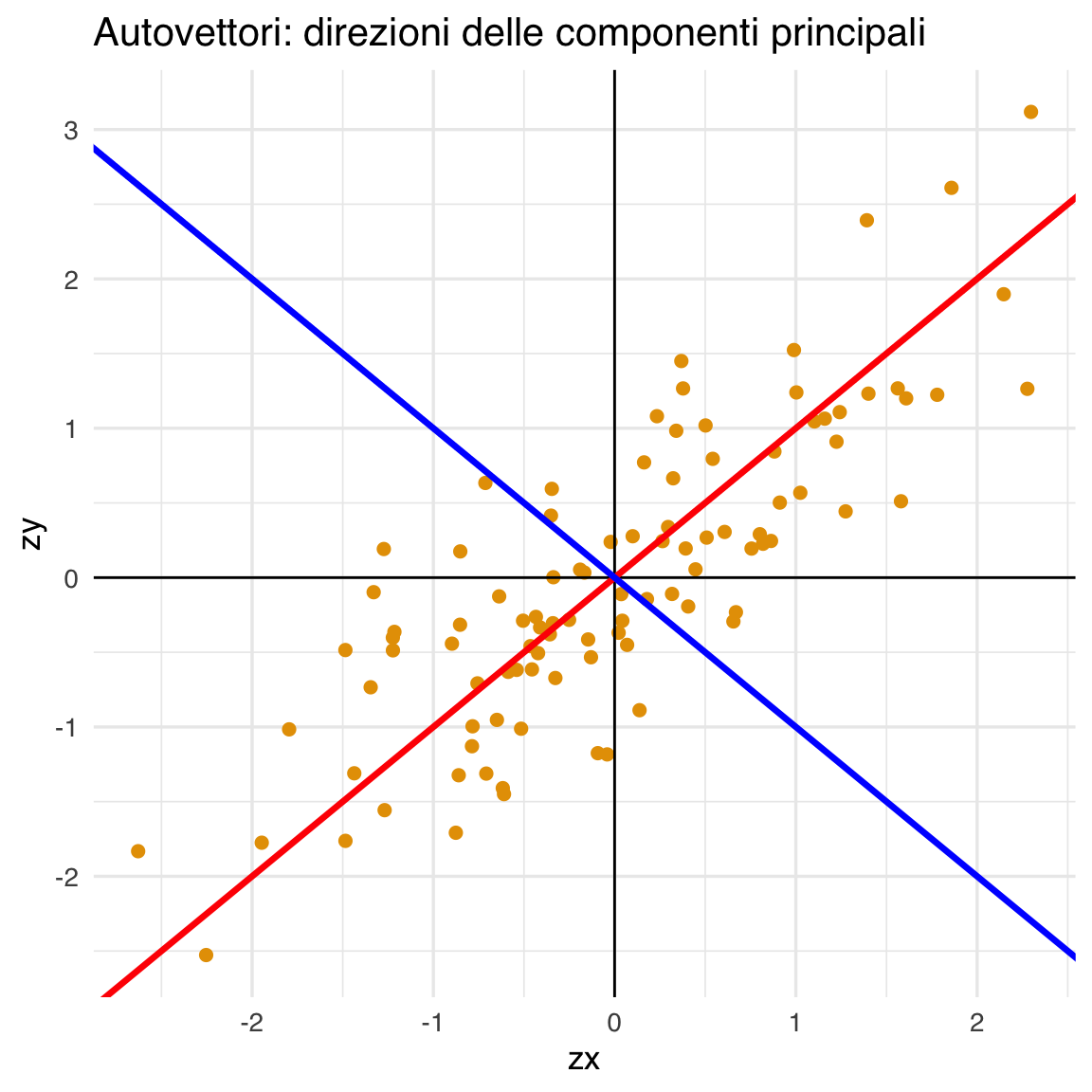
18.9.7 Scree Plot – Percentuale di varianza spiegata
# Percentuale di varianza spiegata
var_per <- tibble(
PC = c("PC1", "PC2"),
Percent = eigenvalues / sum(eigenvalues) * 100
)
ggplot(var_per, aes(x = PC, y = Percent)) +
geom_col(fill = "skyblue", color = "black", width = 0.5) +
ylab("Varianza spiegata (%)") +
xlab("Componente Principale") +
ggtitle("Scree Plot") 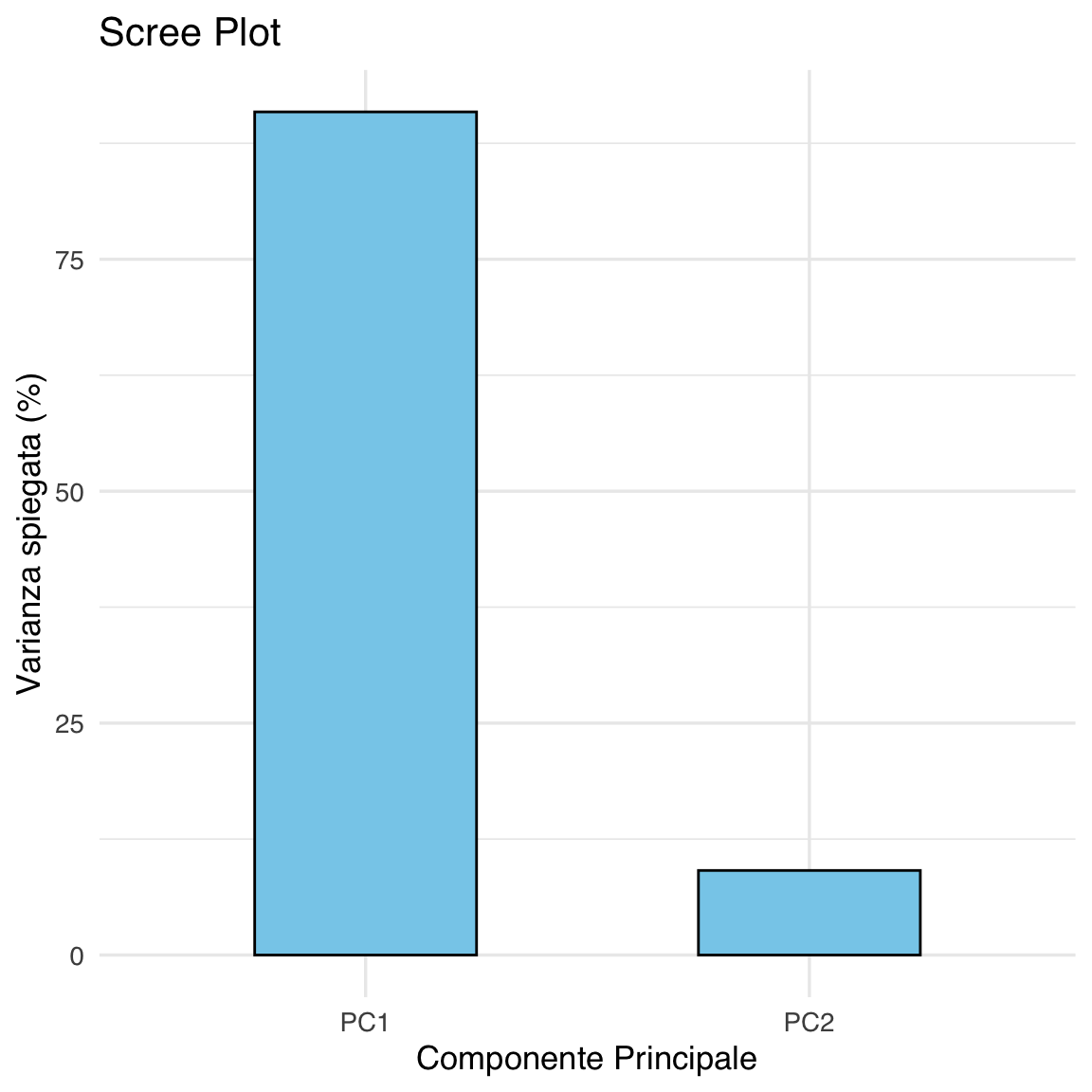
Verifica: somma degli autovalori ≈ somma delle varianze
18.9.8 Passo 5: Visualizzazione geometrica (ellisse + assi principali)
car::dataEllipse(
X_scaled[, 1], X_scaled[, 2],
levels = 0.95, lty = 2,
xlim = c(-3, 3), ylim = c(-3, 3),
asp = 1,
xlab = "x1", ylab = "x2"
)
# Disegniamo gli assi in base agli autovettori
k <- 2.5
arrows(0, 0,
k * sqrt(eigenvalues[1]) * eigenvectors[1, 1],
k * sqrt(eigenvalues[1]) * eigenvectors[2, 1],
col = "red", lwd = 2, code = 2)
arrows(0, 0,
k * sqrt(eigenvalues[2]) * eigenvectors[1, 2],
k * sqrt(eigenvalues[2]) * eigenvectors[2, 2],
col = "red", lwd = 2, code = 2)
18.9.9 Passo 6: Proiezione dei dati (calcolo dei punteggi)
pc_scores <- as.matrix(X_scaled) %*% eigenvectors
colnames(pc_scores) <- c("PC1", "PC2")
head(pc_scores) # Mostra le prime osservazioni nel nuovo spazio
#> PC1 PC2
#> [1,] -0.05606 0.9523
#> [2,] 0.04512 0.5418
#> [3,] 1.98607 -0.2887
#> [4,] 0.15352 0.1844
#> [5,] -0.17413 -0.2344
#> [6,] 2.12408 -0.3930Visualizziamo le osservazioni nello spazio delle componenti:
pc_df <- as.data.frame(pc_scores)
# Grafico ggplot dei punteggi delle componenti principali
ggplot(pc_df, aes(x = PC1, y = PC2)) +
geom_point(color = "blue", size = 2) +
labs(
title = "Punteggi delle Componenti Principali",
x = "PC1",
y = "PC2"
) +
coord_fixed()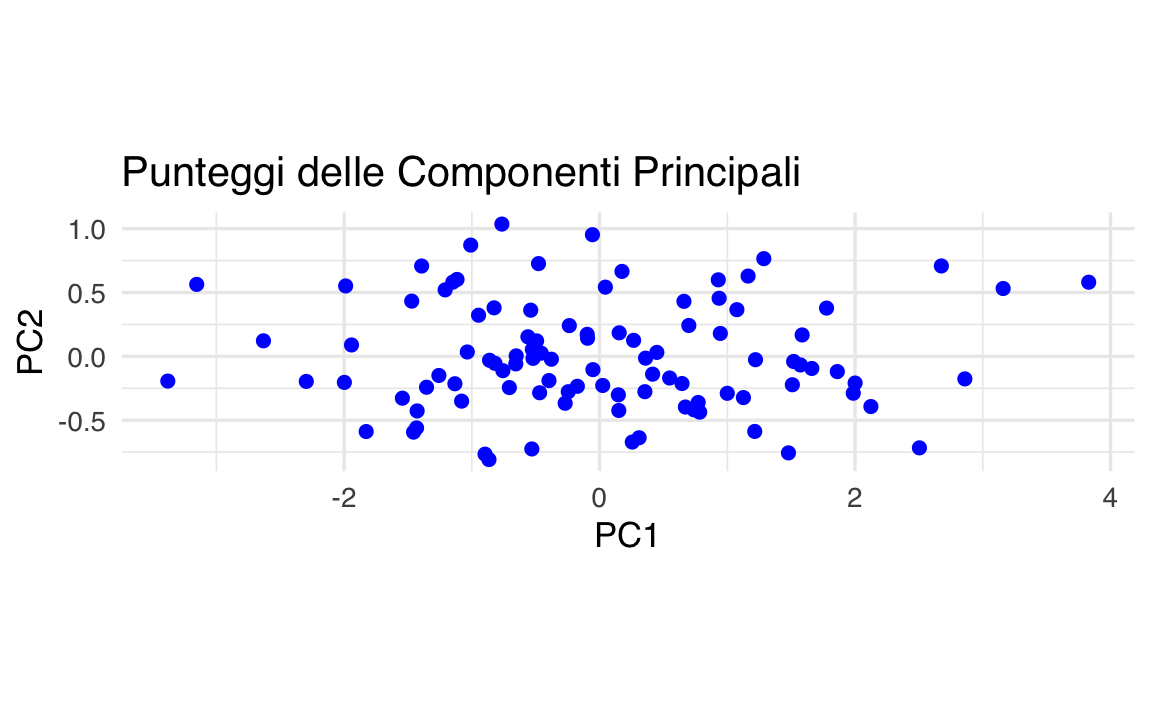
18.9.10 Passo 7: Confronto con prcomp
Verifica: confronta punteggi calcolati a mano con quelli di prcomp
head(pca_auto$x) # Punteggi calcolati da prcomp
#> PC1 PC2
#> [1,] -0.05606 -0.9523
#> [2,] 0.04512 -0.5418
#> [3,] 1.98607 0.2887
#> [4,] 0.15352 -0.1844
#> [5,] -0.17413 0.2344
#> [6,] 2.12408 0.3930head(pc_scores) # Punteggi calcolati a mano
#> PC1 PC2
#> [1,] -0.05606 0.9523
#> [2,] 0.04512 0.5418
#> [3,] 1.98607 -0.2887
#> [4,] 0.15352 0.1844
#> [5,] -0.17413 -0.2344
#> [6,] 2.12408 -0.393018.9.11 Passo 8: Biplot
Il biplot permette di visualizzare contemporaneamente:
- le osservazioni (rappresentate da numeri), proiettate nello spazio delle componenti principali;
- le variabili originali (
x1,x2), rappresentate come frecce rosse, che indicano come ciascuna variabile contribuisce alla definizione delle componenti.
biplot(pca_auto, scale = 0,
main = "Biplot delle Componenti Principali",
xlab = "PC1", ylab = "PC2")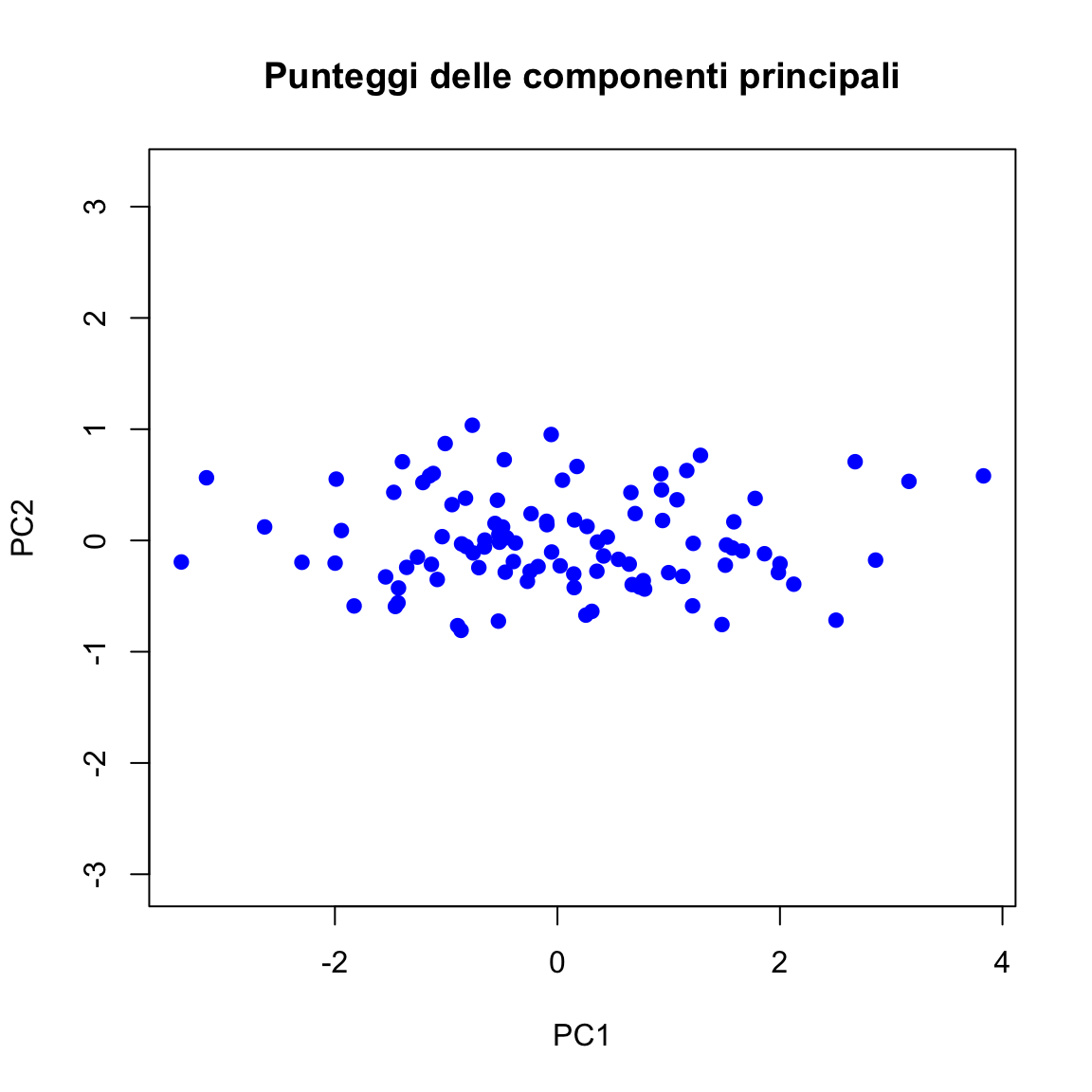
Distribuzione delle osservazioni
- I numeri neri rappresentano le 100 osservazioni.
- Le osservazioni si distribuiscono principalmente lungo la direzione della prima componente principale (PC1), che si estende orizzontalmente.
- C’è relativamente poca variabilità lungo la seconda componente (PC2), che è verticale. Questo conferma che quasi tutta la varianza è catturata da PC1, come osservato nei passaggi precedenti (Scree Plot, autovalori).
Frecce delle variabili originali
Nel biplot vediamo due frecce:
- la freccia x1 punta verso l’alto a destra;
- la freccia x2 punta verso il basso a destra;
- entrambe le frecce sono allineate in parte con l’asse orizzontale (PC1), ma puntano in direzioni opposte lungo PC2.
Interpretazione geometrica:
- Entrambe le variabili contribuiscono positivamente a PC1, perché le componenti orizzontali delle frecce sono entrambe > 0.
- La componente verticale di x1 è positiva, quella di x2 è negativa, quindi le due variabili sono positivamente correlate in generale, ma divergono leggermente lungo PC2.
Questo pattern è coerente con i coefficienti degli autovettori:
pca_auto$rotationEs.:
PC1 PC2
x1 0.71 0.71
x2 0.71 -0.71Cosa significa?
- La PC1 è la somma bilanciata di
x1ex2, e rappresenta la dimensione comune tra le due variabili. - La PC2 è la loro differenza: rappresenta una direzione lungo cui
x1ex2si muovono in modo opposto. Ma in questo caso, la varianza lungo PC2 è molto piccola → questo secondo asse è poco informativo.
Lunghezza delle frecce:
- le frecce hanno lunghezza simile → entrambe le variabili sono ben rappresentate dallo spazio PC1–PC2;
- in un biplot, la lunghezza di una freccia indica quanto bene quella variabile è spiegata dalle componenti principali.
Conclusioni sull’interpretazione del biplot.
- Il biplot mostra che la struttura dei dati può essere riassunta da una sola dimensione latente (PC1).
- Le due variabili
x1ex2sono entrambe fortemente associate a PC1, e debolmente differenziate da PC2.
18.10 Riflessioni Conclusive
Abbiamo completato un’analisi PCA manuale e automatica, verificando ogni passaggio attraverso:
- algebra lineare (autovalori/autovettori);
- visualizzazione geometrica (ellisse, autovettori);
- scree plot;
- proiezione dei dati;
- biplot.
Questa struttura permette di collegare i concetti teorici alla loro implementazione pratica, fornendo agli studenti una comprensione più profonda della PCA.
18.11 ✍️ Suggerimenti per gli Studenti
- Quando standardizzare? Sempre, se le variabili sono su scale diverse (es. punteggi da 0 a 10, da 1 a 100);
- Quando usare la PCA? Quando l’obiettivo è descrittivo, esplorare la struttura latente o semplificare l’analisi;
- Quando non usarla? Se le variabili non sono correlate: la PCA non sarà utile;
- Come interpretare le componenti? Serve analizzare i coefficienti degli autovettori per capire il contributo delle variabili originali a ciascuna componente.
Informazioni sull’Ambiente di Sviluppo
sessionInfo()
#> R version 4.4.2 (2024-10-31)
#> Platform: aarch64-apple-darwin20
#> Running under: macOS Sequoia 15.3.2
#>
#> Matrix products: default
#> BLAS: /Library/Frameworks/R.framework/Versions/4.4-arm64/Resources/lib/libRblas.0.dylib
#> LAPACK: /Library/Frameworks/R.framework/Versions/4.4-arm64/Resources/lib/libRlapack.dylib; LAPACK version 3.12.0
#>
#> locale:
#> [1] C/UTF-8/C/C/C/C
#>
#> time zone: Europe/Rome
#> tzcode source: internal
#>
#> attached base packages:
#> [1] stats graphics grDevices utils datasets methods base
#>
#> other attached packages:
#> [1] ggokabeito_0.1.0 see_0.11.0 MASS_7.3-65 viridis_0.6.5
#> [5] viridisLite_0.4.2 ggpubr_0.6.0 ggExtra_0.10.1 gridExtra_2.3
#> [9] patchwork_1.3.0 bayesplot_1.11.1 semTools_0.5-6 semPlot_1.1.6
#> [13] lavaan_0.6-19 psych_2.4.12 scales_1.3.0 markdown_1.13
#> [17] knitr_1.50 lubridate_1.9.4 forcats_1.0.0 stringr_1.5.1
#> [21] dplyr_1.1.4 purrr_1.0.4 readr_2.1.5 tidyr_1.3.1
#> [25] tibble_3.2.1 ggplot2_3.5.1 tidyverse_2.0.0 here_1.0.1
#>
#> loaded via a namespace (and not attached):
#> [1] rstudioapi_0.17.1 jsonlite_1.9.1 magrittr_2.0.3
#> [4] TH.data_1.1-3 estimability_1.5.1 farver_2.1.2
#> [7] nloptr_2.2.1 rmarkdown_2.29 vctrs_0.6.5
#> [10] minqa_1.2.8 base64enc_0.1-3 rstatix_0.7.2
#> [13] htmltools_0.5.8.1 broom_1.0.7 Formula_1.2-5
#> [16] htmlwidgets_1.6.4 plyr_1.8.9 sandwich_3.1-1
#> [19] emmeans_1.10.7 zoo_1.8-13 igraph_2.1.4
#> [22] mime_0.13 lifecycle_1.0.4 pkgconfig_2.0.3
#> [25] Matrix_1.7-3 R6_2.6.1 fastmap_1.2.0
#> [28] rbibutils_2.3 shiny_1.10.0 digest_0.6.37
#> [31] OpenMx_2.21.13 fdrtool_1.2.18 colorspace_2.1-1
#> [34] rprojroot_2.0.4 Hmisc_5.2-3 labeling_0.4.3
#> [37] timechange_0.3.0 abind_1.4-8 compiler_4.4.2
#> [40] withr_3.0.2 glasso_1.11 htmlTable_2.4.3
#> [43] backports_1.5.0 carData_3.0-5 ggsignif_0.6.4
#> [46] corpcor_1.6.10 gtools_3.9.5 tools_4.4.2
#> [49] pbivnorm_0.6.0 foreign_0.8-88 zip_2.3.2
#> [52] httpuv_1.6.15 nnet_7.3-20 glue_1.8.0
#> [55] quadprog_1.5-8 nlme_3.1-167 promises_1.3.2
#> [58] lisrelToR_0.3 grid_4.4.2 checkmate_2.3.2
#> [61] cluster_2.1.8.1 reshape2_1.4.4 generics_0.1.3
#> [64] gtable_0.3.6 tzdb_0.5.0 data.table_1.17.0
#> [67] hms_1.1.3 car_3.1-3 sem_3.1-16
#> [70] pillar_1.10.1 rockchalk_1.8.157 later_1.4.1
#> [73] splines_4.4.2 lattice_0.22-6 survival_3.8-3
#> [76] kutils_1.73 tidyselect_1.2.1 miniUI_0.1.1.1
#> [79] pbapply_1.7-2 reformulas_0.4.0 stats4_4.4.2
#> [82] xfun_0.51 qgraph_1.9.8 arm_1.14-4
#> [85] stringi_1.8.4 yaml_2.3.10 pacman_0.5.1
#> [88] boot_1.3-31 evaluate_1.0.3 codetools_0.2-20
#> [91] mi_1.1 cli_3.6.4 RcppParallel_5.1.10
#> [94] rpart_4.1.24 xtable_1.8-4 Rdpack_2.6.3
#> [97] munsell_0.5.1 Rcpp_1.0.14 coda_0.19-4.1
#> [100] png_0.1-8 XML_3.99-0.18 parallel_4.4.2
#> [103] jpeg_0.1-10 lme4_1.1-36 mvtnorm_1.3-3
#> [106] openxlsx_4.2.8 rlang_1.1.5 multcomp_1.4-28
#> [109] mnormt_2.1.1