Capitolo 8 Manipolazione dei dati
8.1 Motivazione
Si chiamano “dati grezzi” quelli che provengono dal mondo circostanze, i
dati raccolti per mezzo degli strumenti usati negli esperimenti, per
mezzo di interviste, di questionari, ecc. Questi dati (chiamati
dataset) raramente vengono forniti con una struttura logica precisa.
Per potere elaborarli mediante dei software dobbiamo prima trasformarli
in maniera tale che abbiano una struttura logica organizzata. La
struttura che solitamente si utilizza è quella tabellare (matrice dei
dati), ovvero si dispongono i dati in una tabella nella quale a ciascuna
riga corrisponde ad un’osservazione e ciascuna colonna corrisponde ad
una variabile rilevata. In R una tale struttura è chiamata data frame.
Il pacchetto dplyr, che è al momento uno dei pacchetti più famosi e
utilizzati per la gestione dei dati, offre una serie di funzionalità che
consentono di ottenere il risultato descritto in precedenza e consente
inoltre di eseguire le operazioni più comuni di manipolazione dei dati
in maniera più semplice rispetto a quanto succeda quando usiamo le
funzioni base di R.
8.2 Trattamento dei dati con dplyr
Il pacchetto dplyr include cinque funzioni base: filter(),
select(), mutate(), arrange() e summarise(). A queste cinque
funzioni di base si aggiungono il pipe %>% che serve a concatenare più
operazioni e group_by che serve per il subsetting. In particolare,
considerando una matrice osservazioni per variabili, select() e
mutate() si occupano di organizzare le variabili, filter() e
arrange() i casi, e summarise() i gruppi.
Per introdurre le funzionalità di base di dplyr, considereremo i dati contenuti nel data frame msleep fornito dal pacchetto ggplot2, che elenca le ore di sonno medie per 83 specie di mammiferi (Savage et al., 2007). Carichiamo il pacchetto tidyverse (che contiene ggplot2) e leggiamo nella memoria di lavoro l’oggetto msleep:
library("tidyverse")
data(msleep)
dim(msleep)
#> [1] 83 118.2.1 Operatore pipe
Prima di presentare le funzionalità di tidyverse, introduciamo l’operatore pipe %>% del pacchetto magrittr – ma ora presente anche in base R nella versione |>. L’operatore pipe, %>% o |>, serve a concatenare varie funzioni insieme, in modo da inserire un’operazione dietro l’altra. Una spiegazione intuitiva dell’operatore pipe è stata fornita in un tweet di @andrewheiss. Consideriamo la seguente istruzione in pseudo-codice R:
leave_house(get_dressed(get_out_of_bed(wake_up(me, time = "8:00"), side = "correct"), pants = TRUE, shirt = TRUE), car = TRUE, bike = FALSE)Il listato precedente descrive una serie di (pseudo) funzioni concatenate, le quali costituiscono gli argomenti di altre funzioni. Scritto così, il codice è molto difficile da capire. Possiamo però ottenere lo stesso risultato utilizzando l’operatore pipe che facilita la leggibilità del codice:
me %>%
wake_up(time = "8:00") %>%
get_out_of_bed(side = "correct") %>%
get_dressed(pants = TRUE, shirt = TRUE) %>%
leave_house(car = TRUE, bike = FALSE)In questa seconda versione del (pseudo) codice R si capisce molto meglio ciò che vogliamo fare. Il data.frame me viene passato alla funzione wake_up(). La funzione wake_up() ha come argomento l’ora del giorno: time = "8:00". Una volta “svegliati” (wake up) dobbiamo scendere dal letto. Quindi l’output di wake_up() viene passato alla funzione get_out_of_bed() la quale ha come argomento side = "correct" perché vogliamo scendere dal letto dalla parte giusta. E così via.
8.2.2 Selezionare le colonne del data.frame con select()
Ritorniamo ora all’esempio precedente e supponiamo di volere selezionare le variabili name, vore e sleep_total dal data.frame msleep. Per fare ciò usiamo funzione select() che consente di selezionare un sottoinsieme di variabili in un dataset. Usando pipe scriviamo:
dt <- msleep %>%
dplyr::select(name, vore, sleep_total)
dt
#> # A tibble: 83 x 3
#> name vore sleep_total
#> <chr> <chr> <dbl>
#> 1 Cheetah carni 12.1
#> 2 Owl monkey omni 17
#> 3 Mountain beaver herbi 14.4
#> 4 Greater short-tailed shrew omni 14.9
#> 5 Cow herbi 4
#> 6 Three-toed sloth herbi 14.4
#> # … with 77 more rowsladdove la sequenza di istruzioni precedenti significa che il data.frame dt è stato passato alla funzione select() contenuta nel pacchetto dplyr.
8.2.3 Filtrare le righe del data.frame con filter()
La funzione filter() consente di selezionare un sottoinsieme di
osservazioni in un dataset. Per esempio, possiamo selezionare tutte le
osservazioni nella variabile vore contrassegnate come carni in
questo modo (ovvero, tutti i carnivori):
dt %>%
dplyr::filter(vore == "carni")
#> # A tibble: 19 x 3
#> name vore sleep_total
#> <chr> <chr> <dbl>
#> 1 Cheetah carni 12.1
#> 2 Northern fur seal carni 8.7
#> 3 Dog carni 10.1
#> 4 Long-nosed armadillo carni 17.4
#> 5 Domestic cat carni 12.5
#> 6 Pilot whale carni 2.7
#> # … with 13 more rows
8.2.4 Aggiungere una colonna al data.frame con mutate()
Talvolta vogliamo creare una nuova variabile in uno stesso dataset ad
esempio sommando o dividendo due variabili, oppure calcolandone la
media. A questo scopo si usa la funzione mutate(). Per esempio, se
vogliamo esprimere i valori di sleep_total in minuti, moltiplichiamo
per 60:
8.2.5 Ordinare i dati con arrange()
La funzione arrange() serve a ordinare i dati in base ai valori di una
o più variabili. Per esempio, possiamo ordinare la variabile
sleep_total dal valore più alto al più basso in questo modo:
dt %>%
arrange(desc(sleep_total))
#> # A tibble: 83 x 3
#> name vore sleep_total
#> <chr> <chr> <dbl>
#> 1 Little brown bat insecti 19.9
#> 2 Big brown bat insecti 19.7
#> 3 Thick-tailed opposum carni 19.4
#> 4 Giant armadillo insecti 18.1
#> 5 North American Opossum omni 18
#> 6 Long-nosed armadillo carni 17.4
#> # … with 77 more rows
8.2.6 Raggruppare i dati con group_by()
La funzione group_by() serve a raggruppare insieme i valori in base a
una o più variabili. La vedremo in uso in seguito insieme a
summarise().
8.2.7 Sommario dei dati con summarise()
La funzione summarise() collassa il dataset in una singola riga dove
viene riportato il risultato della statistica richiesta. Per esempio, la
media del tempo totale del sonno è
8.2.8 Operazioni raggruppate
In precedenza abbiamo visto come i mammiferi considerati dormano, in
media, 10.4 ore al giorno. Troviamo ora il sonno medio in funzione di
vore:
dt %>%
group_by(vore) %>%
summarise(
m_sleep = mean(sleep_total, na.rm = TRUE),
n = n()
)
#> # A tibble: 5 x 3
#> vore m_sleep n
#> <chr> <dbl> <int>
#> 1 carni 10.4 19
#> 2 herbi 9.51 32
#> 3 insecti 14.9 5
#> 4 omni 10.9 20
#> 5 <NA> 10.2 7Si noti che, nel caso di 7 osservazioni, il valore di vore non era
specificato. Per tali osservazioni, dunque, la classe di appartenenza è
NA.
8.3 Dati categoriali in R
Consideriamo una variabile che descrive il genere e include le categorie male, female e non-conforming. In R, ci sono due modi per memorizzare queste informazioni. Uno è usare la classe character strings e l’altro è usare la classe factor. Non ci addentrimo qui nelle sottigliezze di questa distinzione, motivata in gran parte per le necessità della programmazione con le funzioni di tidyverse. Per gli scopi di questo insegnamento sarà sufficiente codificare le variabili qualitative usando la classe factor. Una volta codificati i dati qualitativi utilizzando la classe factor, si pongono spesso due problemi:
- modificare le etichette dei livelli (modalità) di un fattore,
- riordinare i livelli di un fattore.
8.3.1 Modificare le etichette dei livelli di un fattore
Esaminiamo l’esempio seguente.
f_1 <- c("old_3", "old_4", "old_1", "old_1", "old_2")
f_1 <- factor(f_1)
y <- 1:5
df <- data.frame(f_1, y)
df
#> f_1 y
#> 1 old_3 1
#> 2 old_4 2
#> 3 old_1 3
#> 4 old_1 4
#> 5 old_2 5Supponiamo di volere che i livelli del fattore f_1 abbiano le etichette new_1, new_2, ecc. Per ottenere questo risultato usiamo la funzione recode() di dplyr:
8.3.2 Riordinare i livelli di un fattore
Spesso i livelli dei fattori hanno un ordinamento naturale. Tuttavia, l’impostazione predefinita in base R è ordinare i livelli in ordine alfabetico. Quindi, gli utenti devono avere un modo per imporre l’ordine desiderato sulla codifica delle loro variabili qualitative. Ciò può essere ottenuto nel modo seguente.
df$f_1 <- factor(df$f_1,
levels = c(
"new_poco", "new_medio", "new_tanto", "new_massimo"
)
)
summary(df$f_1)
#> new_poco new_medio new_tanto new_massimo
#> 2 1 1 1Per approfondire le problematiche della manipolazione di variabili qualitative in R, si veda (mcnamara2018wrangling?).
8.4 Creare grafici con ggplot2()
Il pacchetto ggplot2() è un potente strumento per rappresentare graficamente i dati. Le iniziali del nome, gg, si riferiscono alla “Grammar of Graphics,” che è un modo di pensare le figure come una serie di layer stratificati. Originariamente descritta da (wilkinson2012grammar?), la grammatica dei grafici è stata aggiornata e applicata in R da Hadley Wickham, il creatore del pacchetto.
La funzione da cui si parte per inizializzare un grafico è ggplot(). La funzione ggplot() richiede due argomenti. Il primo è l’oggetto di tipo data.frame che contiene i dati da visualizzare – in alternativa al primo argomento, un dataframe può essere passato a ggplot() mediante l’operatore pipe. Il secondo è una particolare lista che viene generata dalla funzione aes(), la quale determina l’aspetto (aesthetic) del grafico. La funzione aes() richiede necessariamente di specificare “x” e “y,” ovvero i nomi delle colonne del data.frame che è stato utilizzato quale primo argomento di ggplot() (o che è stato passato da pipe), le quali rappresentano le variabili da porre rispettivamente sugli assi orizzontale e verticale.
La definizione della tipologia di grafico e i vari parametri sono poi definiti successivamente, aggiungendo all’oggetto creato da ggplot() tutte le componenti necessarie. Saranno quindi altre funzioni, come geom_bar(), geom_line() o geom_point() a occuparsi di aggiungere al livello di base barre, linee, punti, e così via. Infine, tramite altre funzioni, ad esempio labs(), sarà possibile definire i dettagli più fini.
Gli elementi grafici (bare, punti, segmenti, …) usati da ggplot2 sono chiamati geoms. Mediante queste funzioni è possibile costruire diverse tipologie di grafici:
geom_bar(): crea un layer con delle barre;geom_point(): crea un layer con dei punti (diagramma a dispersione);geom_line(): crea un layer con una linea retta;geom_histogram(): crea un layer con un istogramma;geom_boxplot(): crea un layer con un box-plot;geom_errorbar(): crea un layer con barre che rappresentano intervalli di confidenza;geom_hline()egeom_vline(): crea un layer con una linea orizzontale o verticale definita dall’utente.
Un comando generico ha la seguente forma:
La prima volta che si usa il pacchetto ggplot2 è necessario installarlo. Per fare questo possiamo installare tidyverse che, oltre a caricare ggplot2, carica anche altre utili funzioni per l’analisi dei dati:
install.packages("tidyverse")Per attivare il pacchetto si usa l’istruzione:
Ogni volta che si inizia una sessione R è necessario attivare i pacchetti che si vogliono usare, ma non è necessario istallarli una nuova volta. Se è necessario specificare il pacchetto nel quale è contenuta la funzione (o il data.frame) che vogliamo utilizzare, usiamo la sintassi package::function(). Per esempio, l’istruzione ggplot2::ggplot() rende esplicito che stiamo usando la funzione ggplot() contenuta nel pacchetto ggplot2.
8.4.1 Diagramma a dispersione
Consideriamo nuovamenti i dati contenuti nel data frame msleep e poniamoci il problema di rappresentare graficamente la relazione tra il numero medio di ore di sonno giornaliero (sleep_total) e il peso dell’animale (bodywt). Usando le impostazioni di default di ggplot2, con le istruzioni seguenti, otteniamo il grafico fornito dalla figura seguente.
data(msleep)
p <- msleep %>%
ggplot(
aes(x = bodywt, y = sleep_total)
) +
geom_point()
print(p)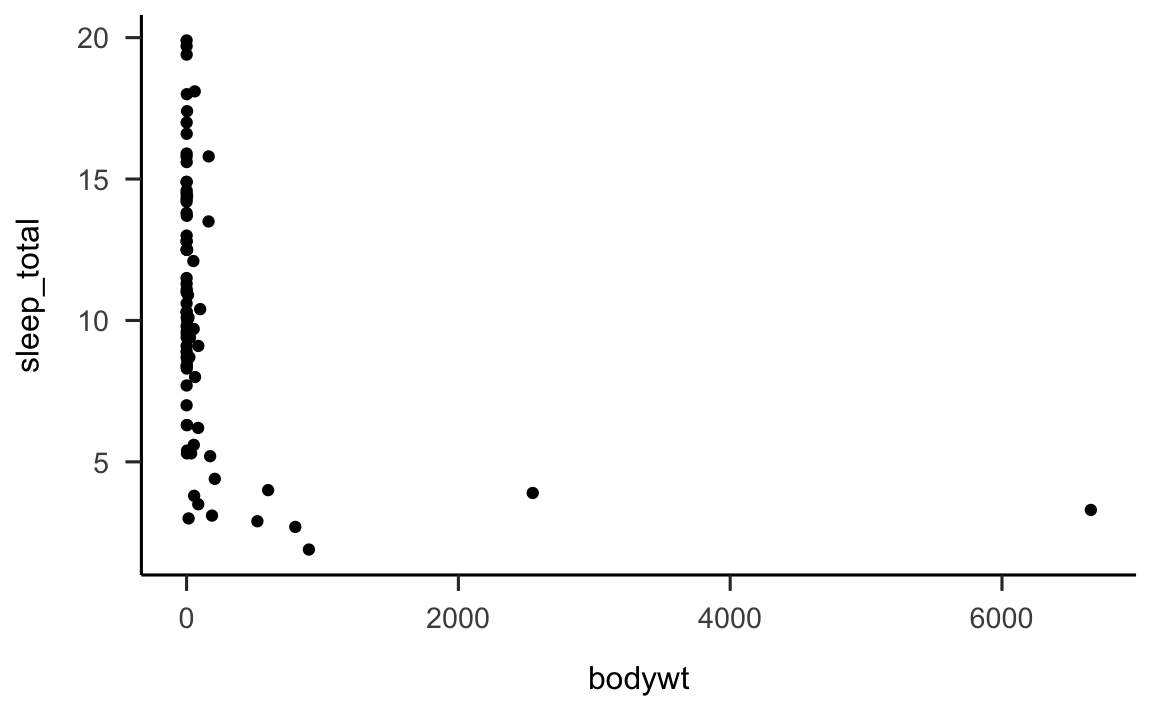
Coloriamo ora in maniera diversa i punti che rappresentano animali carnivori, erbivori, ecc.
p <- msleep %>%
ggplot(
aes(x = bodywt, y = sleep_total, col = vore)
) +
geom_point()
print(p)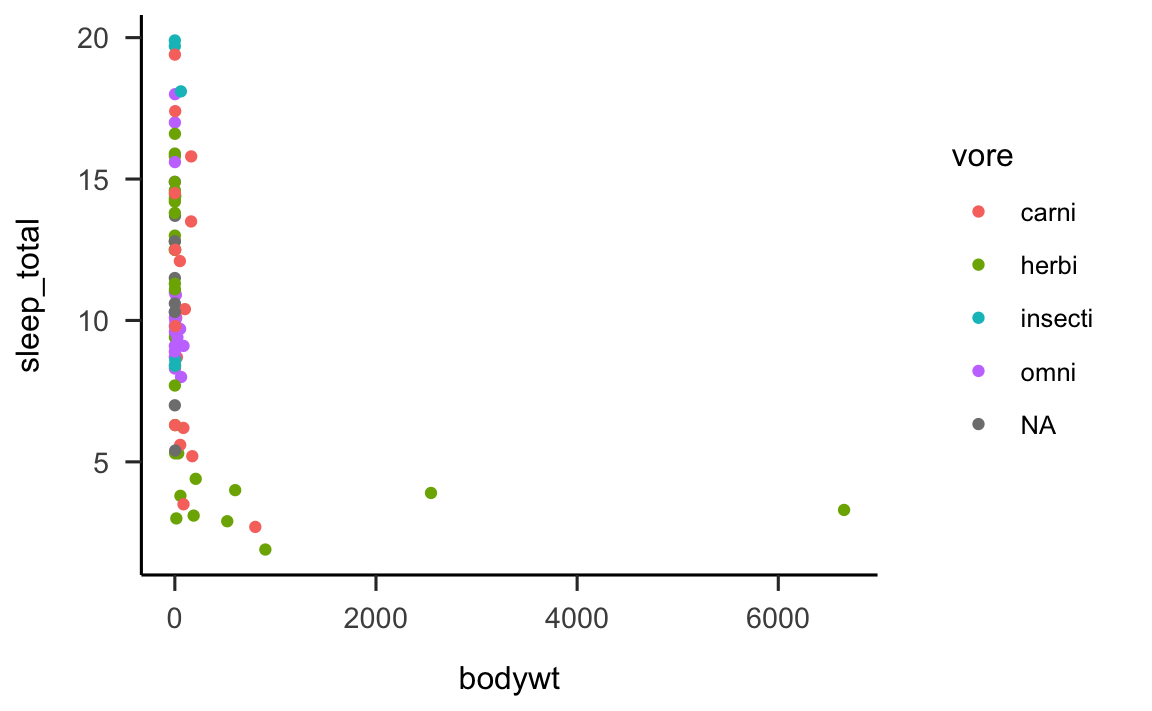
È chiaro, senza fare alcuna analisi statistica, che la relazione tra le due variabili non è lineare. Trasformando in maniera logaritmica i valori dell’asse \(x\) la relazione si linearizza.
p <- msleep %>%
ggplot(
aes(x = log(bodywt), y = sleep_total, col = vore)
) +
geom_point()
print(p)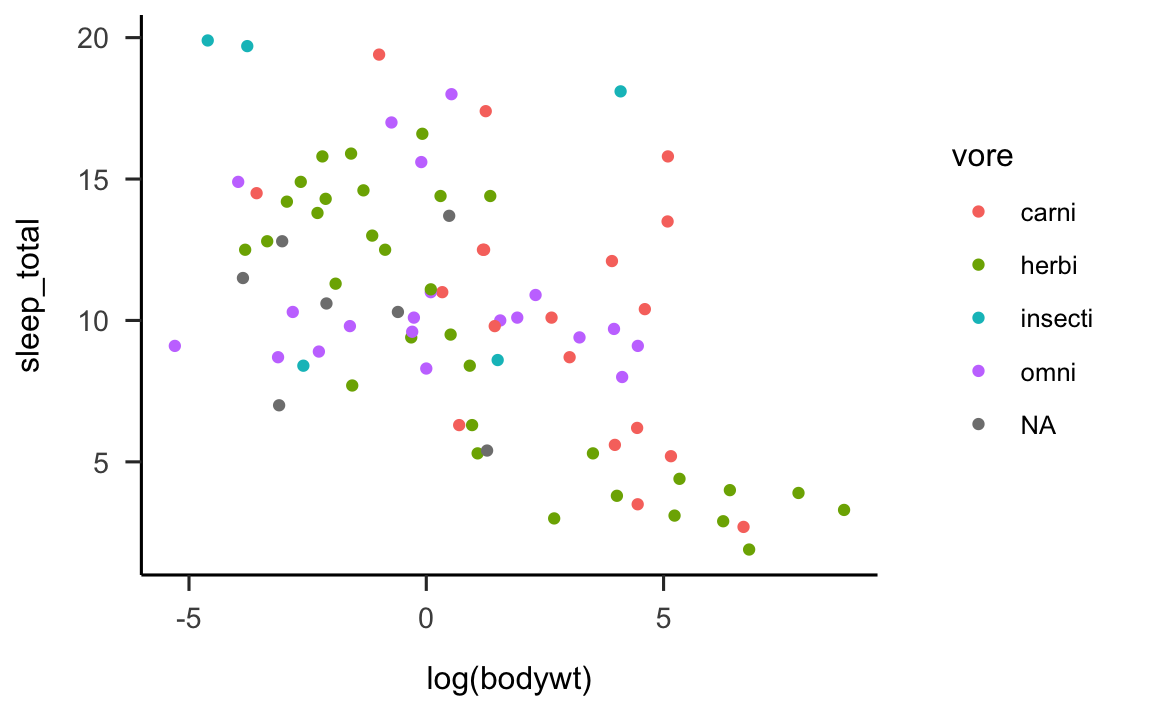
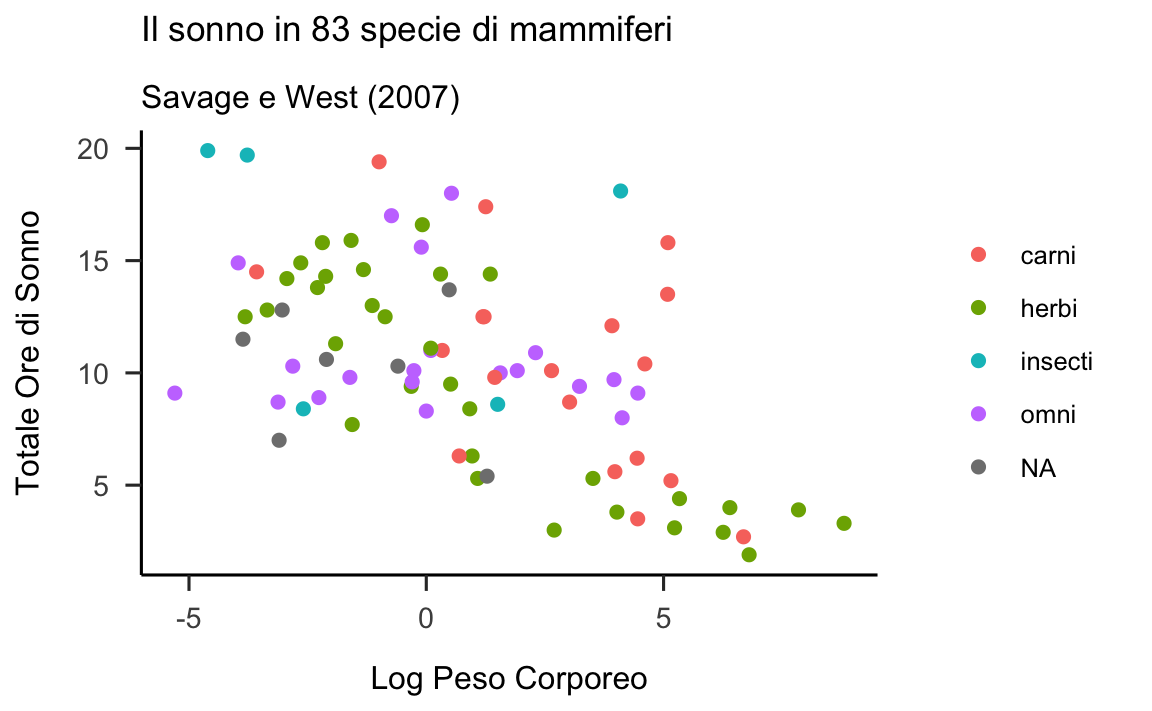
Infine, aggiustiamo il “tema” del grafico, aggiungiamo le etichette sugli assi e il titolo.
msleep %>%
ggplot(
aes(x = log(bodywt), y = sleep_total, col = vore)
) +
geom_point(size = 2) +
theme(legend.title = element_blank()) +
labs(
x = "Log Peso Corporeo",
y = "Totale Ore di Sonno",
title = "Il sonno in 83 specie di mammiferi",
subtitle = "Savage e West (2007)"
)
8.4.2 Istogramma
Creiamo ora un istogramma che rappresenta la distribuzione del (logaritmo del) peso medio del cervello delle 83 specie di mammiferi considerate da (savage2007quantitative?).
msleep %>%
ggplot(
aes(log(brainwt))
) +
geom_histogram(aes(y = ..density..)) +
labs(
x = "Log Peso Cervello",
y = "Frequenza Relativa"
) +
theme(legend.title = element_blank())
#> `stat_bin()` using `bins = 30`. Pick better value with `binwidth`.
#> Warning: Removed 27 rows containing non-finite values (stat_bin).
L’argomento aes(y=..density..) in geom_histogram() produce le frequenze relative. L’opzione di default (senza questo argomento) porta ggplot() a rappresentare le frequenze assolute.
8.4.3 Scrivere il codice in R con stile
Uno stile di programmazione è un insieme di regole per la gestione dell’indentazione dei blocchi di codice, per la creazione dei nomi dei file e delle variabili e per le convenzioni tipografiche che vengono usate. Scrivere il codice in R con stile consente di creare listati più leggibili e semplici da modificare, minimizza la possibilità di errore, e consente correzioni e modifiche più rapide. Vi sono molteplici stili di programmazione che possono essere utilizzati dall’utente, anche se è bene attenersi a quelle che sono le convenzioni maggiormente diffuse, allo scopo di favorire la comunicazione. In ogni caso, l’importante è di essere coerenti, ovvero di adottare le stesse convenzioni in tutte le parti del codice che si scrive. Ad esempio, se si sceglie di usare lo stile snake_case per il nome composto di una variabile (es., personality_trait), non è appropriato usare lo stile lower Camel case per un’altra variabile (es., socialStatus). Dato che questo argomento è stato trattato ampiamente in varie sedi, mi limito qui a rimandare ad uno stile di programmazione molto popolare, quello proposto da Hadley Wickham, il creatore di tidyverse. Potete trovare maggiori informazioni al seguente link: http://style.tidyverse.org/.