68 Introduzione a CmdStanR
Prerequisiti
- Leggere Getting started with CmdStanR.
Concetti e competenze chiave
- Acquisire le competenze necessarie per utilizzare CmdStan con R.
Preparazione del Notebook
68.1 Introduzione
Riprendiamo l’analisi dei dati fittizi di un compito Go/No-go, in cui sono state registrate 6 risposte corrette su 9 prove, già discussa in precedenza. In questa sezione, utilizzeremo il pacchetto cmdstanr in R, invece di cmdstanpy in Python, per eseguire l’analisi. L’obiettivo di questo capitolo è mostrare come utilizzare CmdStan attraverso il linguaggio R, offrendo un’alternativa all’uso di Python.
In R, i dati vengono salvati in una lista, che equivale a un dizionario in Python.
data_list <- list(
"N" = 9,
"y" = 6
)Successivamente, specifichiamo il percorso del file contenente lo script Stan. È importante notare che lo script Stan rimane identico indipendentemente dall’interfaccia utilizzata, sia essa R o Python.
68.2 Compilazione del modello
Per compilare il modello, utilizziamo la funzione cmdstan_model(), che crea un nuovo oggetto CmdStanModel a partire da un file contenente un programma Stan.
mod <- cmdstan_model(file)Dopo aver compilato il modello, possiamo stamparne le informazioni.
mod$print()data {
int<lower=1> N;
int<lower=0> y;
}
parameters {
real<lower=0, upper=1> p;
}
model {
y ~ binomial(N, p); // Likelihood
p ~ beta(1, 1); // Prior
}
generated quantities {
int<lower=0, upper=1> p_gt_chance = p > 0.5;
}68.3 Esecuzione dell’algoritmo MCMC
Il metodo $sample() sugli oggetti CmdStanModel esegue l’algoritmo MCMC predefinito di Stan. L’argomento data accetta una lista di oggetti R con nomi specificati.
fit <- mod$sample(
data = data_list,
seed = 123,
chains = 4,
parallel_chains = 4
)68.4 Statistiche riassuntive del posterior
Il metodo $summary() chiama la funzione summarise_draws() dal pacchetto posterior. Il primo argomento specifica le variabili da riassumere, e gli argomenti successivi sono passati a posterior::summarise_draws() per specificare quali statistiche calcolare, l’uso di più core, ecc.
fit$summary(variables = c("p"))# A tibble: 1 × 10
variable mean median sd mad q5 q95 rhat ess_bulk ess_tail
<chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 p 0.636 0.647 0.141 0.148 0.389 0.854 1.00 1372. 1351.È possibile utilizzare una formula per riassumere funzioni arbitrarie, come ad esempio la probabilità che \(p\) sia minore o uguale a 0.5.
fit$summary("p", pr_lt_half = ~ mean(. <= 0.5))# A tibble: 1 × 2
variable pr_lt_half
<chr> <dbl>
1 p 0.17668.5 Estrazione dei campioni posteriori
68.5.1 Estrazione dei campioni
Il metodo $draws() può essere utilizzato per estrarre i campioni posteriori in formati supportati dal pacchetto posterior. Qui dimostriamo i formati draws_array e draws_df.
# default is a 3-D draws_array object from the posterior package
# iterations x chains x variables
draws_arr <- fit$draws() # or format="array"
str(draws_arr) 'draws_array' num [1:1000, 1:4, 1:3] -7.21 -8.34 -9.16 -7.22 -7.21 ...
- attr(*, "dimnames")=List of 3
..$ iteration: chr [1:1000] "1" "2" "3" "4" ...
..$ chain : chr [1:4] "1" "2" "3" "4"
..$ variable : chr [1:3] "lp__" "p" "p_gt_chance"Oppure, possiamo usare as_draws_df() per creare un data frame:
draws <- as_draws_df(fit)
head(draws)# A draws_df: 6 iterations, 1 chains, and 3 variables
lp__ p p_gt_chance
1 -7.2 0.63 1
2 -8.3 0.41 0
3 -9.2 0.34 0
4 -7.2 0.66 1
5 -7.2 0.63 1
6 -7.2 0.64 1
# ... hidden reserved variables {'.chain', '.iteration', '.draw'}Lo stesso risultato si ottiene nel modo seguente:
draws_df <- as_draws_df(draws_arr)
head(draws_df)# A draws_df: 6 iterations, 1 chains, and 3 variables
lp__ p p_gt_chance
1 -7.2 0.63 1
2 -8.3 0.41 0
3 -9.2 0.34 0
4 -7.2 0.66 1
5 -7.2 0.63 1
6 -7.2 0.64 1
# ... hidden reserved variables {'.chain', '.iteration', '.draw'}Una volta creato un data frame, possiamo facilmente calcolare le statistiche descrittive. Per esempio:
draws_df$p |>
mean()[1] 0.6364032In questo modo possiamo calcolare la probabilità che, ad esempio, \(p\) sia compreso tra 0.5 e 0.75:
# A tibble: 1 × 1
p_between_0.5_and_0.75
<dbl>
1 0.595partion_vector <- c("italic(p)<0.5", "{0.5<italic(p)}<0.75", "lower~80*'%'", "middle~80*'%'")
draws_df |>
mutate(
`italic(p)<0.5` = p < 0.5,
`{0.5<italic(p)}<0.75` = p > 0.5 & p < 0.75,
`lower~80*'%'` = p < quantile(p, probs = 0.8),
`middle~80*'%'` = p > quantile(p, probs = 0.1) & p < quantile(p, probs = 0.9)
) |>
pivot_longer(cols = `italic(p)<0.5`:`middle~80*'%'`) |>
mutate(name = factor(name, levels = partion_vector)) |>
ggplot(aes(x = p, fill = value)) +
geom_histogram(boundary = 0, binwidth = 0.01) +
scale_x_continuous(expression(proportion ~ water ~ (italic(p))), limits = 0:1) +
scale_y_continuous(NULL, breaks = NULL) +
scale_fill_viridis_d(end = 0.6, breaks = NULL) +
facet_wrap(~name, labeller = label_parsed)Warning: Dropping 'draws_df' class as required metadata was removed.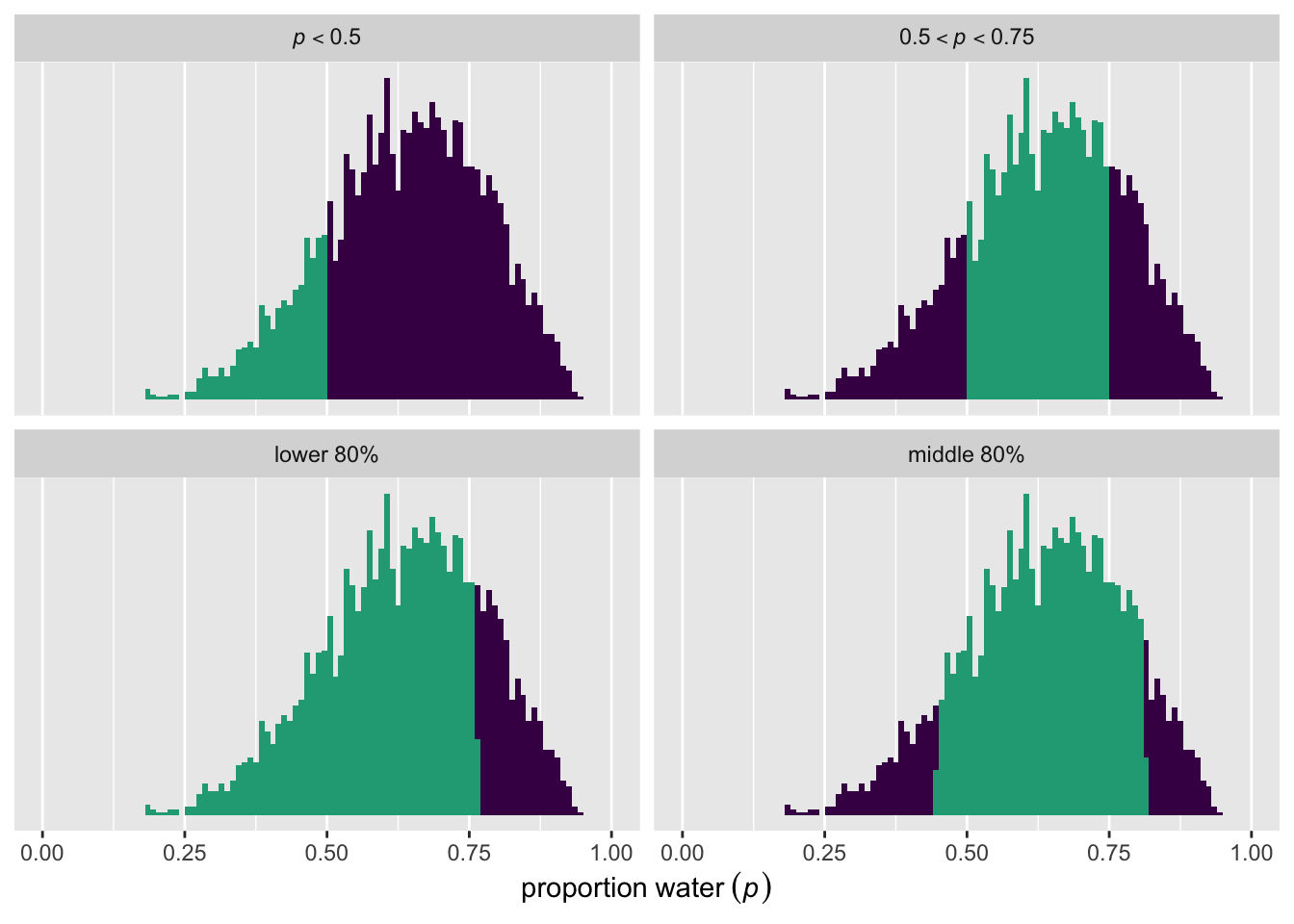
68.5.2 Visualizzazione dei campioni
Visualizzare le distribuzioni posteriori è semplice: basta passare l’oggetto restituito dal metodo $draws() direttamente alle funzioni di plotting del pacchetto bayesplot.
68.6 Diagnostica del campionatore
Il metodo $sampler_diagnostics() estrae i valori dei parametri del campionatore (come treedepth__, divergent__, ecc.) in formati supportati dal pacchetto posterior.
str(fit$sampler_diagnostics(format = "df"))draws_df [4,000 × 9] (S3: draws_df/draws/tbl_df/tbl/data.frame)
$ treedepth__ : num [1:4000] 2 1 1 2 1 2 2 1 1 2 ...
$ divergent__ : num [1:4000] 0 0 0 0 0 0 0 0 0 0 ...
$ energy__ : num [1:4000] 7.21 8.57 9.3 9.06 7.23 ...
$ accept_stat__: num [1:4000] 1 0.762 0.816 1 0.999 ...
$ stepsize__ : num [1:4000] 0.937 0.937 0.937 0.937 0.937 ...
$ n_leapfrog__ : num [1:4000] 3 3 1 3 3 3 3 1 1 3 ...
$ .chain : int [1:4000] 1 1 1 1 1 1 1 1 1 1 ...
$ .iteration : int [1:4000] 1 2 3 4 5 6 7 8 9 10 ...
$ .draw : int [1:4000] 1 2 3 4 5 6 7 8 9 10 ...fit$diagnostic_summary()$num_divergent
[1] 0 0 0 0
$num_max_treedepth
[1] 0 0 0 0
$ebfmi
[1] 1.1950436 0.9774677 1.2009506 0.9659775Questo processo consente di esaminare in dettaglio le prestazioni del campionatore e di verificare eventuali problemi o inefficienze durante l’esecuzione del modello.
Informazioni sull’Ambiente di Sviluppo
R version 4.4.2 (2024-10-31)
Platform: aarch64-apple-darwin20
Running under: macOS Sequoia 15.1.1
Matrix products: default
BLAS: /Library/Frameworks/R.framework/Versions/4.4-arm64/Resources/lib/libRblas.0.dylib
LAPACK: /Library/Frameworks/R.framework/Versions/4.4-arm64/Resources/lib/libRlapack.dylib; LAPACK version 3.12.0
locale:
[1] C/UTF-8/C/C/C/C
time zone: Europe/Rome
tzcode source: internal
attached base packages:
[1] stats graphics grDevices utils datasets methods base
other attached packages:
[1] here_1.0.1 bayesplot_1.11.1 posterior_1.6.0
[4] cmdstanr_0.8.1.9000 lubridate_1.9.3 forcats_1.0.0
[7] stringr_1.5.1 dplyr_1.1.4 purrr_1.0.2
[10] readr_2.1.5 tidyr_1.3.1 tibble_3.2.1
[13] ggplot2_3.5.1 tidyverse_2.0.0
loaded via a namespace (and not attached):
[1] tensorA_0.36.2.1 utf8_1.2.4 generics_0.1.3
[4] stringi_1.8.4 hms_1.1.3 digest_0.6.37
[7] magrittr_2.0.3 evaluate_1.0.1 grid_4.4.2
[10] timechange_0.3.0 fastmap_1.2.0 plyr_1.8.9
[13] rprojroot_2.0.4 jsonlite_1.8.9 processx_3.8.4
[16] backports_1.5.0 ps_1.8.1 fansi_1.0.6
[19] viridisLite_0.4.2 scales_1.3.0 abind_1.4-8
[22] cli_3.6.3 rlang_1.1.4 munsell_0.5.1
[25] withr_3.0.2 yaml_2.3.10 tools_4.4.2
[28] reshape2_1.4.4 tzdb_0.4.0 checkmate_2.3.2
[31] colorspace_2.1-1 vctrs_0.6.5 R6_2.5.1
[34] matrixStats_1.4.1 lifecycle_1.0.4 htmlwidgets_1.6.4
[37] pkgconfig_2.0.3 pillar_1.9.0 gtable_0.3.6
[40] Rcpp_1.0.13-1 data.table_1.16.2 glue_1.8.0
[43] xfun_0.49 tidyselect_1.2.1 knitr_1.49
[46] farver_2.1.2 htmltools_0.5.8.1 labeling_0.4.3
[49] rmarkdown_2.29 compiler_4.4.2 distributional_0.5.0