import logging
import os
import warnings
warnings.simplefilter(action="ignore", category=FutureWarning)
import cmdstanpy
from cmdstanpy import CmdStanModel
cmdstanpy.utils.get_logger().setLevel(logging.ERROR)
import numpy as np
import pandas as pd
import seaborn as sns
import matplotlib.pyplot as plt
import arviz as az
import scipy.stats as stats61 Modello di Poisson (1)
Prerequisiti
Concetti e competenze chiave
Preparazione del Notebook
seed: int = sum(map(ord, "stan_poisson_model"))
rng: np.random.Generator = np.random.default_rng(seed=seed)
sns.set_theme(palette="colorblind")
az.style.use("arviz-darkgrid")
%config InlineBackend.figure_format = "retina"
# Get the home directory
home_directory = os.path.expanduser("~")
# Construct the path to the Quarto project directory
project_directory = os.path.join(
home_directory, '_repositories', 'psicometria')Introduction
Nel Capitolo 48, abbiamo determinato la distribuzione a posteriori sia attraverso il metodo basato su griglia, sia tramite la derivazione analitica utilizzando la famiglia coniugata gamma-poisson. In questo capitolo, affronteremo lo stesso problema utilizzando Stan.
61.1 Dati
Riconsideriamo lo stesso problema discusso nella sezione ?sec-poisson-model. I dati disponibili sono:
y = np.array([2, 1, 3, 2, 2, 1, 1, 1])61.2 Modello di Poisson con Stan
Nel modello Stan utilizzeremo una verosimiglianza di Poisson e una distribuzione a priori per il parametro \(\lambda\) modellata da una distribuzione Gamma con parametri \(\alpha_{\text{prior}} = 9\) e \(\beta_{\text{prior}} = 2\).
stan_file = os.path.join(project_directory, "stan", "gamma_poisson_mcmc.stan")
model = CmdStanModel(stan_file=stan_file)print(model.code())data {
int<lower=0> N; // numero di osservazioni
array[N] int<lower=0> y; // dati osservati
real<lower=0> alpha_prior; // parametro alpha della priori Gamma
real<lower=0> beta_prior; // parametro beta della priori Gamma
}
parameters {
real<lower=0> lambda; // parametro di interesse
}
model {
// Priori
lambda ~ gamma(alpha_prior, beta_prior);
// Verosimiglianza
y ~ poisson(lambda);
}
generated quantities {
real alpha_post = alpha_prior + sum(y);
real beta_post = beta_prior + N;
}
Sistemiamo i dati nel formato richiesto da Stan.
N = len(y)
alpha_prior = 9
beta_prior = 2
# Preparazione dei dati per Stan
stan_data = {"N": N, "y": y, "alpha_prior": alpha_prior, "beta_prior": beta_prior}
print(stan_data){'N': 8, 'y': array([2, 1, 3, 2, 2, 1, 1, 1]), 'alpha_prior': 9, 'beta_prior': 2}Eseguiamo il campionamento.
fit = model.sample(
data=stan_data,
iter_warmup=2_000,
iter_sampling=2_000,
seed=123,
show_progress=False,
show_console=False,
)Estraiamo un campione casuale dalla distribuzione a posteriori di lambda.
lambda_samples = fit.stan_variable("lambda")Calcoliamo i parametri della Gamma a posteriori teorica.
alpha_post = alpha_prior + np.sum(y)
beta_post = beta_prior + NCreiamo un istogramma con i campioni della distribuzione a posteriori di \(\lambda\) e sovrapposta la densità teorica della distribuzione a posteriori.
plt.figure(figsize=(10, 6))
# Istogramma normalizzato dei campioni MCMC
plt.hist(
lambda_samples,
bins=50,
density=True,
alpha=0.7,
label="Distribuzione a posteriori empirica",
)
# Gamma teorica
x = np.linspace(0, max(lambda_samples), 200)
gamma_pdf = stats.gamma.pdf(x, a=alpha_post, scale=1 / beta_post)
plt.plot(x, gamma_pdf, "r-", lw=2, label="Distribuzione Gamma teorica")
# Personalizzazione del grafico
plt.title("Distribuzione a posteriori di lambda")
plt.xlabel("lambda")
plt.ylabel("Densità")
plt.legend()
# Aggiungiamo informazioni sui parametri
plt.text(
0.95,
0.95,
f"alpha_post = {alpha_post:.2f}\nbeta_post = {beta_post:.2f}",
transform=plt.gca().transAxes,
verticalalignment="top",
horizontalalignment="right",
bbox=dict(boxstyle="round", facecolor="white", alpha=0.8),
)Text(0.95, 0.95, 'alpha_post = 22.00\nbeta_post = 10.00')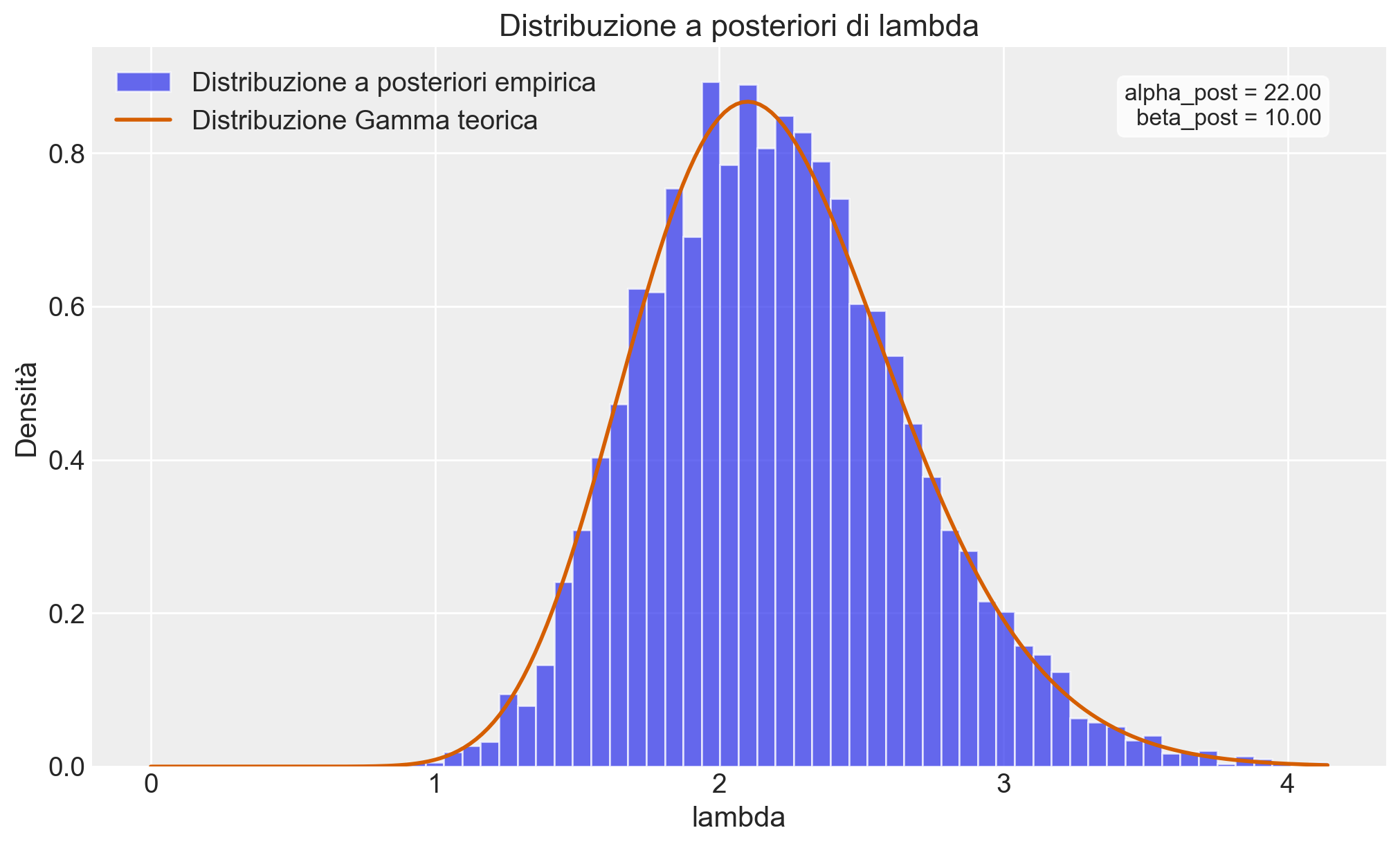
Usiamo ArviZ per un sommario della distribuzione a posteriori del parametro \(\lambda\).
az.summary(fit, var_names="lambda")| mean | sd | hdi_3% | hdi_97% | mcse_mean | mcse_sd | ess_bulk | ess_tail | r_hat | |
|---|---|---|---|---|---|---|---|---|---|
| lambda | 2.214 | 0.467 | 1.395 | 3.11 | 0.008 | 0.006 | 3262.0 | 4279.0 | 1.0 |
Generiamo l’intervallo di credibilità al 94% per la distribuzione a posteriori del parametro lambda.
az.plot_posterior(fit, var_names="lambda")
plt.show()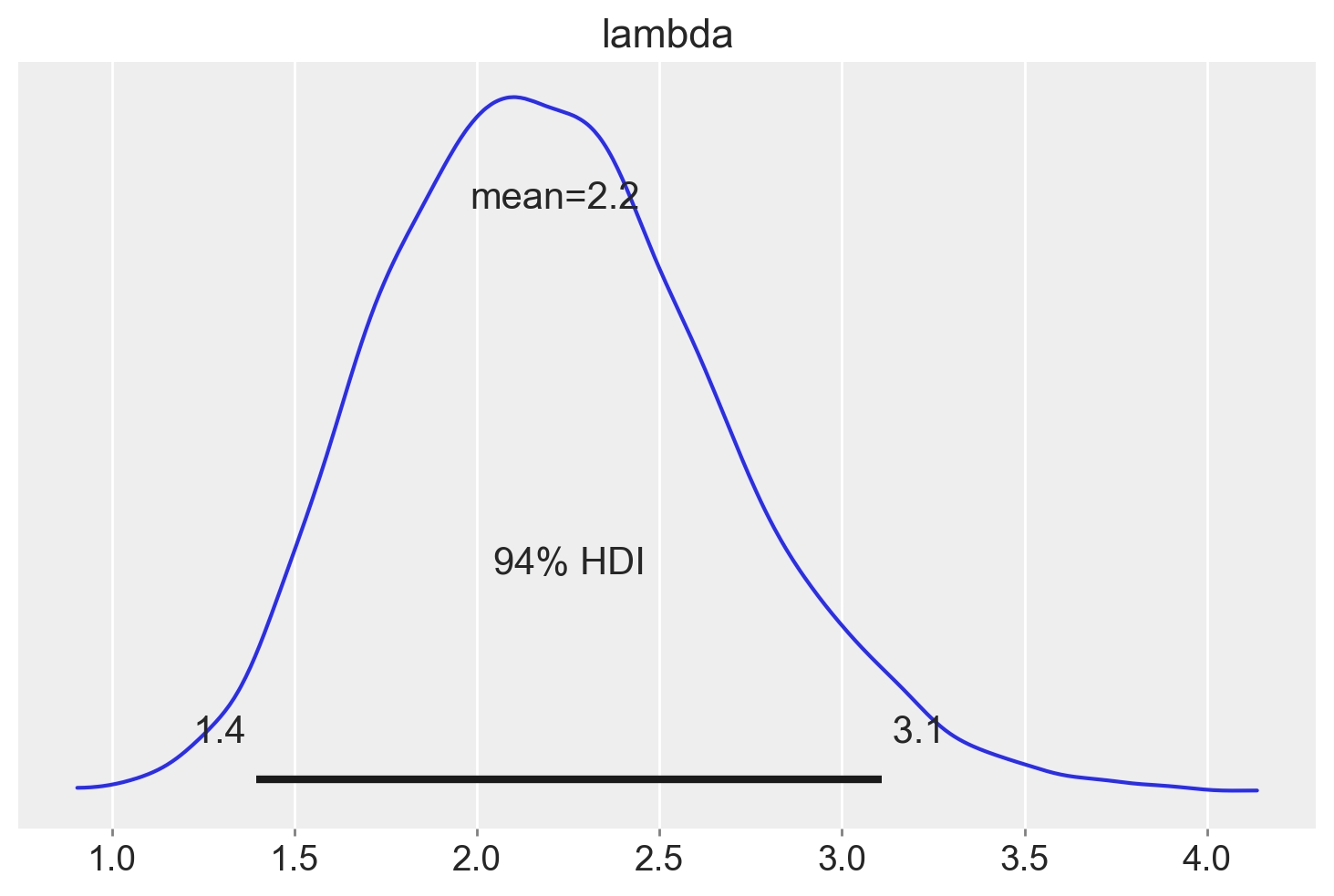
In sintesi, analizzando i dati disponibili e utilizzando una distribuzione a priori Gamma(9, 2), possiamo affermare con un grado di certezza soggettivo del 94% che il tasso stimato di occorrenza dell’evento considerato sia di 2.2 compulsioni all’ora, con un intervallo di credibilità compreso tra 1.4 e 3.1.
Informazioni sull’Ambiente di Sviluppo
%load_ext watermark
%watermark -n -u -v -iv -m Last updated: Fri Aug 16 2024
Python implementation: CPython
Python version : 3.12.4
IPython version : 8.26.0
Compiler : Clang 16.0.6
OS : Darwin
Release : 23.6.0
Machine : arm64
Processor : arm
CPU cores : 8
Architecture: 64bit
pandas : 2.2.2
arviz : 0.18.0
scipy : 1.14.0
logging : 0.5.1.2
seaborn : 0.13.2
numpy : 1.26.4
matplotlib: 3.9.1
cmdstanpy : 1.2.4