35. Modelli di crescita latenti bivariati#
I ricercatori spesso studiano più costrutti nel tempo per comprendere il loro sviluppo congiunto e le correlazioni tra di loro. Esistono diversi modelli statistici per studiare il cambiamento in più entità contemporaneamente. In questo capitolo ne copriamo due: il modello di crescita multivariato (MGM), che esamina le interrelazioni tra due processi di crescita distinti, e il modello di crescita con covariata variabile nel tempo (TVC), che stima l’effetto di una variabile che cambia nel tempo sui punteggi mentre modella contemporaneamente il cambiamento in quei punteggi con un modello di crescita. Questi modelli sono comuni nella ricerca sullo sviluppo e rispondono a domande specifiche riguardo le associazioni tra entità che cambiano simultaneamente e tra i cambiamenti per individui correlati.
Carichiamo i pacchetti necessari.
Questo tutorial mostra come adattare modelli di crescita lineare multivariati (bivariati) nel framework SEM in R. I dati sono tratti dal Capitolo 8 Grimm et al. [GRE16]. In particolare, utilizzando il set di dati NLSY-CYA, esaminiamo come le differenze individuali nella variazione del rendimento in matematica dei bambini durante la scuola siano correlate alle differenze individuali nella variazione dell’iperattività dei bambini (valutata dagli insegnanti). Iniziamo a leggere i dati.
# set filepath for data file
filepath <- "https://raw.githubusercontent.com/LRI-2/Data/main/GrowthModeling/nlsy_math_hyp_long_R.dat"
# read in the text data file using the url() function
dat <- read.table(
file = url(filepath),
na.strings = "."
) # indicates the missing data designator
# copy data with new name
nlsy_math_hyp_long <- dat
# Add names the columns of the data set
names(nlsy_math_hyp_long) <- c(
"id", "female", "lb_wght", "anti_k1",
"math", "comp", "rec", "bpi", "as", "anx", "hd",
"hyp", "dp", "wd",
"grade", "occ", "age", "men", "spring", "anti"
)
# reducing to variables of interest
nlsy_math_hyp_long <- nlsy_math_hyp_long[, c("id", "grade", "math", "hyp")]
# view the first few observations in the data set
head(nlsy_math_hyp_long, 10)
| id | grade | math | hyp | |
|---|---|---|---|---|
| <int> | <int> | <int> | <int> | |
| 1 | 201 | 3 | 38 | 0 |
| 2 | 201 | 5 | 55 | 0 |
| 3 | 303 | 2 | 26 | 1 |
| 4 | 303 | 5 | 33 | 1 |
| 5 | 2702 | 2 | 56 | 2 |
| 6 | 2702 | 4 | 58 | 3 |
| 7 | 2702 | 8 | 80 | 3 |
| 8 | 4303 | 3 | 41 | 1 |
| 9 | 4303 | 4 | 58 | 1 |
| 10 | 5002 | 4 | 46 | 3 |
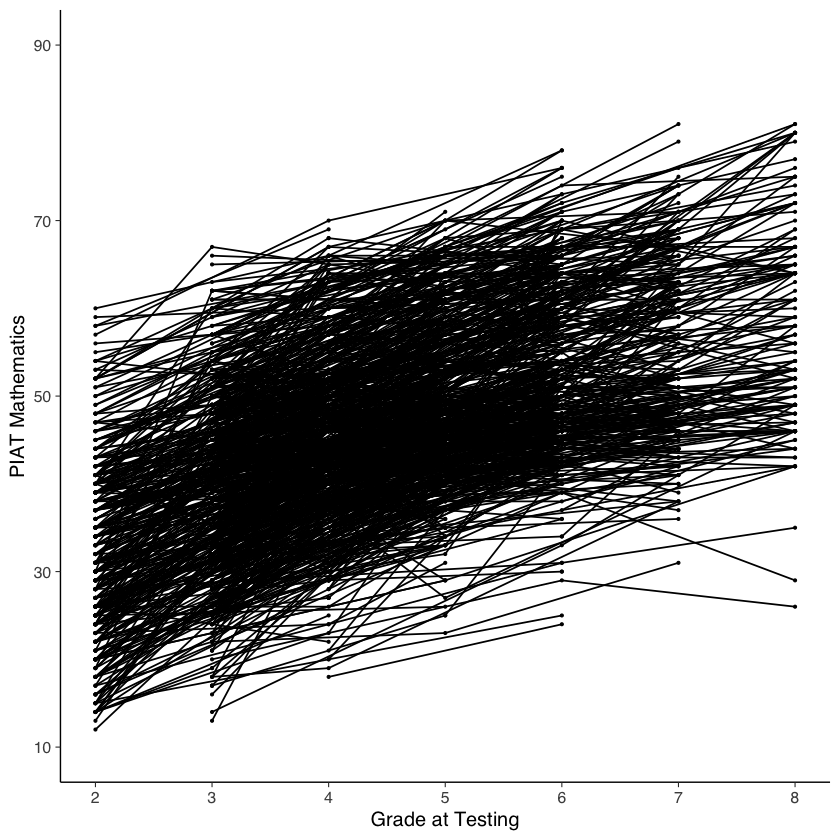
Il nostro interesse specifico è il cambiamento intra-individuale nelle misure ripetute di matematica e iperattività durante il periodo scolastico (grade).
# intraindividual change trajetories
ggplot(
data = nlsy_math_hyp_long, # data set
aes(x = grade, y = math, group = id)
) + # setting variables
geom_point(size = .5) + # adding points to plot
geom_line() + # adding lines to plot
# setting the x-axis with breaks and labels
scale_x_continuous(
limits = c(2, 8),
breaks = c(2, 3, 4, 5, 6, 7, 8),
name = "Grade at Testing"
) +
# setting the y-axis with limits breaks and labels
scale_y_continuous(
limits = c(10, 90),
breaks = c(10, 30, 50, 70, 90),
name = "PIAT Mathematics"
)
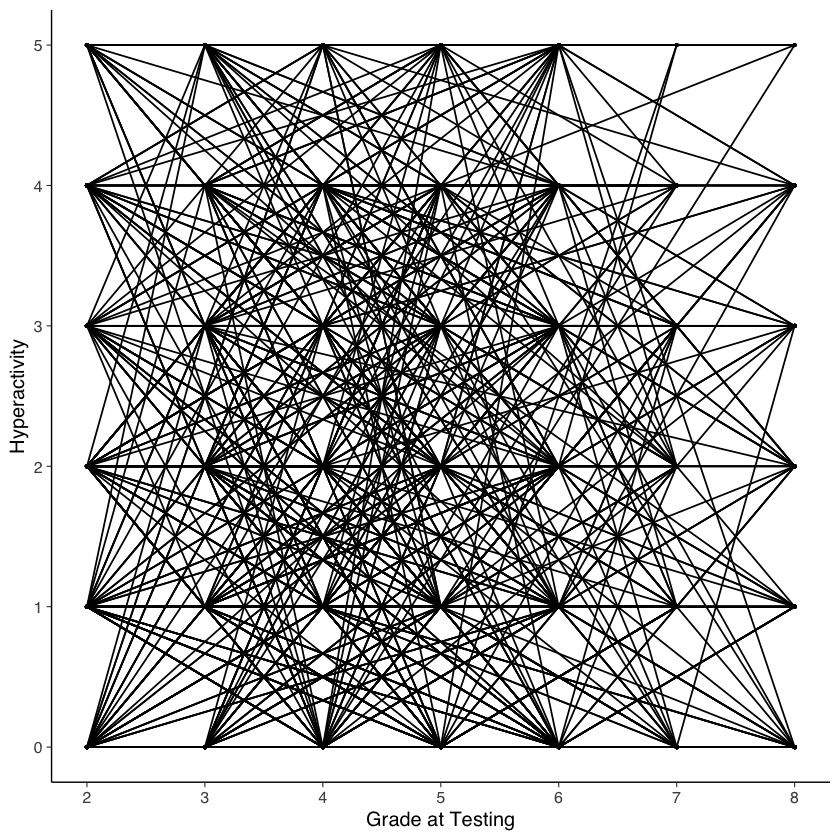
Esaminiamo i punteggi di iperattività in funzione di grade.
# intraindividual change trajetories
ggplot(
data = nlsy_math_hyp_long, # data set
aes(x = grade, y = hyp, group = id)
) + # setting variables
geom_point(size = .5) + # adding points to plot
geom_line() + # adding lines to plot
# setting the x-axis with breaks and labels
scale_x_continuous(
limits = c(2, 8),
breaks = c(2, 3, 4, 5, 6, 7, 8),
name = "Grade at Testing"
) +
# setting the y-axis with limits breaks and labels
scale_y_continuous(
limits = c(0, 5),
breaks = c(0, 1, 2, 3, 4, 5),
name = "Hyperactivity"
)
Warning message:
“Removed 51 rows containing missing values (`geom_point()`).”
Warning message:
“Removed 44 rows containing missing values (`geom_line()`).”
Per semplicità, leggiamo i dati in formato long da file.
# set filepath for data file
filepath <- "https://raw.githubusercontent.com/LRI-2/Data/main/GrowthModeling/nlsy_math_hyp_wide_R.dat"
# read in the text data file using the url() function
dat <- read.table(
file = url(filepath),
na.strings = "."
) # indicates the missing data designator
# copy data with new name
nlsy_math_hyp_wide <- dat
# Add names the columns of the data set
# Give the variable names
names(nlsy_math_hyp_wide) <- c(
"id", "female", "lb_wght", "anti_k1",
"math2", "math3", "math4", "math5", "math6", "math7", "math8",
"comp2", "comp3", "comp4", "comp5", "comp6", "comp7", "comp8",
"rec2", "rec3", "rec4", "rec5", "rec6", "rec7", "rec8",
"bpi2", "bpi3", "bpi4", "bpi5", "bpi6", "bpi7", "bpi8",
"asl2", "asl3", "asl4", "asl5", "asl6", "asl7", "asl8",
"ax2", "ax3", "ax4", "ax5", "ax6", "ax7", "ax8",
"hds2", "hds3", "hds4", "hds5", "hds6", "hds7", "hds8",
"hyp2", "hyp3", "hyp4", "hyp5", "hyp6", "hyp7", "hyp8",
"dpn2", "dpn3", "dpn4", "dpn5", "dpn6", "dpn7", "dpn8",
"wdn2", "wdn3", "wdn4", "wdn5", "wdn6", "wdn7", "wdn8",
"age2", "age3", "age4", "age5", "age6", "age7", "age8",
"men2", "men3", "men4", "men5", "men6", "men7", "men8",
"spring2", "spring3", "spring4", "spring5", "spring6", "spring7", "spring8",
"anti2", "anti3", "anti4", "anti5", "anti6", "anti7", "anti8"
)
# reducing to variables of interest
nlsy_multi_wide <- nlsy_math_hyp_wide[, c(
"id",
"math2", "math3", "math4", "math5", "math6", "math7", "math8",
"hyp2", "hyp3", "hyp4", "hyp5", "hyp6", "hyp7", "hyp8"
)]
# view the first few observations in the data set
head(nlsy_multi_wide, 10)
| id | math2 | math3 | math4 | math5 | math6 | math7 | math8 | hyp2 | hyp3 | hyp4 | hyp5 | hyp6 | hyp7 | hyp8 | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| <int> | <int> | <int> | <int> | <int> | <int> | <int> | <int> | <int> | <int> | <int> | <int> | <int> | <int> | <int> | |
| 1 | 201 | NA | 38 | NA | 55 | NA | NA | NA | NA | 0 | NA | 0 | NA | NA | NA |
| 2 | 303 | 26 | NA | NA | 33 | NA | NA | NA | 1 | NA | NA | 1 | NA | NA | NA |
| 3 | 2702 | 56 | NA | 58 | NA | NA | NA | 80 | 2 | NA | 3 | NA | NA | NA | 3 |
| 4 | 4303 | NA | 41 | 58 | NA | NA | NA | NA | NA | 1 | 1 | NA | NA | NA | NA |
| 5 | 5002 | NA | NA | 46 | NA | 54 | NA | 66 | NA | NA | 3 | NA | 2 | NA | 3 |
| 6 | 5005 | 35 | NA | 50 | NA | 60 | NA | 59 | 0 | NA | 3 | NA | 0 | NA | 1 |
| 7 | 5701 | NA | 62 | 61 | NA | NA | NA | NA | NA | 4 | 3 | NA | NA | NA | NA |
| 8 | 6102 | NA | NA | 55 | 67 | NA | 81 | NA | NA | NA | 2 | 0 | NA | 0 | NA |
| 9 | 6801 | NA | 54 | NA | 62 | NA | 66 | NA | NA | 0 | NA | 1 | NA | 1 | NA |
| 10 | 6802 | NA | 55 | NA | 66 | NA | 68 | NA | NA | 0 | NA | 0 | NA | 0 | NA |
Per l’implementazione SEM, utilizziamo i punteggi di rendimento in matematica e iperattività e le covariate invarianti nel tempo dai dati Wide. Specifichiamo un modello di crescita lineare bivariato usando la sintassi lavaan.
# writing out linear growth model in full SEM way
bivariate_lavaan_model <- "
# latent variable definitions
#intercept for math
eta_1 =~ 1*math2
eta_1 =~ 1*math3
eta_1 =~ 1*math4
eta_1 =~ 1*math5
eta_1 =~ 1*math6
eta_1 =~ 1*math7
eta_1 =~ 1*math8
#linear slope for math
eta_2 =~ 0*math2
eta_2 =~ 1*math3
eta_2 =~ 2*math4
eta_2 =~ 3*math5
eta_2 =~ 4*math6
eta_2 =~ 5*math7
eta_2 =~ 6*math8
#intercept for hyp
eta_3 =~ 1*hyp2
eta_3 =~ 1*hyp3
eta_3 =~ 1*hyp4
eta_3 =~ 1*hyp5
eta_3 =~ 1*hyp6
eta_3 =~ 1*hyp7
eta_3 =~ 1*hyp8
#linear slope for hyp
eta_4 =~ 0*hyp2
eta_4 =~ 1*hyp3
eta_4 =~ 2*hyp4
eta_4 =~ 3*hyp5
eta_4 =~ 4*hyp6
eta_4 =~ 5*hyp7
eta_4 =~ 6*hyp8
# factor variances
eta_1 ~~ eta_1
eta_2 ~~ eta_2
eta_3 ~~ eta_3
eta_4 ~~ eta_4
# covariances among factors
eta_1 ~~ eta_2 + eta_3 + eta_4
eta_2 ~~ eta_3 + eta_4
eta_3 ~~ eta_4
# factor means
eta_1 ~ start(35)*1
eta_2 ~ start(4)*1
eta_3 ~ start(2)*1
eta_4 ~ start(.1)*1
# manifest variances for math (made equivalent by naming theta1)
math2 ~~ theta1*math2
math3 ~~ theta1*math3
math4 ~~ theta1*math4
math5 ~~ theta1*math5
math6 ~~ theta1*math6
math7 ~~ theta1*math7
math8 ~~ theta1*math8
# manifest variances for hyp (made equivalent by naming theta2)
hyp2 ~~ theta2*hyp2
hyp3 ~~ theta2*hyp3
hyp4 ~~ theta2*hyp4
hyp5 ~~ theta2*hyp5
hyp6 ~~ theta2*hyp6
hyp7 ~~ theta2*hyp7
hyp8 ~~ theta2*hyp8
# residual covariances (made equivalent by naming theta12)
math2 ~~ theta12*hyp2
math3 ~~ theta12*hyp3
math4 ~~ theta12*hyp4
math5 ~~ theta12*hyp5
math6 ~~ theta12*hyp6
math7 ~~ theta12*hyp7
math8 ~~ theta12*hyp8
# manifest means for math (fixed at zero)
math2 ~ 0*1
math3 ~ 0*1
math4 ~ 0*1
math5 ~ 0*1
math6 ~ 0*1
math7 ~ 0*1
math8 ~ 0*1
# manifest means for hyp (fixed at zero)
hyp2 ~ 0*1
hyp3 ~ 0*1
hyp4 ~ 0*1
hyp5 ~ 0*1
hyp6 ~ 0*1
hyp7 ~ 0*1
hyp8 ~ 0*1
" # end of model definition
Adattiamo il modello ai dati.
bivariate_lavaan_fit <- sem(bivariate_lavaan_model,
data = nlsy_multi_wide,
meanstructure = TRUE,
estimator = "ML",
missing = "fiml"
)
Warning message in lav_data_full(data = data, group = group, cluster = cluster, :
“lavaan WARNING: some cases are empty and will be ignored:
741”
Warning message in lav_data_full(data = data, group = group, cluster = cluster, :
“lavaan WARNING:
due to missing values, some pairwise combinations have less than
10% coverage; use lavInspect(fit, "coverage") to investigate.”
Warning message in lav_mvnorm_missing_h1_estimate_moments(Y = X[[g]], wt = WT[[g]], :
“lavaan WARNING:
Maximum number of iterations reached when computing the sample
moments using EM; use the em.h1.iter.max= argument to increase the
number of iterations”
Esaminiamo i risultati.
out = summary(bivariate_lavaan_fit, fit.measures = TRUE)
print(out)
lavaan 0.6.15 ended normally after 67 iterations
Estimator ML
Optimization method NLMINB
Number of model parameters 35
Number of equality constraints 18
Used Total
Number of observations 932 933
Number of missing patterns 96
Model Test User Model:
Test statistic 318.885
Degrees of freedom 102
P-value (Chi-square) 0.000
Model Test Baseline Model:
Test statistic 1532.041
Degrees of freedom 91
P-value 0.000
User Model versus Baseline Model:
Comparative Fit Index (CFI) 0.849
Tucker-Lewis Index (TLI) 0.866
Robust Comparative Fit Index (CFI) 1.000
Robust Tucker-Lewis Index (TLI) -6.557
Loglikelihood and Information Criteria:
Loglikelihood user model (H0) -11790.476
Loglikelihood unrestricted model (H1) -11631.033
Akaike (AIC) 23614.951
Bayesian (BIC) 23697.186
Sample-size adjusted Bayesian (SABIC) 23643.196
Root Mean Square Error of Approximation:
RMSEA 0.048
90 Percent confidence interval - lower 0.042
90 Percent confidence interval - upper 0.054
P-value H_0: RMSEA <= 0.050 0.724
P-value H_0: RMSEA >= 0.080 0.000
Robust RMSEA 0.000
90 Percent confidence interval - lower 0.000
90 Percent confidence interval - upper 0.000
P-value H_0: Robust RMSEA <= 0.050 1.000
P-value H_0: Robust RMSEA >= 0.080 0.000
Standardized Root Mean Square Residual:
SRMR 0.097
Parameter Estimates:
Standard errors Standard
Information Observed
Observed information based on Hessian
Latent Variables:
Estimate Std.Err z-value P(>|z|)
eta_1 =~
math2 1.000
math3 1.000
math4 1.000
math5 1.000
math6 1.000
math7 1.000
math8 1.000
eta_2 =~
math2 0.000
math3 1.000
math4 2.000
math5 3.000
math6 4.000
math7 5.000
math8 6.000
eta_3 =~
hyp2 1.000
hyp3 1.000
hyp4 1.000
hyp5 1.000
hyp6 1.000
hyp7 1.000
hyp8 1.000
eta_4 =~
hyp2 0.000
hyp3 1.000
hyp4 2.000
hyp5 3.000
hyp6 4.000
hyp7 5.000
hyp8 6.000
Covariances:
Estimate Std.Err z-value P(>|z|)
eta_1 ~~
eta_2 -0.204 1.154 -0.176 0.860
eta_3 -2.979 0.673 -4.426 0.000
eta_4 0.107 0.164 0.654 0.513
eta_2 ~~
eta_3 0.098 0.161 0.608 0.543
eta_4 -0.040 0.038 -1.061 0.289
eta_3 ~~
eta_4 -0.020 0.031 -0.644 0.520
.math2 ~~
.hyp2 (th12) -0.011 0.233 -0.046 0.963
.math3 ~~
.hyp3 (th12) -0.011 0.233 -0.046 0.963
.math4 ~~
.hyp4 (th12) -0.011 0.233 -0.046 0.963
.math5 ~~
.hyp5 (th12) -0.011 0.233 -0.046 0.963
.math6 ~~
.hyp6 (th12) -0.011 0.233 -0.046 0.963
.math7 ~~
.hyp7 (th12) -0.011 0.233 -0.046 0.963
.math8 ~~
.hyp8 (th12) -0.011 0.233 -0.046 0.963
Intercepts:
Estimate Std.Err z-value P(>|z|)
eta_1 35.259 0.356 99.178 0.000
eta_2 4.343 0.088 49.139 0.000
eta_3 1.903 0.058 32.702 0.000
eta_4 -0.057 0.014 -3.950 0.000
.math2 0.000
.math3 0.000
.math4 0.000
.math5 0.000
.math6 0.000
.math7 0.000
.math8 0.000
.hyp2 0.000
.hyp3 0.000
.hyp4 0.000
.hyp5 0.000
.hyp6 0.000
.hyp7 0.000
.hyp8 0.000
Variances:
Estimate Std.Err z-value P(>|z|)
eta_1 64.711 5.664 11.424 0.000
eta_2 0.752 0.329 2.282 0.023
eta_3 1.542 0.156 9.908 0.000
eta_4 0.005 0.009 0.539 0.590
.math2 (tht1) 36.126 1.863 19.390 0.000
.math3 (tht1) 36.126 1.863 19.390 0.000
.math4 (tht1) 36.126 1.863 19.390 0.000
.math5 (tht1) 36.126 1.863 19.390 0.000
.math6 (tht1) 36.126 1.863 19.390 0.000
.math7 (tht1) 36.126 1.863 19.390 0.000
.math8 (tht1) 36.126 1.863 19.390 0.000
.hyp2 (tht2) 1.104 0.057 19.404 0.000
.hyp3 (tht2) 1.104 0.057 19.404 0.000
.hyp4 (tht2) 1.104 0.057 19.404 0.000
.hyp5 (tht2) 1.104 0.057 19.404 0.000
.hyp6 (tht2) 1.104 0.057 19.404 0.000
.hyp7 (tht2) 1.104 0.057 19.404 0.000
.hyp8 (tht2) 1.104 0.057 19.404 0.000
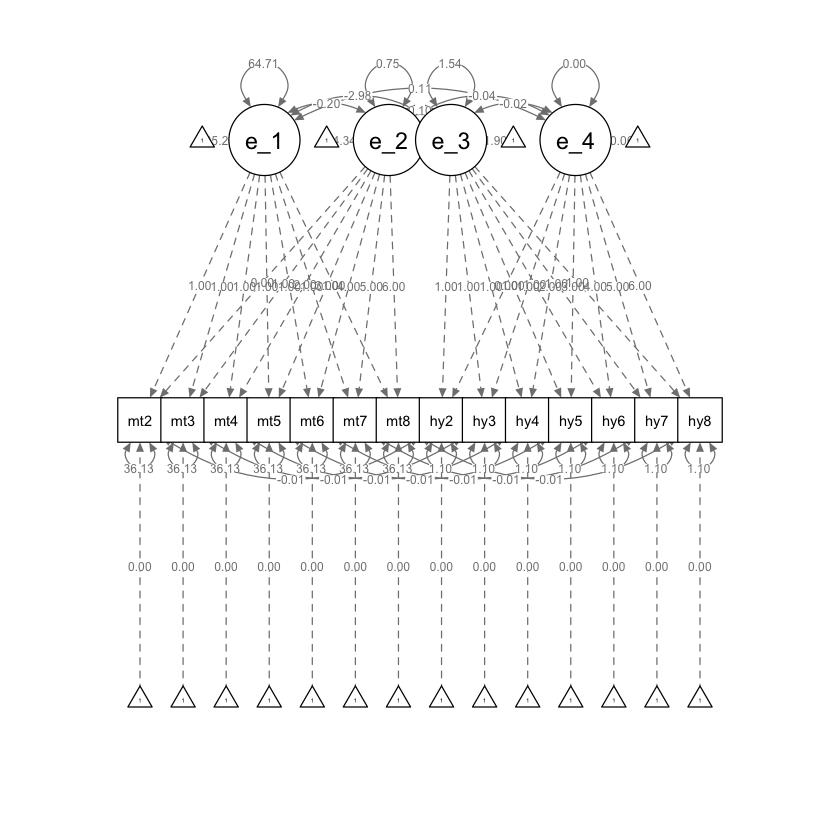
Il parametro principale di interesse, la covarianza pendenza-pendenza eta_3 ~~eta_4, non è significativamente diverso da zero.
Generiamo un diagramma di percorso.
semPaths(bivariate_lavaan_fit, what = "path", whatLabels = "par")
35.1. Conclusione#
Questo tutorial ha presentato come il modello di crescita multivariato possa essere utilizzato per esaminare le interrelazioni nel cambiamento - in particolare la correlazione between-person tra le pendenze.
