30. LGM e modelli misti#
I modelli di crescita latente (LGM) sono una particolare forma di analisi fattoriale confermativa (CFA) che prevedono la fissazione dei valori delle saturazioni fattoriali a valori predefiniti e in molti casi la traiettoria nel tempo può essere descritta come una funzione lineare o quadratica. I fattori di crescita, che rappresentano le differenze individuali nei dati, vengono rappresentati da variabili latenti continue, note come growth factors. Per aiutare a comprendere meglio i modelli LGM, presenteremo questi modelli attraverso un confronto con i modelli misti [HW22] in questo capitolo.
30.1. Modelli misti#
I modelli lineari standard presuppongono che ogni osservazione sia indipendente dalle altre. Tuttavia, ci sono casi in cui abbiamo più misure ripetute per lo stesso individuo nel tempo, e queste osservazioni sono quindi correlate. In questi casi, si possono utilizzare i modelli a effetti misti, che tengono conto della correlazione tra le osservazioni raggruppate.
I modelli misti rappresentano un compromesso tra la modellizzazione pooled e quella separata. La modellizzazione pooled combina tutti i dati in un unico dataset, ma può portare a una perdita di informazioni sulle differenze tra i gruppi di osservazioni. La modellizzazione separata adatta un modello per ogni gruppo di osservazioni, ma può portare a problemi di sovrapparametrizzazione e scarsa generalizzazione ai nuovi dati.
I modelli misti risolvono questi problemi combinando la modellizzazione pooled e quella separata. In questo modo, il modello tiene conto delle differenze tra i gruppi di dati e sfrutta l’informazione comune per migliorare la precisione delle stime dei parametri. Per motivi storici, i dati entro il soggetto sono tipicamente chiamati dati di livello 1 e i dati tra soggetti sono noti come dati di livello 2.
Come i modelli di regressione tradizionali, i modelli misti includono un’intercetta fissa, pendenze fissi dei predittori e un residuo del modello (per la deviazione del risultato effettivo dal risultato previsto dal modello). Ma a differenza dei modelli di regressione tradizionali, l’intercetta e le pendenze dei predittori nei modelli misti possono variare tra le unità di campionamento.
In termini formali, il modello statistico applicato per lo studio di dati dipendenti può essere descritto nel modo seguente. Data l’unità statistica \(i\) appartenente al gruppo \(j\) (con \(i = 1, ..., n_j\) e \(j = 1, ..., N\)) per \(N\) gruppi di numerosità \(n_j\), facendo riferimento al caso di una variabile dipendente \(Y\), di una variabile individuale indipendente \(x\) e di una variabile \(z\) di gruppo, il modello lineare multilivello può essere rappresentato mediante un’equazione di primo livello,
che rappresenta in base ai coefficienti \(\beta_{0j}\) , \(\beta_{1j}\) la relazione esistente tra le variabili (\(Y\) e \(x\)) riferite all’unità statistica di primo livello, nella quale il termine d’errore \(\varepsilon_{ij}\) si assume con distribuzione normale, con media pari a zero e varianza costante pari a \(\sigma^2\), e mediante le equazioni di secondo livello,
che rappresentano in base ai coefficienti \(\gamma_{00}, \gamma_{01}, \gamma_{10}, \gamma_{11}\) le relazioni esistenti tra la variabile osservata \(z\) su ciascun gruppo (unità di secondo livello) e i coefficienti \(\beta_{0j}, \beta_{1j}\) inseriti nell’equazione di primo livello. I termini d’errore \(U_{0j}, U_{1j}\) si assumono con media zero, varianze costanti \(\tau_0^2, \tau_1^2\), e indipendenti tra loro e dal termine di errore \(\varepsilon_{ij}\), che figura nell’equazione di primo livello 1.
Per quanto riguarda la componente \(\sigma^2\) (detta within) della varianza della variabile \(Y\) tra le unità statistiche all’interno dei gruppi e la componente \(\tau_0^2\) (detta between) della varianza esistente tra i gruppi, il rapporto
tra la componente di varianza fra gruppi (\(\tau_0^2\)) e la varianza totale (\(\tau_0^2 + \sigma^2\)) viene definito coefficiente di correlazione intragruppo, ed è utilizzato come una misura della relazione esistente tra i valori della variabile \(Y\) all’interno di ogni gruppo o unità di secondo livello.
30.2. Simulare effetti casuali#
Possiamo fornire una dimostrazione del funzionamento dei modelli misti con una simulazione. Ciò ci permetterà di meglio comprendere i modelli a crescita latente. Simuleremo dei dati bilanciati, con punteggi su quattro rilevazioni temporali per 500 individui (soggetti). Esamineremo il tasso di crescita (‘growth’) e consentiremo la presenza di intercette e pendenze specifiche per i diversi soggetti.
Le istruzioni seguenti generano i dati (per i nostri scopi, non è importante capire i dettagli di questa porzione di codice).
set.seed(12345)
n <- 500
timepoints <- 4
time <- rep(0:3, times = n)
subject <- rep(1:n, each = 4)
intercept <- .5
slope <- .25
randomEffectsCorr <- matrix(c(1, .2, .2, 1), ncol = 2)
randomEffects <- MASS::mvrnorm(
n,
mu = c(0, 0), Sigma = randomEffectsCorr, empirical = T
) %>%
data.frame()
colnames(randomEffects) <- c("Int", "Slope")
La simulazione comprende gli effetti ‘fissi’, ovvero l’intercetta e la pendenza della regressione standard, impostati rispettivamente a 0.5 e 0.25. Verrà simulata una correlazione di 0.2 tra intercetta e pendenza specifiche per ciascun soggetto. Per questa ragione, i dati verranno estratti da una distribuzione normale multivariata. Porremo uguale a 1 la varianza per entrambi gli effetti.
Esaminiamo i dati ottenuti.
data.frame(Subject = subject, time = time, randomEffects[subject, ]) %>%
head(10)
| Subject | time | Int | Slope | |
|---|---|---|---|---|
| <int> | <int> | <dbl> | <dbl> | |
| 1 | 1 | 0 | -1.3322902 | -0.9548087 |
| 1.1 | 1 | 1 | -1.3322902 | -0.9548087 |
| 1.2 | 1 | 2 | -1.3322902 | -0.9548087 |
| 1.3 | 1 | 3 | -1.3322902 | -0.9548087 |
| 2 | 2 | 0 | -2.1261548 | -1.7813625 |
| 2.1 | 2 | 1 | -2.1261548 | -1.7813625 |
| 2.2 | 2 | 2 | -2.1261548 | -1.7813625 |
| 2.3 | 2 | 3 | -2.1261548 | -1.7813625 |
| 3 | 3 | 0 | 0.4606242 | 0.3039838 |
| 3.1 | 3 | 1 | 0.4606242 | 0.3039838 |
Per ottenere una variabile target, sommiamo semplicemente gli effetti casuali così ottenuti all’intercetta complessiva e facciamo lo stesso per le pendenze. Sommeremo ai dati un rumore gaussiano con deviazione standard uguale a \(\sigma\) = 0.5.
set.seed(12345)
sigma <- .5
y1 <- (intercept + randomEffects$Int[subject]) + # random intercepts
(slope + randomEffects$Slope[subject]) * time + # random slopes
rnorm(n * timepoints, mean = 0, sd = sigma)
d <- data.frame(subject, time, y1)
d %>%
head(10)
| subject | time | y1 | |
|---|---|---|---|
| <int> | <int> | <dbl> | |
| 1 | 1 | 0 | -0.5395258 |
| 2 | 1 | 1 | -1.1823659 |
| 3 | 1 | 2 | -2.2965593 |
| 4 | 1 | 3 | -3.1734649 |
| 5 | 2 | 0 | -1.3232110 |
| 6 | 2 | 1 | -4.0664952 |
| 7 | 2 | 2 | -4.3738304 |
| 8 | 2 | 3 | -6.3583342 |
| 9 | 3 | 0 | 0.8185443 |
| 10 | 3 | 1 | 1.0549470 |
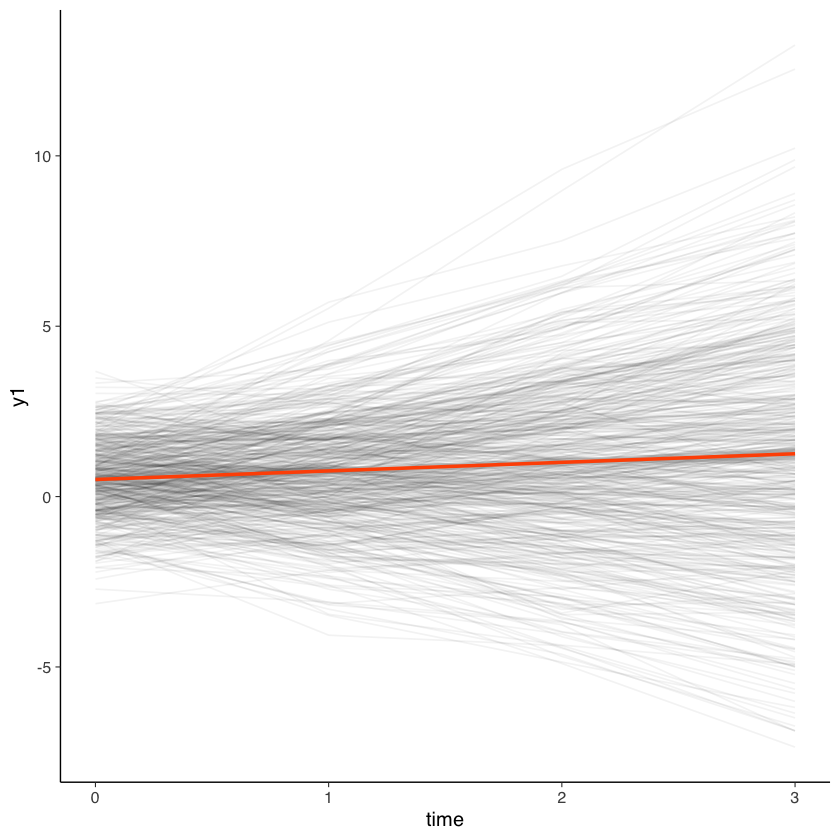
Il grafico seguente mostra le rette di regressione per ciascuno dei 500 soggetti.
ggplot(d, aes(x = time, y = y1)) +
geom_path(aes(group = subject), alpha = .05) +
geom_smooth(method = "lm", color = "#ff5500")
`geom_smooth()` using formula = 'y ~ x'
Adattiamo ai dati un modello misto utilizzando la funzione lmer del pacchetto lme4.
var(d$y1)
m0 <- lmer(y1 ~ 1 + (1 | subject), data = d)
VarCorr(m0)
Groups Name Std.Dev.
subject (Intercept) 1.8306
Residual 1.4352
1.8306^2 / (1.8306^2 + 1.4352^2)
multilevelTools::iccMixed(
dv = "y1",
id = c("subject"),
data = d
)
| Var | Sigma | ICC |
|---|---|---|
| <chr> | <dbl> | <dbl> |
| subject | 3.350987 | 0.6193062 |
| Residual | 2.059886 | 0.3806938 |
m1 <- lmer(y1 ~ time + (1 + time | subject), data = d)
summary(m1)
Linear mixed model fit by REML ['lmerMod']
Formula: y1 ~ time + (1 + time | subject)
Data: d
REML criterion at convergence: 5881.3
Scaled residuals:
Min 1Q Median 3Q Max
-3.03499 -0.46249 0.00414 0.48241 2.74992
Random effects:
Groups Name Variance Std.Dev. Corr
subject (Intercept) 1.0245 1.0122
time 1.0301 1.0149 0.15
Residual 0.2412 0.4911
Number of obs: 2000, groups: subject, 500
Fixed effects:
Estimate Std. Error t value
(Intercept) 0.50159 0.04885 10.267
time 0.25157 0.04644 5.417
Correlation of Fixed Effects:
(Intr)
time 0.072
1.02271^2 + 1.02403^2 + 0.48906^2
mix_mod <- lmer(y1 ~ time + (1 + time | subject), data = d)
summary(mix_mod)
Linear mixed model fit by REML ['lmerMod']
Formula: y1 ~ time + (1 + time | subject)
Data: d
REML criterion at convergence: 5881.3
Scaled residuals:
Min 1Q Median 3Q Max
-3.03499 -0.46249 0.00414 0.48241 2.74992
Random effects:
Groups Name Variance Std.Dev. Corr
subject (Intercept) 1.0245 1.0122
time 1.0301 1.0149 0.15
Residual 0.2412 0.4911
Number of obs: 2000, groups: subject, 500
Fixed effects:
Estimate Std. Error t value
(Intercept) 0.50159 0.04885 10.267
time 0.25157 0.04644 5.417
Correlation of Fixed Effects:
(Intr)
time 0.072
Gli effetti fissi che abbiamo ottenuto (\(\alpha\) = 0.50159, \(\beta\) = 0.25157) sono simili ai valori che abbiamo impostato nella simulazione per l’intercetta e la pendenza globale.
VarCorr(mix_mod)
Groups Name Std.Dev. Corr
subject (Intercept) 1.01217
time 1.01494 0.150
Residual 0.49108
Le varianze degli effetti casuali stimati (\(1.0122^2\), \(1.0149^2\)) sono molto simili al valore impostato di 1, la correlazione (0.15) è simile al valore impostato di 0.2 e la deviazione standard dei residui (0.4911) è simile al valore impostato di 0.5.
30.3. Modello di crescita latente#
Analizziamo ora gli stessi dati con un modello LGM. Possiamo pensare ai modelli LGM come ad un’estensione del modello CFA nel quale ipotizziamo la presenza di due fattori latenti: una variabile latente per le intercette casuali (ovvero, per la variazione delle intercette dei partecipanti) e una seconda variabile latente le pendenze casuali (ovvero, per la variazione delle pendenze dei partecipanti). Le intercette e le pendenze si riferiscono alla retta di regressione che, per ciascun partecipante, descrive la relazione tra la variabile esaminata e il tempo.
Dato che il modello deve spiegare la relazione tra le medie dei punteggi dei partecipanti in funzione del tempo, non possiamo usare in input la matrice di covarianza campionaria, ma dobbiamo invece utilizzare i dati grezzi (ovvero, le singole osservazioni per ciascun partecipante).
Come in precedenza, useremo lavaan, ma ora con una sintassi diversa, perché dobbiamo fissare le saturazioni fattoriali a valori specifici per implementare i vincoli del modello. Questo porta anche a un output non standard rispetto ad altri modelli SEM, poiché i parametri del modello saranno fissi non devono essere stimati.
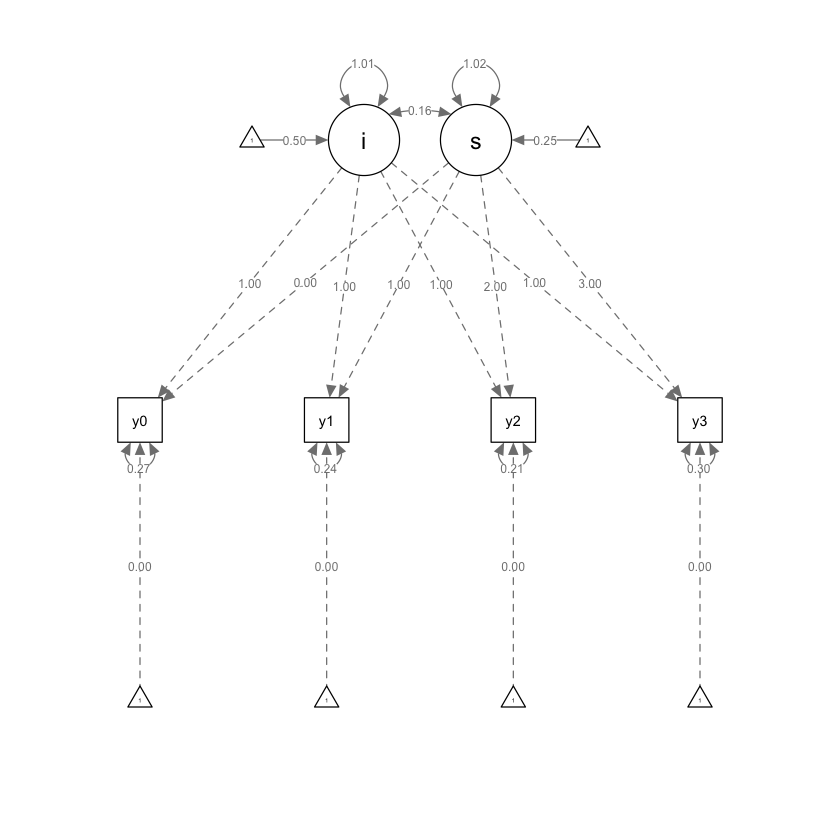
Per il fattore che rappresenta le intercette, i valori delle saturazioni fattoriali sono fissati a 1. Possiamo pensare al valore 1 come corrispondente alla colonna corrispondente all’intercetta nella matrice \(\boldsymbol{X}\) nel modello di regressione multipla.
Le saturazioni per il fattore che specifica le pendenze casuali sono fissate ai valori che descrivono la variazione temporale: qui i valori \(\lambda\) da 0 a 3 riflettono la spaziatura temporale tra le misurazioni \(y\). Iniziare la codifica da 0 consente di assegnare allo 0 un’interpretazione dotata di significato. Possiamo pensare ai valori 0, 1, 2, 3 come ai valori che, nella matrice \(\boldsymbol{X}\) nel modello di regressione multipla, corrispondono alla colonna associata alla pendenza del modello di regressione.
Il modello di crescita latente può dunque essere specificato dal seguente modello a variabili latenti:
dove
\(y_j\) è la variabile di interesse che cambia nel tempo, con \(j = 0, \dots, 3\).
\(\alpha_0\) rappresenta l’intercetta della retta di regressione al tempo \(t = 0\) (il punto di partenza della linea nera sopra).
\(\alpha_1 \lambda_j\) è il tasso medio di crescita nel tempo (la pendenza della linea nera nel grafico sopra). Qui \(\lambda_j\) è solo l’indice dei punti temporali considerati (0, 1, 2, 3).
\(\zeta_{00}\) è la varianza tra i soggetti nel punto \(t = 0\).
\(\zeta_{11} \lambda_j\) è la varianza del tasso di crescita tra i soggetti.
\(\epsilon_j\) è la varianza di ciascun soggetto attorno alla sua retta di regressione.
Un requisito degli LGM è che i dati devono essere forniti del formato wide (mentre per il precedente modello misto abbiamo usato il formato long), il che significa che ogni colonna rappresenta la variabile di esito in un diverso momento nel tempo. Si presume che ogni osservazione o riga sia indipendente dalle altre; le colonne mostrano invece una dipendenza temporale. Trasformiamo dunque i dati nel formato richiesto.
dwide <- d %>%
spread(time, y1) %>%
rename_at(vars(-subject), function(x) paste0("y", x))
head(dwide)
| subject | y0 | y1 | y2 | y3 | |
|---|---|---|---|---|---|
| <int> | <dbl> | <dbl> | <dbl> | <dbl> | |
| 1 | 1 | -0.5395258 | -1.1823659 | -2.2965593 | -3.173465 |
| 2 | 2 | -1.3232110 | -4.0664952 | -4.3738304 | -6.358334 |
| 3 | 3 | 0.8185443 | 1.0549470 | 2.0104678 | 3.531232 |
| 4 | 4 | 0.4469440 | -0.3162615 | -1.7896354 | -1.843919 |
| 5 | 5 | 1.8959902 | 5.5259110 | 9.6045869 | 12.546123 |
| 6 | 6 | 2.1829579 | 1.6287374 | -0.3136214 | -1.660328 |
Il modello misto che abbiamo descritto in precedenza corrisponde dunque ad un modello fattoriale con due variabili latenti: un fattore (\(\eta_0\)) che rappresenta il “punteggio vero” delle intercette individuali e un fattore (\(\eta_1\)) che rappresenta il “punteggio vero” delle pendenze delle rette di regressione per i singoli individui.
Nella sintassi di lavaan il modello diventa:
model <- "
i =~ 1*y0 + 1*y1 + 1*y2 + 1*y3
s =~ 0*y0 + 1*y1 + 2*y2 + 3*y3
"
Possiamo adattare il modello ai dati usando una funzione specifica di lavaan, ovvero growth, che può essere usata per questa classe di modelli.
growth_curve_model <- growth(model, data = dwide)
summary(growth_curve_model) |>
print()
lavaan 0.6.15 ended normally after 41 iterations
Estimator ML
Optimization method NLMINB
Number of model parameters 9
Number of observations 500
Model Test User Model:
Test statistic 4.212
Degrees of freedom 5
P-value (Chi-square) 0.519
Parameter Estimates:
Standard errors Standard
Information Expected
Information saturated (h1) model Structured
Latent Variables:
Estimate Std.Err z-value P(>|z|)
i =~
y0 1.000
y1 1.000
y2 1.000
y3 1.000
s =~
y0 0.000
y1 1.000
y2 2.000
y3 3.000
Covariances:
Estimate Std.Err z-value P(>|z|)
i ~~
s 0.162 0.051 3.137 0.002
Intercepts:
Estimate Std.Err z-value P(>|z|)
.y0 0.000
.y1 0.000
.y2 0.000
.y3 0.000
i 0.501 0.049 10.263 0.000
s 0.252 0.046 5.428 0.000
Variances:
Estimate Std.Err z-value P(>|z|)
.y0 0.268 0.042 6.356 0.000
.y1 0.237 0.022 10.713 0.000
.y2 0.209 0.029 7.262 0.000
.y3 0.299 0.066 4.556 0.000
i 1.007 0.078 12.996 0.000
s 1.021 0.068 14.953 0.000
Usiamo l’oggetto creato da growth per creare un diagramma di percorso.
semPaths(growth_curve_model, what = "path", whatLabels = "par")
30.4. Confronto tra modelli misti e LGM#
Nell’output, Intercepts corrisponde agli effetti fissi:
Intercepts:
Estimate Std.Err z-value P(>|z|)
i 0.510 0.048 10.542 0.000
s 0.234 0.046 5.133 0.000
Potrebbe sembrare strano chiamare gli effetti fissi ‘intercette’, ma ha senso se pensiamo al modello a crescita latente come al modello misto descritto in precedenza. Si noti che le stime ottenute sono molto simili a quelle fornite dal modello misto
print(fixef(mix_mod))
(Intercept) time
0.5015932 0.2515722
Si noti inoltre che le stime degli effetti fissi del modello misto sono identiche a quelle che vengono trovate usando un modello di regressione standard:
lm(y1 ~ time, data = d)
Call:
lm(formula = y1 ~ time, data = d)
Coefficients:
(Intercept) time
0.5016 0.2516
Consideriamo ora le stime della varianza nel modello a crescita latente.
Covariances:
Estimate Std.Err z-value P(>|z|)
i ~~
s 0.220 0.050 4.371 0.000
Variances:
Estimate Std.Err z-value P(>|z|)
.y0 0.310 0.042 7.308 0.000
.y1 0.220 0.021 10.338 0.000
.y2 0.230 0.029 7.935 0.000
.y3 0.275 0.064 4.295 0.000
i 0.973 0.076 12.854 0.000
s 0.986 0.066 14.889 0.000
Confrontiamo questi valori con quelli ottenuti dal modello misto.
VarCorr(mix_mod) |>
print()
Groups Name Std.Dev. Corr
subject (Intercept) 1.01217
time 1.01494 0.150
Residual 0.49108
Si noti che il modello a crescita latente, per impostazione predefinita, assume una varianza eterogenea per ogni rilevazione temporale. I modelli misti, invece, per impostazione predefinita assumono la stessa varianza per ogni punto temporale (anche se questo vincolo può essere allentato).
È però possibile specificare una stima separata della varianza nelle diverse rilevazioni temporali. Se però vincoliamo le varianze ad essere uguali per ciascuna rilevazione temporale nel modello LGM, i due modelli producono delle stime identiche.
La sintassi seguente viene utilizzata per forzare l’uguaglianza delle varianze in ciascuna rilevazione temporale.
model <- "
# intercept and slope with fixed coefficients
i =~ 1*y0 + 1*y1 + 1*y2 + 1*y3
s =~ 0*y0 + 1*y1 + 2*y2 + 3*y3
y0 ~~ resvar*y0
y1 ~~ resvar*y1
y2 ~~ resvar*y2
y3 ~~ resvar*y3
"
Adattiamo il nuovo modello ai dati.
growth_curve_model <- growth(model, data = dwide)
Esaminiamo i risultati.
summary(growth_curve_model) |>
print()
lavaan 0.6.15 ended normally after 27 iterations
Estimator ML
Optimization method NLMINB
Number of model parameters 9
Number of equality constraints 3
Number of observations 500
Model Test User Model:
Test statistic 6.180
Degrees of freedom 8
P-value (Chi-square) 0.627
Parameter Estimates:
Standard errors Standard
Information Expected
Information saturated (h1) model Structured
Latent Variables:
Estimate Std.Err z-value P(>|z|)
i =~
y0 1.000
y1 1.000
y2 1.000
y3 1.000
s =~
y0 0.000
y1 1.000
y2 2.000
y3 3.000
Covariances:
Estimate Std.Err z-value P(>|z|)
i ~~
s 0.154 0.051 3.034 0.002
Intercepts:
Estimate Std.Err z-value P(>|z|)
.y0 0.000
.y1 0.000
.y2 0.000
.y3 0.000
i 0.502 0.049 10.278 0.000
s 0.252 0.046 5.423 0.000
Variances:
Estimate Std.Err z-value P(>|z|)
.y0 (rsvr) 0.241 0.011 22.361 0.000
.y1 (rsvr) 0.241 0.011 22.361 0.000
.y2 (rsvr) 0.241 0.011 22.361 0.000
.y3 (rsvr) 0.241 0.011 22.361 0.000
i 1.022 0.076 13.502 0.000
s 1.028 0.068 15.095 0.000
Per lme4 abbiamo:
print(VarCorr(mix_mod), comp = "Var")
Groups Name Variance Cov
subject (Intercept) 1.02448
time 1.03011 0.154
Residual 0.24116
In entrambi i casi, la varianza residua è uguale a 0.241 e la correlazione tra intercette e pendenze casuali è uguale a 0.154.
Inoltre, le stime dei coefficienti casuali del modello misto sono identiche a quelle delle variabili latenti.
coef(mix_mod)[[1]] %>% head()
| (Intercept) | time | |
|---|---|---|
| <dbl> | <dbl> | |
| 1 | -0.3967486 | -0.9050565 |
| 2 | -1.5328622 | -1.5945199 |
| 3 | 0.5429873 | 0.8759706 |
| 4 | 0.3095727 | -0.7887484 |
| 5 | 2.0327226 | 3.5319068 |
| 6 | 2.0645454 | -1.1411935 |
lavPredict(growth_curve_model) %>%
head()
| i | s |
|---|---|
| -0.3966515 | -0.9050631 |
| -1.5324914 | -1.5946260 |
| 0.5430942 | 0.8759036 |
| 0.3094388 | -0.7886563 |
| 2.0328124 | 3.5317637 |
| 2.0637121 | -1.1407804 |
30.5. Conclusioni#
In conclusione, abbiamo visto che, nel caso più semplice, i modelli LGM producono risultati identici ai modelli misti. Tuttavia, la concettualizzazione del cambiamento nei termini di un modello a crescita latente offre molti vantaggi rispetto alla descrizione dei dati nei termini dei modelli misti in quanto i modelli LGM sono più flessibili e consentono la verifica di ipotesi statistiche che non possono essere esaminate nel constesto dei modelli misti.
