10. Il modello multifattoriale#
10.1. Modello multifattoriale: fattori ortogonali#
La teoria dei due fattori ha orientato per diversi anni le ricerche sull’intelligenza, finché Thurstone (1945) non propose una sua modifica, conosciuta come teoria multifattoriale. Secondo Thurstone la covariazione tra le variabili manifeste non può essere spiegata da un unico fattore generale. Invece è necessario ipotizzare l’azione causale di diversi fattori, definiti comuni, i quali si riferiscono solo ad alcune delle variabili considerate.
Il modello plurifattoriale assume che ciascuna variabile manifesta sia espressa come funzione lineare di un certo numero \(m\) di fattori comuni, \(\xi_1, \xi_2, \dots, \xi_m\), responsabili della correlazione con le altre variabili, e di un solo fattore specifico (termine d’errore), responsabile della variabilità della variabile stessa. Per \(p\) variabili manifeste, \(Y_1, Y_2, \dots, Y_p\), il modello fattoriale diventa quello indicato dal sistema di equazioni lineari descritto di seguito. Idealmente, \(m\) dovrebbe essere molto più piccolo di \(p\) così da consentire una descrizione parsimoniosa delle variabili manifeste in funzione di pochi fattori soggiacenti.
Le variabili manifeste \(Y\) sono indicizzate da \(i = 1, \dots, p.\) Le variabili latenti \(\xi\) (fattori) sono indicizzate da \(j = 1, \dots, m.\) I fattori specifici \(\delta\) sono indicizzati da \(i = 1, \dots, p.\) Le saturazioni fattoriali si distinguono dunque tramite due indici, \(i\) e \(j\): il primo indice si riferisce alle variabili manifeste, il secondo si riferisce ai fattori latenti.
Indichiamo con \(\mu_i\), con \(i=1, \dots, p\) le medie delle \(p\) variabili manifeste \(Y_1, Y_2, \dots, Y_p\). Se non vi è alcun effetto delle variabili comuni latenti, allora la variabile \(Y_{ijk}\), dove \(k\) è l’indice usato per i soggetti, sarà uguale a:
Se invece le variabili manifeste rappresentano la somma dell’effetto causale di \(m\) fattori comuni e di \(p\) fattori specifici, allora possiamo scrivere:
Nel precedente sistema di equazioni lineari,
\(\xi_j\), con \(j=1, \dots, m\), rappresenta la \(j\)-esima variabile inosservabile a fattore comune (ossia il \(j\)-esimo fattore comune a tutte le variabili \(Y_i\));
\(\lambda_{ij}\) rappresenta il parametro, detto saturazione o peso fattoriale, che riflette l’importanza del \(j\)-esimo fattore comune nella composizione della \(i\)-esima variabile osservabile;
\(\delta_i\) rappresenta il fattore specifico (o unico) di ogni variabile manifesta \(Y_i\).
In conclusione, secondo il modello multifattoriale, le variabili manifeste \(Y_i\), con \(i=1, \dots, p\), sono il risultato di una combinazione lineare di \(m < p\) fattori inosservabili ad esse comuni \(\xi_j\), con \(j=1, \dots, m\), e di \(p\) fattori specifici \(\delta_i\), con \(i=1, \dots, p\), anch’essi inosservabili e di natura residua.
10.1.1. Assunzioni del modello multifattoriale#
Le variabili inosservabili a fattore comune \(\xi_j\), con \(j=1, \dots, m\), in quanto latenti, non possiedono unità di misura. Pertanto, per semplicità si assume che abbiano media zero, \(\mathbb{E}(\xi_j)=0\), abbiano varianza unitaria, \(\mathbb{V} (\xi_j)= \mathbb{E}(\xi_j^2) - [\mathbb{E}(\xi_j)]^2=1\), e siano incorrelate tra loro, \(Cov(\xi_j, \xi_h)=0\), con \(j, h = 1, \dots, m; \;j \neq h\). Si assume inoltre che le variabili a fattore specifico \(\delta_i\) siano tra loro incorrelate, \(Cov(\delta_i,\delta_k)=0\), con \(i, k = 1, \dots, p, \; i \neq k\), abbiano media zero, \(\mathbb{E}(\delta_i)=0\), e varianza uguale a \(\mathbb{V} (\delta_i) = \psi_{ii}\). La varianza \(\psi_{ii}\) è detta varianza specifica o unicità della \(i\)-esima variabile manifesta \(Y_i\). Si assume infine che i fattori specifici siano linearmente incorrelati con i fattori comuni, ovvero \(Cov(\xi_j, \delta_i)=0\) per ogni \(j=1, \dots, m\) e per ogni \(i=1\dots,p\).
10.1.2. Interpretazione dei parametri del modello#
10.1.2.1. Covarianza tra variabili e fattori#
Nell’ipotesi che le variabili \(Y_i\) abbiano media nulla, la covarianza tra \(Y_i\) e \(\xi_j\) è uguale alla saturazione fattoriale \(\lambda_{ij}\):
Anche nel modello multifattoriale, dunque, le saturazioni fattoriali rappresentano le covarianze tra le variabili e i fattori:
Naturalmente, se le variabili sono standardizzate, le saturazioni fattoriali diventano correlazioni:
10.1.2.2. Espressione fattoriale della varianza#
Come nel modello monofattoriale, la varianza delle variabili manifeste si decompone in una componente dovuta ai fattori comuni, chiamata comunalità, e in una componente specifica alle \(Y_i\), chiamata unicità. Nell’ipotesi che le variabili \(Y_i\) abbiano media nulla, la varianza di \(Y_i\) è uguale a
Come si sviluppa il polinomio precedente? Il quadrato di un polinomio è uguale alla somma dei quadrati di tutti i termini più il doppio prodotto di ogni termine per ciascuno di quelli che lo seguono. Il valore atteso del quadrato del primo termine è uguale a \(\lambda_{i1}^2\mathbb{E}(\xi_1^2)\) ma, essendo la varianza di \(\xi_1\) uguale a \(1\), otteniamo semplicemente \(\lambda_{i1}^2\). Lo stesso vale per i quadrati di tutti i termini seguenti tranne l’ultimo. Infatti, \(\mathbb{E}(\delta_i^2)=\psi_{ii}\). Per quel che riguarda i doppi prodotti, sono tutti nulli. In primo luogo perché, nel caso di fattori ortogonali, la covarianza tra i fattori comuni è nulla, \(\mathbb{E}(\xi_j \xi_h)=0\), con \(j \neq h\). In secondo luogo perché il fattori comuni cono incorrelati con i fattori specifici, quindi \(\mathbb{E}(\delta_i \xi_j)=0\).
In conclusione,
la varianza della variabile manifesta \(Y_i\) è suddivisa in due parti: il primo addendo è definito comunalità poiché rappresenta la parte di variabilità della \(Y_i\) spiegata dai fattori comuni; il secondo addendo è invece definito varianza specifica (o unicità) poiché esprime la parte di variabilità della \(Y_i\) non spiegata dai fattori comuni.
10.1.2.3. Espressione fattoriale della covarianza#
Quale esempio, consideriamo il caso di \(p=5\) variabili osservabili e \(m=2\) fattori ortogonali. Se le variabili manifeste sono ‘centrate’ (ovvero, se a ciascuna di esse sottraiamo la rispettiva media), allora il modello multifattoriale diventa
Nell’ipotesi che le variabili \(Y_i\) abbiano media nulla, la covarianza tra \(Y_1\) e \(Y_2\), ad esempio, è uguale a:
In conclusione, la covarianza tra le variabili manifeste \(Y_l\) e \(Y_m\) riprodotta dal modello è data dalla somma dei prodotti delle saturazioni \(\lambda_l \lambda_m\) nei due fattori.
Esempio. Consideriamo i dati riportati da Brown [Bro15], ovvero otto misure di personalità raccolte su un campione di 250 pazienti che hanno concluso un programma di psicoterapia. Le scale sono le seguenti:
anxiety (N1),
hostility (N2),
depression (N3),
self-consciousness (N4),
warmth (E1),
gregariousness (E2),
assertiveness (E3),
positive emotions (E4).
varnames <- c("N1", "N2", "N3", "N4", "E1", "E2", "E3", "E4")
sds <- '5.7 5.6 6.4 5.7 6.0 6.2 5.7 5.6'
cors <- '
1.000
0.767 1.000
0.731 0.709 1.000
0.778 0.738 0.762 1.000
-0.351 -0.302 -0.356 -0.318 1.000
-0.316 -0.280 -0.300 -0.267 0.675 1.000
-0.296 -0.289 -0.297 -0.296 0.634 0.651 1.000
-0.282 -0.254 -0.292 -0.245 0.534 0.593 0.566 1.000'
psychot_cor_mat <- getCov(cors, names = varnames)
n <- 250
Eseguiamo l’analisi fattoriale esplorativa con il metodo della massima verosimiglianza ipotizzando due fattori comuni incorrelati:
n_facs <- 2
fit_efa <- factanal(
covmat = psychot_cor_mat,
factors = n_facs,
rotation = "varimax",
n.obs = n
)
Esaminiamo le saturazioni fattoriali:
lambda <- fit_efa$loadings
print(lambda)
Loadings:
Factor1 Factor2
N1 0.854 -0.228
N2 0.826 -0.194
N3 0.811 -0.233
N4 0.865 -0.186
E1 -0.202 0.773
E2 -0.139 0.829
E3 -0.158 0.771
E4 -0.147 0.684
Factor1 Factor2
SS loadings 2.923 2.526
Proportion Var 0.365 0.316
Cumulative Var 0.365 0.681
La soluzione fattoriale conferma la presenza di due fattori: il primo fattore satura sulle scale di neutoricismo, il secono sulle scale di estroversione.
La correlazione riprodotta \(r_{12}\) è uguale a \(\lambda_{11}\lambda_{21} + \lambda_{12}\lambda_{22}\)
lambda[1, 1] * lambda[2, 1] + lambda[1, 2] * lambda[2, 2]
e corrisponde da vicino alla correlazione osservata 0.767.
L’intera matrice di correlazioni riprodotte è \(\boldsymbol{\Lambda} \boldsymbol{\Lambda}^{\mathsf{T}} + \boldsymbol{\psi}\):
Rr <- lambda %*% t(lambda) + diag(fit_efa$uniq)
Rr %>%
round(3)
| N1 | N2 | N3 | N4 | E1 | E2 | E3 | E4 | |
|---|---|---|---|---|---|---|---|---|
| N1 | 1.000 | 0.749 | 0.745 | 0.781 | -0.348 | -0.307 | -0.311 | -0.281 |
| N2 | 0.749 | 1.000 | 0.715 | 0.751 | -0.317 | -0.276 | -0.281 | -0.254 |
| N3 | 0.745 | 0.715 | 1.000 | 0.745 | -0.344 | -0.306 | -0.308 | -0.279 |
| N4 | 0.781 | 0.751 | 0.745 | 1.000 | -0.318 | -0.274 | -0.280 | -0.254 |
| E1 | -0.348 | -0.317 | -0.344 | -0.318 | 1.000 | 0.669 | 0.628 | 0.558 |
| E2 | -0.307 | -0.276 | -0.306 | -0.274 | 0.669 | 1.000 | 0.661 | 0.587 |
| E3 | -0.311 | -0.281 | -0.308 | -0.280 | 0.628 | 0.661 | 1.000 | 0.550 |
| E4 | -0.281 | -0.254 | -0.279 | -0.254 | 0.558 | 0.587 | 0.550 | 1.000 |
La differenza tra la matrice di correlazioni riprodotte e la matrice di correlazioni osservate è uguale a:
(psychot_cor_mat - Rr) %>%
round(3)
| N1 | N2 | N3 | N4 | E1 | E2 | E3 | E4 | |
|---|---|---|---|---|---|---|---|---|
| N1 | 0.000 | 0.018 | -0.014 | -0.003 | -0.003 | -0.009 | 0.015 | -0.001 |
| N2 | 0.018 | 0.000 | -0.006 | -0.013 | 0.015 | -0.004 | -0.008 | 0.000 |
| N3 | -0.014 | -0.006 | 0.000 | 0.017 | -0.012 | 0.006 | 0.011 | -0.013 |
| N4 | -0.003 | -0.013 | 0.017 | 0.000 | 0.000 | 0.007 | -0.016 | 0.009 |
| E1 | -0.003 | 0.015 | -0.012 | 0.000 | 0.000 | 0.006 | 0.006 | -0.024 |
| E2 | -0.009 | -0.004 | 0.006 | 0.007 | 0.006 | 0.000 | -0.010 | 0.006 |
| E3 | 0.015 | -0.008 | 0.011 | -0.016 | 0.006 | -0.010 | 0.000 | 0.016 |
| E4 | -0.001 | 0.000 | -0.013 | 0.009 | -0.024 | 0.006 | 0.016 | 0.000 |
Esempio. Consideriamo nuovamente i dati precedenti ma, in questo caso, eseguiamo un’analisi fattoriale confermativa. Usando lavaan il modello diventa:
cfa_mod <- "
N =~ N1 + N2 + N3 + N4
E =~ E1 + E2 + E3 + E4
"
fit_cfa <- lavaan::cfa(
cfa_mod,
sample.cov = psychot_cor_mat,
sample.nobs = n,
orthogonal = TRUE,
std.lv = TRUE
)
Il path diagram si ottiene nel modo seguente:
semPaths(
fit_cfa,
"std",
posCol = c("black"),
edge.label.cex = 1.2,
sizeMan = 7
)
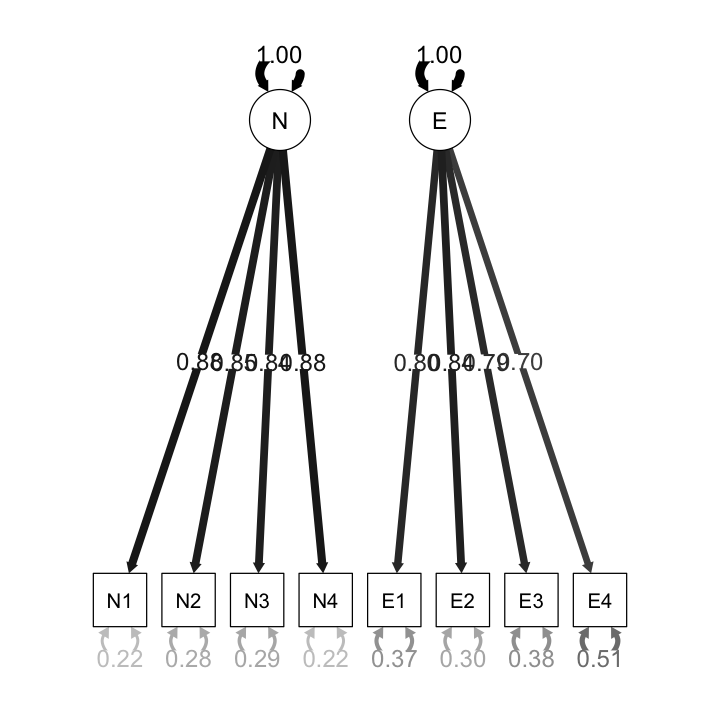
Esaminiamo le saturazioni fattoriali:
parameterEstimates(fit_cfa, standardized = TRUE) %>%
dplyr::filter(op == "=~") %>%
dplyr::select(
"Latent Factor" = lhs,
Indicator = rhs,
B = est,
SE = se,
Z = z,
"p-value" = pvalue,
Beta = std.all) %>%
knitr::kable(digits = 3, booktabs = TRUE, format = "markdown",
caption = "Factor Loadings")
Table: Factor Loadings
|Latent Factor |Indicator | B| SE| Z| p-value| Beta|
|:-------------|:---------|-----:|-----:|------:|-------:|-----:|
|N |N1 | 0.882| 0.051| 17.422| 0| 0.884|
|N |N2 | 0.847| 0.052| 16.340| 0| 0.849|
|N |N3 | 0.840| 0.052| 16.134| 0| 0.842|
|N |N4 | 0.882| 0.051| 17.432| 0| 0.884|
|E |E1 | 0.795| 0.056| 14.276| 0| 0.796|
|E |E2 | 0.838| 0.054| 15.369| 0| 0.839|
|E |E3 | 0.788| 0.056| 14.097| 0| 0.789|
|E |E4 | 0.697| 0.058| 11.942| 0| 0.699|
Il risultato sembra sensato: le saturazioni su ciascun fattore sono molto alte. Tuttavia, la matrice delle correlazioni residue
cor_table <- residuals(fit_cfa, type = "cor")$cov
knitr::kable(
cor_table,
digits = 3,
format = "markdown",
booktabs = TRUE
)
| | N1| N2| N3| N4| E1| E2| E3| E4|
|:--|------:|------:|------:|------:|------:|------:|------:|------:|
|N1 | 0.000| 0.017| -0.013| -0.003| -0.351| -0.316| -0.296| -0.282|
|N2 | 0.017| 0.000| -0.006| -0.012| -0.302| -0.280| -0.289| -0.254|
|N3 | -0.013| -0.006| 0.000| 0.018| -0.356| -0.300| -0.297| -0.292|
|N4 | -0.003| -0.012| 0.018| 0.000| -0.318| -0.267| -0.296| -0.245|
|E1 | -0.351| -0.302| -0.356| -0.318| 0.000| 0.007| 0.006| -0.022|
|E2 | -0.316| -0.280| -0.300| -0.267| 0.007| 0.000| -0.011| 0.007|
|E3 | -0.296| -0.289| -0.297| -0.296| 0.006| -0.011| 0.000| 0.015|
|E4 | -0.282| -0.254| -0.292| -0.245| -0.022| 0.007| 0.015| 0.000|
rivela che il modello ipotizzato dall’analisi fattoriale confermativa non è adeguato.
10.2. Modello fattoriale: Fattori obliqui#
Anche nel caso di fattori comuni correlati è possibile esprimere nei termini dei parametri del modello la covarianza teorica tra una variabile manifesta \(Y_i\) e uno dei fattori comuni, la covarianza teorica tra due variabili manifeste, e la comunalità di ciascuna variabile manifesta. Dato però che i fattori comuni risultano correlati, l’espressione fattoriale di tali quantità è più complessa che nel caso di fattori comuni ortogonali.
10.2.1. Covarianza teorica tra variabili e fattori#
In base al modello multifattoriale con \(m\) fattori comuni la variabile \(Y_i\) è
Poniamoci il problema di trovare la covarianza teorica tra la variabile manifesta \(Y_i\) e il fattore comune \(\xi_j\). Come in precedenza, il problema si riduce a quello di trovare \(\mathbb{E}(Y_i \xi_j)\). Ne segue che
Ad esempio, nel caso di tre fattori comuni \(\xi_1, \xi_2, \xi_3\), la covarianza tra \(Y_1\) e \(\xi_{1}\) diventa
10.2.2. Espressione fattoriale della varianza#
Poniamoci ora il problema di trovare la varianza teorica della variabile manifesta \(Y_i\). In base al modello fattoriale, la variabile \(Y_i\) è specificata come nella @ref(eq:mod-multifact). La varianza di \(Y_i\) è \(\mathbb{V}(Y_i) = \mathbb{E}(Y_i^2) -[\mathbb{E}(Y_i)]^2\). Però, avendo espresso \(Y_i\) nei termini della differenza dalla sua media, l’espressione della varianza si riduce a \(\mathbb{V}(Y_i) = \mathbb{E}(Y_i^2)\). Dobbiamo dunque sviluppare l’espressione
In conclusione, la varianza teorica di \(Y_i\) è uguale a
Ad esempio, nel caso di tre fattori comuni, \(\xi_1, \xi_2, \xi_3\), la varianza di \(Y_1\) è
10.2.3. Covarianza teorica tra due variabili#
Consideriamo ora il caso più semplice di due soli fattori comuni correlati e calcoliamo la covarianza tra \(Y_1\) e \(Y_2\):
Distribuendo l’operatore di valore atteso, dato che \(\mathbb{E}(\xi^2)=1\) e \(\mathbb{E}(\xi \delta)=0\), otteniamo
In termini matriciali si scrive
dove \(\boldsymbol{\Phi}\) è la matrice di ordine \(m \times m\) di varianze e covarianze tra i fattori comuni e \(\boldsymbol{\Psi}\) è una matrice diagonale di ordine \(p\) con le unicità delle variabili.
Esempio. Consideriamo nuovamente i dati esaminati negli esercizi precedenti, ma questa volta il modello consente una correlazione tra i due fattori comuni:
fit2_cfa <- lavaan::cfa(
cfa_mod,
sample.cov = psychot_cor_mat,
sample.nobs = n,
orthogonal = FALSE,
std.lv = TRUE
)
Visualizziamo il modello nel modo seguente:
semPaths(
fit2_cfa,
"std",
posCol = c("black"),
edge.label.cex = 1.1,
sizeMan = 7
)
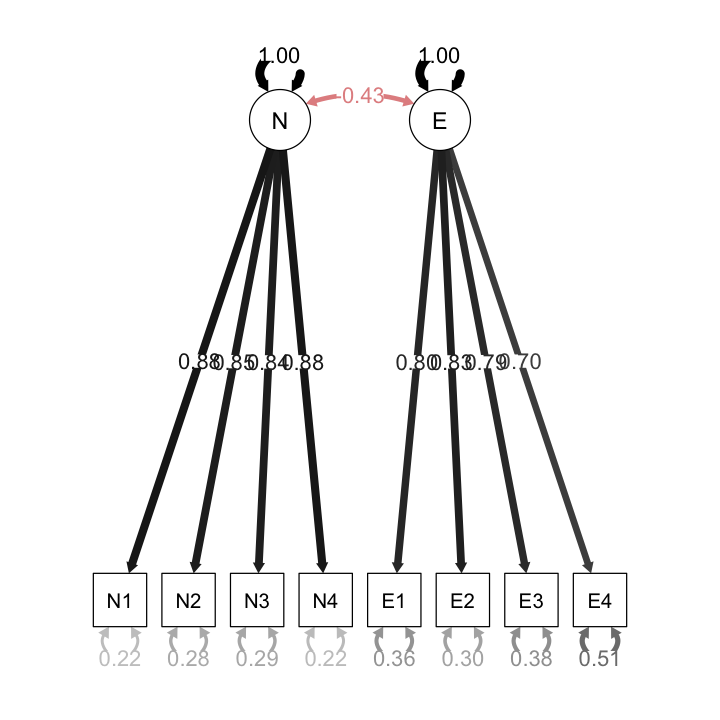
Esaminiamo le saturazioni fattoriali:
parameterEstimates(fit2_cfa, standardized = TRUE) %>%
dplyr::filter(op == "=~") %>%
dplyr::select(
"Latent Factor" = lhs,
Indicator = rhs,
B = est,
SE = se,
Z = z,
"p-value" = pvalue,
Beta = std.all) %>%
knitr::kable(digits = 3, booktabs = TRUE, format = "markdown",
caption = "Factor Loadings")
Table: Factor Loadings
|Latent Factor |Indicator | B| SE| Z| p-value| Beta|
|:-------------|:---------|-----:|-----:|------:|-------:|-----:|
|N |N1 | 0.883| 0.051| 17.472| 0| 0.885|
|N |N2 | 0.847| 0.052| 16.337| 0| 0.849|
|N |N3 | 0.842| 0.052| 16.190| 0| 0.844|
|N |N4 | 0.880| 0.051| 17.381| 0| 0.882|
|E |E1 | 0.800| 0.055| 14.465| 0| 0.802|
|E |E2 | 0.832| 0.054| 15.294| 0| 0.834|
|E |E3 | 0.788| 0.056| 14.150| 0| 0.789|
|E |E4 | 0.698| 0.058| 11.974| 0| 0.699|
Le saturazioni sono simili a quelle che abbiamo trovato in precedenza, In questo caso, però, la matrice delle correlazioni residue è adeguata:
cor_table <- residuals(fit2_cfa, type = "cor")$cov
knitr::kable(
cor_table,
digits = 3,
format = "markdown",
booktabs = TRUE
)
| | N1| N2| N3| N4| E1| E2| E3| E4|
|:--|------:|------:|------:|------:|------:|------:|------:|------:|
|N1 | 0.000| 0.016| -0.015| -0.002| -0.042| 0.005| 0.008| -0.013|
|N2 | 0.016| 0.000| -0.007| -0.010| -0.006| 0.028| 0.002| 0.004|
|N3 | -0.015| -0.007| 0.000| 0.018| -0.062| 0.006| -0.007| -0.035|
|N4 | -0.002| -0.010| 0.018| 0.000| -0.010| 0.053| 0.007| 0.023|
|E1 | -0.042| -0.006| -0.062| -0.010| 0.000| 0.006| 0.001| -0.027|
|E2 | 0.005| 0.028| 0.006| 0.053| 0.006| 0.000| -0.007| 0.010|
|E3 | 0.008| 0.002| -0.007| 0.007| 0.001| -0.007| 0.000| 0.014|
|E4 | -0.013| 0.004| -0.035| 0.023| -0.027| 0.010| 0.014| 0.000|
Esempio. Esaminiamo più da vicino la matrice di correlazioni riprodotta dal modello, nel caso di fattori obliqui. Le saturazioni fattoriali sono:
lambda <- inspect(fit2_cfa, what="std")$lambda
lambda
| N | E | |
|---|---|---|
| N1 | 0.8848214 | 0.0000000 |
| N2 | 0.8485128 | 0.0000000 |
| N3 | 0.8436432 | 0.0000000 |
| N4 | 0.8819736 | 0.0000000 |
| E1 | 0.0000000 | 0.8018485 |
| E2 | 0.0000000 | 0.8337599 |
| E3 | 0.0000000 | 0.7894530 |
| E4 | 0.0000000 | 0.6990366 |
La matrice di intercorrelazoni fattoriali è
Phi <- inspect(fit2_cfa, what="std")$psi
Phi
| N | E | |
|---|---|---|
| N | 1.000000 | -0.434962 |
| E | -0.434962 | 1.000000 |
Le varianze residue sono:
Psi <- inspect(fit2_cfa, what="std")$theta
Psi
| N1 | N2 | N3 | N4 | E1 | E2 | E3 | E4 | |
|---|---|---|---|---|---|---|---|---|
| N1 | 0.217091 | 0.0000000 | 0.0000000 | 0.0000000 | 0.000000 | 0.0000000 | 0.000000 | 0.0000000 |
| N2 | 0.000000 | 0.2800261 | 0.0000000 | 0.0000000 | 0.000000 | 0.0000000 | 0.000000 | 0.0000000 |
| N3 | 0.000000 | 0.0000000 | 0.2882661 | 0.0000000 | 0.000000 | 0.0000000 | 0.000000 | 0.0000000 |
| N4 | 0.000000 | 0.0000000 | 0.0000000 | 0.2221225 | 0.000000 | 0.0000000 | 0.000000 | 0.0000000 |
| E1 | 0.000000 | 0.0000000 | 0.0000000 | 0.0000000 | 0.357039 | 0.0000000 | 0.000000 | 0.0000000 |
| E2 | 0.000000 | 0.0000000 | 0.0000000 | 0.0000000 | 0.000000 | 0.3048445 | 0.000000 | 0.0000000 |
| E3 | 0.000000 | 0.0000000 | 0.0000000 | 0.0000000 | 0.000000 | 0.0000000 | 0.376764 | 0.0000000 |
| E4 | 0.000000 | 0.0000000 | 0.0000000 | 0.0000000 | 0.000000 | 0.0000000 | 0.000000 | 0.5113478 |
Mediante i parametri del modello la matrice di correlazione si riproduce nel modo seguente:
In \(\textsf{R}\) scriviamo:
R_hat <- lambda %*% Phi %*% t(lambda) + Psi
R_hat %>%
round(3)
| N1 | N2 | N3 | N4 | E1 | E2 | E3 | E4 | |
|---|---|---|---|---|---|---|---|---|
| N1 | 1.000 | 0.751 | 0.746 | 0.780 | -0.309 | -0.321 | -0.304 | -0.269 |
| N2 | 0.751 | 1.000 | 0.716 | 0.748 | -0.296 | -0.308 | -0.291 | -0.258 |
| N3 | 0.746 | 0.716 | 1.000 | 0.744 | -0.294 | -0.306 | -0.290 | -0.257 |
| N4 | 0.780 | 0.748 | 0.744 | 1.000 | -0.308 | -0.320 | -0.303 | -0.268 |
| E1 | -0.309 | -0.296 | -0.294 | -0.308 | 1.000 | 0.669 | 0.633 | 0.561 |
| E2 | -0.321 | -0.308 | -0.306 | -0.320 | 0.669 | 1.000 | 0.658 | 0.583 |
| E3 | -0.304 | -0.291 | -0.290 | -0.303 | 0.633 | 0.658 | 1.000 | 0.552 |
| E4 | -0.269 | -0.258 | -0.257 | -0.268 | 0.561 | 0.583 | 0.552 | 1.000 |
Le correlazioni residue sono:
(psychot_cor_mat - R_hat) %>%
round(3)
| N1 | N2 | N3 | N4 | E1 | E2 | E3 | E4 | |
|---|---|---|---|---|---|---|---|---|
| N1 | 0.000 | 0.016 | -0.015 | -0.002 | -0.042 | 0.005 | 0.008 | -0.013 |
| N2 | 0.016 | 0.000 | -0.007 | -0.010 | -0.006 | 0.028 | 0.002 | 0.004 |
| N3 | -0.015 | -0.007 | 0.000 | 0.018 | -0.062 | 0.006 | -0.007 | -0.035 |
| N4 | -0.002 | -0.010 | 0.018 | 0.000 | -0.010 | 0.053 | 0.007 | 0.023 |
| E1 | -0.042 | -0.006 | -0.062 | -0.010 | 0.000 | 0.006 | 0.001 | -0.027 |
| E2 | 0.005 | 0.028 | 0.006 | 0.053 | 0.006 | 0.000 | -0.007 | 0.010 |
| E3 | 0.008 | 0.002 | -0.007 | 0.007 | 0.001 | -0.007 | 0.000 | 0.014 |
| E4 | -0.013 | 0.004 | -0.035 | 0.023 | -0.027 | 0.010 | 0.014 | 0.000 |
Questo risultato riproduce ciò che abbiamo trovato estraendo la matrice di correlazioni residue dall’oggetto creato dal lavaan::cfa mediante l’istruzione residuals(fit2_cfa, type = "cor")$cov.
Per fare un esempio relativo alla correlazione tra due indicatori, calcoliamo la correlazione predetta dal modello tra le variabili \(Y_1\) e \(Y_2\):
lambda[1, 1] * lambda[2, 1] + lambda[1, 2] * lambda[2, 2] +
lambda[1, 1] * lambda[2, 2] * Phi[1, 2] +
lambda[1, 2] * lambda[2, 1] * Phi[1, 2]
Questo valore si avvicina al valore contenuto dell’elemento (1, 2) della matrice di correlazioni osservate:
psychot_cor_mat[1, 2]
Usando le funzonalità di lavaan la matrice di correlazione predetta si ottiene con:
fitted(fit2_cfa)$cov |>
print()
N1 N2 N3 N4 E1 E2 E3 E4
N1 0.996
N2 0.748 0.996
N3 0.743 0.713 0.996
N4 0.777 0.745 0.741 0.996
E1 -0.307 -0.295 -0.293 -0.306 0.996
E2 -0.320 -0.306 -0.305 -0.319 0.666 0.996
E3 -0.303 -0.290 -0.289 -0.302 0.630 0.656 0.996
E4 -0.268 -0.257 -0.255 -0.267 0.558 0.580 0.550 0.996
La matrice dei residui è
resid(fit2_cfa)$cov |>
print()
N1 N2 N3 N4 E1 E2 E3 E4
N1 0.000
N2 0.016 0.000
N3 -0.015 -0.007 0.000
N4 -0.002 -0.010 0.018 0.000
E1 -0.042 -0.006 -0.062 -0.010 0.000
E2 0.005 0.028 0.006 0.053 0.006 0.000
E3 0.008 0.002 -0.007 0.007 0.001 -0.007 0.000
E4 -0.013 0.004 -0.035 0.023 -0.026 0.010 0.014 0.000
La matrice dei residui standardizzati è
resid(fit2_cfa, type = "standardized")$cov |>
print()
N1 N2 N3 N4 E1 E2 E3 E4
N1 0.000
N2 1.674 0.000
N3 -1.769 -0.569 0.000
N4 -0.350 -1.152 1.746 0.000
E1 -1.214 -0.161 -1.646 -0.294 0.000
E2 0.154 0.794 0.168 1.626 0.637 0.000
E3 0.219 0.062 -0.191 0.193 0.075 -0.693 0.000
E4 -0.314 0.092 -0.824 0.552 -1.481 0.624 0.690 0.000
I valori precedenti possono essere considerati come punti z, dove i valori con un valore assoluto maggiore di 2 possono essere ritenuti problematici. Tuttavia, è importante considerare che in questo modo si stanno eseguendo molteplici confronti, pertanto, si dovrebbe considerare l’opportunità di applicare una qualche forma di correzione per i confronti multipli.
## EFA con `lavaan`
Una funzionalità sperimentale di `lavaan` (ancora non ufficiale) è quella che consente di svolgere l'analisi fattoriale esplorativa con la funzione `efa()`. Consideriamo nuovamente i dati di @brown2015confirmatory, ovvero otto misure di personalità raccolte su un campione di 250 pazienti che hanno concluso un programma di psicoterapia.
Definiamo un modello ad un solo fattore comune.
# 1-factor model
f1 <- '
efa("efa")*f1 =~ N1 + N2 + N3 + N4 + E1 + E2 + E3 + E4
'
Definiamo un modello con due fattori comuni.
# 2-factor model
f2 <- '
efa("efa")*f1 +
efa("efa")*f2 =~ N1 + N2 + N3 + N4 + E1 + E2 + E3 + E4
'
Adattiamo ai dati il modello ad un fattore comune.
efa_f1 <-
cfa(
model = f1,
sample.cov = psychot_cor_mat,
sample.nobs = 250,
rotation = "oblimin"
)
Esaminiamo la soluzione ottenuta.
summary(
efa_f1,
fit.measures = TRUE,
standardized = TRUE,
rsquare = TRUE
) |>
print()
lavaan 0.6.15 ended normally after 2 iterations
Estimator ML
Optimization method NLMINB
Number of model parameters 16
Rotation method OBLIMIN OBLIQUE
Oblimin gamma 0
Rotation algorithm (rstarts) GPA (30)
Standardized metric TRUE
Row weights None
Number of observations 250
Model Test User Model:
Test statistic 375.327
Degrees of freedom 20
P-value (Chi-square) 0.000
Model Test Baseline Model:
Test statistic 1253.791
Degrees of freedom 28
P-value 0.000
User Model versus Baseline Model:
Comparative Fit Index (CFI) 0.710
Tucker-Lewis Index (TLI) 0.594
Loglikelihood and Information Criteria:
Loglikelihood user model (H0) -2394.637
Loglikelihood unrestricted model (H1) -2206.974
Akaike (AIC) 4821.275
Bayesian (BIC) 4877.618
Sample-size adjusted Bayesian (SABIC) 4826.897
Root Mean Square Error of Approximation:
RMSEA 0.267
90 Percent confidence interval - lower 0.243
90 Percent confidence interval - upper 0.291
P-value H_0: RMSEA <= 0.050 0.000
P-value H_0: RMSEA >= 0.080 1.000
Standardized Root Mean Square Residual:
SRMR 0.187
Parameter Estimates:
Standard errors Standard
Information Expected
Information saturated (h1) model Structured
Latent Variables:
Estimate Std.Err z-value P(>|z|) Std.lv Std.all
f1 =~ efa
N1 0.879 0.051 17.333 0.000 0.879 0.880
N2 0.841 0.052 16.154 0.000 0.841 0.842
N3 0.841 0.052 16.175 0.000 0.841 0.843
N4 0.870 0.051 17.065 0.000 0.870 0.872
E1 -0.438 0.062 -7.041 0.000 -0.438 -0.439
E2 -0.398 0.063 -6.327 0.000 -0.398 -0.398
E3 -0.398 0.063 -6.342 0.000 -0.398 -0.399
E4 -0.364 0.063 -5.746 0.000 -0.364 -0.364
Variances:
Estimate Std.Err z-value P(>|z|) Std.lv Std.all
.N1 0.224 0.028 7.915 0.000 0.224 0.225
.N2 0.289 0.033 8.880 0.000 0.289 0.290
.N3 0.288 0.032 8.866 0.000 0.288 0.289
.N4 0.239 0.029 8.174 0.000 0.239 0.240
.E1 0.804 0.073 10.963 0.000 0.804 0.807
.E2 0.838 0.076 11.008 0.000 0.838 0.841
.E3 0.837 0.076 11.007 0.000 0.837 0.841
.E4 0.864 0.078 11.041 0.000 0.864 0.867
f1 1.000 1.000 1.000
R-Square:
Estimate
N1 0.775
N2 0.710
N3 0.711
N4 0.760
E1 0.193
E2 0.159
E3 0.159
E4 0.133
Adattiamo ai dati il modello a due fattori comuni.
efa_f2 <-
cfa(
model = f2,
sample.cov = psychot_cor_mat,
sample.nobs = 250,
rotation = "oblimin"
)
Esaminiamo la soluzione ottenuta.
summary(
efa_f2,
fit.measures = TRUE,
standardized = TRUE,
rsquare = TRUE
) |>
print()
lavaan 0.6.15 ended normally after 1 iteration
Estimator ML
Optimization method NLMINB
Number of model parameters 23
Rotation method OBLIMIN OBLIQUE
Oblimin gamma 0
Rotation algorithm (rstarts) GPA (30)
Standardized metric TRUE
Row weights None
Number of observations 250
Model Test User Model:
Test statistic 9.811
Degrees of freedom 13
P-value (Chi-square) 0.709
Model Test Baseline Model:
Test statistic 1253.791
Degrees of freedom 28
P-value 0.000
User Model versus Baseline Model:
Comparative Fit Index (CFI) 1.000
Tucker-Lewis Index (TLI) 1.006
Loglikelihood and Information Criteria:
Loglikelihood user model (H0) -2211.879
Loglikelihood unrestricted model (H1) -2206.974
Akaike (AIC) 4469.758
Bayesian (BIC) 4550.752
Sample-size adjusted Bayesian (SABIC) 4477.840
Root Mean Square Error of Approximation:
RMSEA 0.000
90 Percent confidence interval - lower 0.000
90 Percent confidence interval - upper 0.048
P-value H_0: RMSEA <= 0.050 0.957
P-value H_0: RMSEA >= 0.080 0.001
Standardized Root Mean Square Residual:
SRMR 0.010
Parameter Estimates:
Standard errors Standard
Information Expected
Information saturated (h1) model Structured
Latent Variables:
Estimate Std.Err z-value P(>|z|) Std.lv Std.all
f1 =~ efa
N1 0.874 0.053 16.592 0.000 0.874 0.876
N2 0.851 0.055 15.551 0.000 0.851 0.853
N3 0.826 0.054 15.179 0.000 0.826 0.828
N4 0.896 0.053 16.802 0.000 0.896 0.898
E1 -0.046 0.040 -1.138 0.255 -0.046 -0.046
E2 0.035 0.034 1.030 0.303 0.035 0.035
E3 0.000 0.040 0.010 0.992 0.000 0.000
E4 -0.006 0.049 -0.131 0.896 -0.006 -0.006
f2 =~ efa
N1 -0.017 0.032 -0.539 0.590 -0.017 -0.017
N2 0.011 0.035 0.322 0.748 0.011 0.011
N3 -0.035 0.036 -0.949 0.343 -0.035 -0.035
N4 0.031 0.031 0.994 0.320 0.031 0.031
E1 0.776 0.059 13.125 0.000 0.776 0.778
E2 0.854 0.058 14.677 0.000 0.854 0.855
E3 0.785 0.060 13.106 0.000 0.785 0.787
E4 0.695 0.063 10.955 0.000 0.695 0.697
Covariances:
Estimate Std.Err z-value P(>|z|) Std.lv Std.all
f1 ~~
f2 -0.432 0.059 -7.345 0.000 -0.432 -0.432
Variances:
Estimate Std.Err z-value P(>|z|) Std.lv Std.all
.N1 0.218 0.028 7.790 0.000 0.218 0.219
.N2 0.279 0.032 8.693 0.000 0.279 0.280
.N3 0.287 0.032 8.907 0.000 0.287 0.289
.N4 0.216 0.029 7.578 0.000 0.216 0.217
.E1 0.361 0.044 8.226 0.000 0.361 0.362
.E2 0.292 0.043 6.787 0.000 0.292 0.293
.E3 0.379 0.046 8.315 0.000 0.379 0.381
.E4 0.509 0.053 9.554 0.000 0.509 0.511
f1 1.000 1.000 1.000
f2 1.000 1.000 1.000
R-Square:
Estimate
N1 0.781
N2 0.720
N3 0.711
N4 0.783
E1 0.638
E2 0.707
E3 0.619
E4 0.489
Anche se abbiamo introdotto finora soltanto la misura di bontà di adattamento del chi-quadrato, aggiungiamo qui il calcolo di altre misure di bontà di adattamento che discuteremo in seguito.
# define the fit measures
fit_measures_robust <- c(
"chisq", "df", "pvalue",
"cfi", "rmsea", "srmr"
)
Confrontiamo le misure di bontà di adattamento del modello che ipotizza un solo fattore comune e il modello che ipotizza la presenza di due fattori comuni.
# collect them for each model
rbind(
fitmeasures(efa_f1, fit_measures_robust),
fitmeasures(efa_f2, fit_measures_robust)
) %>%
# wrangle
data.frame() %>%
mutate(
chisq = round(chisq, digits = 0),
df = as.integer(df),
pvalue = ifelse(pvalue == 0, "< .001", pvalue)
) %>%
mutate_at(vars(cfi:srmr), ~ round(., digits = 3))
| chisq | df | pvalue | cfi | rmsea | srmr |
|---|---|---|---|---|---|
| <dbl> | <int> | <chr> | <dbl> | <dbl> | <dbl> |
| 375 | 20 | < .001 | 0.71 | 0.267 | 0.187 |
| 10 | 13 | 0.709310449320098 | 1.00 | 0.000 | 0.010 |
L’evidenza empirica supporta la superiorità del modello a due fattori rispetto a quello ad un solo fattore comune. In particolare, l’analisi fattoriale esplorativa svolta mediante la funzione efa() evidenzia la capacità del modello a due fattori di fornire una descrizione adeguata della struttura dei dati e di distinguere in modo sensato tra i due fattori ipotizzati.
Esercizio. Si utilizzino i dati dass21.txt che corrispondono alla somministrazione del test DASS-21 a 334 partecipanti. Lo schema di codifica si può trovare seguendo questo link. Si adatti ai dati un modello a tre fattori usando l’analisi fattoriale esplorativa con la funzione lavaan::efa(). Usando le saturazioni fattoriali e la matrice di inter-correlazioni fattoriali, si trovi la matrice di correlazioni riprodotta dal modello. Senza usare l’albebra matriciale, si trovi la correlazione predetta tra gli indicatori DASS-1 e DASS-2.
